
Hola Habr!
El otoño pasado, Kaggle realizó un concurso para la clasificación de imágenes de reconocimiento de dibujo rápido dibujadas a mano, en el que, entre otros, participó un equipo de R-schiks compuesto por
Artem Klevtsov ,
Philippe Pravitelev y
Andrey Ogurtsov . No describiremos la competencia en detalle, esto ya se hizo en una
publicación reciente .
Esta vez no hubo medallas con productos farmacéuticos agrícolas, pero se obtuvo mucha experiencia valiosa, por lo que me gustaría contarle a la comunidad sobre algunas de las cosas más interesantes y útiles en Kagl y en el trabajo diario. Entre los temas discutidos: vida dura sin
OpenCV , análisis de JSON (estos ejemplos
muestran la integración de código C ++ en scripts o paquetes en R usando
Rcpp ), parametrización de scripts y dockerización de la solución final. Todo el código del mensaje en una forma adecuada para el lanzamiento está disponible en el
repositorio .
Contenido:
- Carga efectiva de datos desde CSV a la base de datos MonetDB
- Preparación de lotes
- Iteradores para descargar lotes de la base de datos
- Selección de arquitectura de modelo
- Parametrización de guiones
- Guiones de acoplamiento
- Uso de múltiples GPU en Google Cloud
- En lugar de una conclusión
1. Carga efectiva de datos desde CSV a la base de datos MonetDB
Los datos en esta competencia no se proporcionan en forma de imágenes ya hechas, sino en forma de 340 archivos CSV (un archivo para cada clase) que contienen JSON con coordenadas de puntos. Conectando estos puntos con líneas, obtenemos la imagen final con un tamaño de 256x256 píxeles. Además, para cada registro, se da una etiqueta si la imagen fue reconocida correctamente por el clasificador utilizado en el momento en que se recopiló el conjunto de datos, el código de dos letras del país de residencia del autor, un identificador único, sello de tiempo y nombre de clase que coincida con el nombre del archivo. Una versión simplificada de los datos de origen pesa 7,4 GB en el archivo y aproximadamente 20 GB después del desempaquetado, los datos completos después del desempaquetado toman 240 GB. Los organizadores aseguraron que ambas versiones reproducen los mismos dibujos, es decir, la versión completa es redundante. En cualquier caso, almacenar 50 millones de imágenes en archivos gráficos o en matrices se consideró inmediatamente no rentable, y decidimos fusionar todos los archivos CSV del archivo train_simplified.zip en una base de datos con la generación posterior de imágenes del tamaño correcto sobre la marcha para cada lote .
El bien establecido MonetDB fue elegido como DBMS, a saber, la implementación de R en forma de paquete MonetDBLite . El paquete incluye una versión incrustada del servidor de base de datos y le permite levantar el servidor directamente de la sesión R y trabajar con él allí. La creación de una base de datos y la conexión a ella se realizan mediante un comando:
con <- DBI::dbConnect(drv = MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR"))
Tendremos que crear dos tablas: una para todos los datos, la otra para la información general sobre los archivos descargados (útil si algo sale mal y el proceso tendrá que reanudarse después de cargar varios archivos):
Crear tablas if (!DBI::dbExistsTable(con, "doodles")) { DBI::dbCreateTable( con = con, name = "doodles", fields = c( "countrycode" = "char(2)", "drawing" = "text", "key_id" = "bigint", "recognized" = "bool", "timestamp" = "timestamp", "word" = "text" ) ) } if (!DBI::dbExistsTable(con, "upload_log")) { DBI::dbCreateTable( con = con, name = "upload_log", fields = c( "id" = "serial", "file_name" = "text UNIQUE", "uploaded" = "bool DEFAULT false" ) ) }
La forma más rápida de cargar datos en la base de datos fue copiar directamente los archivos CSV utilizando SQL: el comando COPY OFFSET 2 INTO tablename FROM path USING DELIMITERS ',','\\n','\"' NULL AS '' BEST EFFORT , donde el nombre de la tablename es el nombre de la tabla y la path es la ruta al archivo. Más tarde , se descubrió otra forma de aumentar la velocidad: simplemente reemplace BEST EFFORT por LOCKED BEST EFFORT . Al trabajar con el archivo, resultó que la implementación de unzip incorporada en R no funciona correctamente con varios archivos del archivo, por lo que utilizamos unzip sistema (usando el getOption("unzip") ).
Función para escribir en la base de datos. #' @title #' #' @description #' CSV- ZIP- #' #' @param con ( `MonetDBEmbeddedConnection`). #' @param tablename . #' @oaram zipfile ZIP-. #' @oaram filename ZIP-. #' @param preprocess , . #' `data` ( `data.table`). #' #' @return `TRUE`. #' upload_file <- function(con, tablename, zipfile, filename, preprocess = NULL) { # checkmate::assert_class(con, "MonetDBEmbeddedConnection") checkmate::assert_string(tablename) checkmate::assert_string(filename) checkmate::assert_true(DBI::dbExistsTable(con, tablename)) checkmate::assert_file_exists(zipfile, access = "r", extension = "zip") checkmate::assert_function(preprocess, args = c("data"), null.ok = TRUE) # path <- file.path(tempdir(), filename) unzip(zipfile, files = filename, exdir = tempdir(), junkpaths = TRUE, unzip = getOption("unzip")) on.exit(unlink(file.path(path))) # if (!is.null(preprocess)) { .data <- data.table::fread(file = path) .data <- preprocess(data = .data) data.table::fwrite(x = .data, file = path, append = FALSE) rm(.data) } # CSV sql <- sprintf( "COPY OFFSET 2 INTO %s FROM '%s' USING DELIMITERS ',','\\n','\"' NULL AS '' BEST EFFORT", tablename, path ) # DBI::dbExecute(con, sql) # DBI::dbExecute(con, sprintf("INSERT INTO upload_log(file_name, uploaded) VALUES('%s', true)", filename)) return(invisible(TRUE)) }
En caso de que necesite convertir la tabla antes de escribir en la base de datos, es suficiente pasar la función que convertirá los datos en el argumento de preprocess .
Código para la carga secuencial de datos en la base de datos:
Escribir datos en la base de datos # files <- unzip(zipfile, list = TRUE)$Name # , to_skip <- DBI::dbGetQuery(con, "SELECT file_name FROM upload_log")[[1L]] files <- setdiff(files, to_skip) if (length(files) > 0L) { # tictoc::tic() # pb <- txtProgressBar(min = 0L, max = length(files), style = 3) for (i in seq_along(files)) { upload_file(con = con, tablename = "doodles", zipfile = zipfile, filename = files[i]) setTxtProgressBar(pb, i) } close(pb) # tictoc::toc() } # 526.141 sec elapsed - SSD->SSD # 558.879 sec elapsed - USB->SSD
El tiempo de carga de datos puede variar según las características de velocidad de la unidad utilizada. En nuestro caso, leer y escribir en el mismo SSD o desde una unidad flash USB (archivo fuente) en un SSD (base de datos) lleva menos de 10 minutos.
Se necesitan unos segundos más para crear una columna con una etiqueta de clase entera y una columna de índice ( ORDERED INDEX ) con números de línea, que se utilizarán para seleccionar casos al crear lotes:
Crear columnas e índices adicionales message("Generate lables") invisible(DBI::dbExecute(con, "ALTER TABLE doodles ADD label_int int")) invisible(DBI::dbExecute(con, "UPDATE doodles SET label_int = dense_rank() OVER (ORDER BY word) - 1")) message("Generate row numbers") invisible(DBI::dbExecute(con, "ALTER TABLE doodles ADD id serial")) invisible(DBI::dbExecute(con, "CREATE ORDERED INDEX doodles_id_ord_idx ON doodles(id)"))
Para resolver el problema de crear un lote "sobre la marcha", necesitábamos alcanzar la velocidad máxima de extracción de cadenas aleatorias de la tabla de doodles . Para esto utilizamos 3 trucos. El primero fue reducir la dimensión del tipo en el que se almacena la identificación de observación. En el conjunto de datos original, se requiere el tipo bigint para almacenar la ID, pero el número de observaciones permite ajustar sus identificadores iguales al número de serie en el tipo int . La búsqueda es mucho más rápida. El segundo truco fue usar el ORDERED INDEX : esta decisión se tomó empíricamente, clasificando todas las opciones disponibles. El tercero era usar consultas parametrizadas. La esencia del método es ejecutar el comando PREPARE una vez y luego usar la expresión preparada para crear un montón del mismo tipo de consultas, pero en realidad la ganancia en comparación con el SELECT simple SELECT en el área de error estadístico.
El proceso de llenado de datos no consume más de 450 MB de RAM. Es decir, el enfoque descrito le permite rotar conjuntos de datos que pesan decenas de gigabytes en casi cualquier hardware económico, incluidas algunas computadoras de una sola placa, lo cual es bastante bueno.
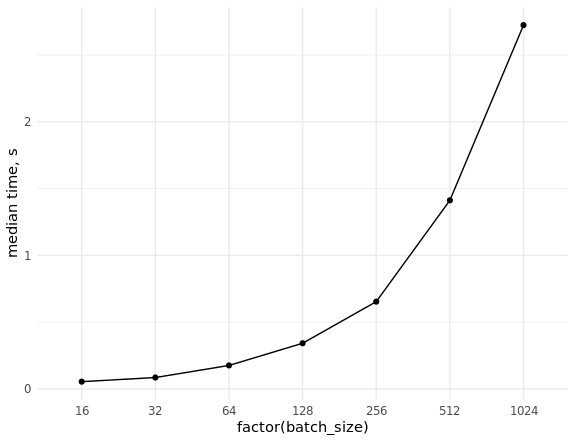
Queda por tomar medidas de la tasa de extracción de datos (aleatorios) y evaluar la escala al muestrear lotes de diferentes tamaños:
Base de datos de referencia library(ggplot2) set.seed(0) # con <- DBI::dbConnect(MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR")) # prep_sql <- function(batch_size) { sql <- sprintf("PREPARE SELECT id FROM doodles WHERE id IN (%s)", paste(rep("?", batch_size), collapse = ",")) res <- DBI::dbSendQuery(con, sql) return(res) } # fetch_data <- function(rs, batch_size) { ids <- sample(seq_len(n), batch_size) res <- DBI::dbFetch(DBI::dbBind(rs, as.list(ids))) return(res) } # res_bench <- bench::press( batch_size = 2^(4:10), { rs <- prep_sql(batch_size) bench::mark( fetch_data(rs, batch_size), min_iterations = 50L ) } ) # cols <- c("batch_size", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # batch_size min median max `itr/sec` total_time n_itr # <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 16 23.6ms 54.02ms 93.43ms 18.8 2.6s 49 # 2 32 38ms 84.83ms 151.55ms 11.4 4.29s 49 # 3 64 63.3ms 175.54ms 248.94ms 5.85 8.54s 50 # 4 128 83.2ms 341.52ms 496.24ms 3.00 16.69s 50 # 5 256 232.8ms 653.21ms 847.44ms 1.58 31.66s 50 # 6 512 784.6ms 1.41s 1.98s 0.740 1.1m 49 # 7 1024 681.7ms 2.72s 4.06s 0.377 2.16m 49 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = 1)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() DBI::dbDisconnect(con, shutdown = TRUE)
2. Preparación de lotes
Todo el proceso de preparación de lotes consta de los siguientes pasos:
- Analizando múltiples JSON que contienen vectores de línea con coordenadas de puntos.
- Dibujar líneas coloreadas por las coordenadas de los puntos en la imagen del tamaño deseado (por ejemplo, 256x256 o 128x128).
- Convierta las imágenes resultantes en un tensor.
En el marco de la competencia entre los kernel-s en Python, el problema se resolvió principalmente mediante OpenCV . Uno de los análogos más simples y obvios en R se verá así:
Implemente JSON para la conversión de tensor en R r_process_json_str <- function(json, line.width = 3, color = TRUE, scale = 1) { # JSON coords <- jsonlite::fromJSON(json, simplifyMatrix = FALSE) tmp <- tempfile() # on.exit(unlink(tmp)) png(filename = tmp, width = 256 * scale, height = 256 * scale, pointsize = 1) # plot.new() # plot.window(xlim = c(256 * scale, 0), ylim = c(256 * scale, 0)) # cols <- if (color) rainbow(length(coords)) else "#000000" for (i in seq_along(coords)) { lines(x = coords[[i]][[1]] * scale, y = coords[[i]][[2]] * scale, col = cols[i], lwd = line.width) } dev.off() # 3- res <- png::readPNG(tmp) return(res) } r_process_json_vector <- function(x, ...) { res <- lapply(x, r_process_json_str, ...) # 3- 4- res <- do.call(abind::abind, c(res, along = 0)) return(res) }
El dibujo se realiza utilizando herramientas R estándar y se guarda en un PNG temporal almacenado en RAM (en Linux, los directorios R temporales se encuentran en el /tmp montado en RAM). Luego, este archivo se lee en forma de una matriz tridimensional con números en el rango de 0 a 1. Esto es importante ya que el BMP más común se leería en una matriz sin formato con códigos de color hexadecimales.
Prueba el resultado:
zip_file <- file.path("data", "train_simplified.zip") csv_file <- "cat.csv" unzip(zip_file, files = csv_file, exdir = tempdir(), junkpaths = TRUE, unzip = getOption("unzip")) tmp_data <- data.table::fread(file.path(tempdir(), csv_file), sep = ",", select = "drawing", nrows = 10000) arr <- r_process_json_str(tmp_data[4, drawing]) dim(arr) # [1] 256 256 3 plot(magick::image_read(arr))

El lote en sí se formará de la siguiente manera:
res <- r_process_json_vector(tmp_data[1:4, drawing], scale = 0.5) str(res) # num [1:4, 1:128, 1:128, 1:3] 1 1 1 1 1 1 1 1 1 1 ... # - attr(*, "dimnames")=List of 4 # ..$ : NULL # ..$ : NULL # ..$ : NULL # ..$ : NULL
Esta implementación nos pareció no óptima, ya que la formación de grandes lotes lleva mucho tiempo indecente, y decidimos usar la experiencia de nuestros colegas que usan la poderosa biblioteca OpenCV . En ese momento, no había un paquete listo para R (no hay ninguno incluso ahora), por lo que se escribió una implementación mínima de la funcionalidad requerida en C ++ con integración en el código R usando Rcpp .
Para resolver el problema, se utilizaron los siguientes paquetes y bibliotecas:
- OpenCV para imágenes y dibujos lineales. Utilizamos bibliotecas del sistema preinstaladas y archivos de encabezado, así como enlaces dinámicos.
- xtensor para trabajar con matrices multidimensionales y tensores. Utilizamos los archivos de encabezado incluidos en el paquete R del mismo nombre. La biblioteca le permite trabajar con matrices multidimensionales, tanto en orden principal de fila como en orden principal de columna.
- ndjson para analizar JSON. Esta biblioteca se usa en xtensor automáticamente si existe en el proyecto.
- RcppThread para organizar el procesamiento de subprocesos múltiples de un vector de JSON. Usó los archivos de encabezado proporcionados por este paquete. El paquete difiere del RcppParallel más popular, entre otras cosas, por su mecanismo de interrupción incorporado.
Vale la pena señalar que xtensor resultó ser solo un hallazgo: además de tener una amplia funcionalidad y un alto rendimiento, sus desarrolladores resultaron ser muy receptivos y rápidos y en detalle respondieron preguntas que surgieron. Con su ayuda, fue posible implementar la transformación de las matrices OpenCV en tensores xtensores, así como un método para combinar tensores de imágenes tridimensionales en un tensor de 4 dimensiones de la dimensión correcta (en realidad el lote).
Materiales de estudio para Rcpp, xtensor y RcppThread Para compilar archivos utilizando archivos del sistema y enlaces dinámicos con bibliotecas instaladas en el sistema, utilizamos el mecanismo de complemento implementado en el paquete Rcpp . Para encontrar automáticamente rutas e indicadores, utilizamos la popular utilidad de Linux pkg-config .
Implementación de un complemento Rcpp para usar la biblioteca OpenCV Rcpp::registerPlugin("opencv", function() { # pkg_config_name <- c("opencv", "opencv4") # pkg-config pkg_config_bin <- Sys.which("pkg-config") # checkmate::assert_file_exists(pkg_config_bin, access = "x") # OpenCV pkg-config check <- sapply(pkg_config_name, function(pkg) system(paste(pkg_config_bin, pkg))) if (all(check != 0)) { stop("OpenCV config for the pkg-config not found", call. = FALSE) } pkg_config_name <- pkg_config_name[check == 0] list(env = list( PKG_CXXFLAGS = system(paste(pkg_config_bin, "--cflags", pkg_config_name), intern = TRUE), PKG_LIBS = system(paste(pkg_config_bin, "--libs", pkg_config_name), intern = TRUE) )) })
Como resultado del complemento, durante la compilación, se sustituirán los siguientes valores:
Rcpp:::.plugins$opencv()$env # $PKG_CXXFLAGS # [1] "-I/usr/include/opencv" # # $PKG_LIBS # [1] "-lopencv_shape -lopencv_stitching -lopencv_superres -lopencv_videostab -lopencv_aruco -lopencv_bgsegm -lopencv_bioinspired -lopencv_ccalib -lopencv_datasets -lopencv_dpm -lopencv_face -lopencv_freetype -lopencv_fuzzy -lopencv_hdf -lopencv_line_descriptor -lopencv_optflow -lopencv_video -lopencv_plot -lopencv_reg -lopencv_saliency -lopencv_stereo -lopencv_structured_light -lopencv_phase_unwrapping -lopencv_rgbd -lopencv_viz -lopencv_surface_matching -lopencv_text -lopencv_ximgproc -lopencv_calib3d -lopencv_features2d -lopencv_flann -lopencv_xobjdetect -lopencv_objdetect -lopencv_ml -lopencv_xphoto -lopencv_highgui -lopencv_videoio -lopencv_imgcodecs -lopencv_photo -lopencv_imgproc -lopencv_core"
El código para implementar el análisis JSON y crear un lote para transferir al modelo se proporciona en el spoiler. Primero, agregue el directorio del proyecto local para buscar archivos de encabezado (necesarios para ndjson):
Sys.setenv("PKG_CXXFLAGS" = paste0("-I", normalizePath(file.path("src"))))
Implementación de conversión JSON a tensor en C ++ // [[Rcpp::plugins(cpp14)]] // [[Rcpp::plugins(opencv)]] // [[Rcpp::depends(xtensor)]] // [[Rcpp::depends(RcppThread)]] #include <xtensor/xjson.hpp> #include <xtensor/xadapt.hpp> #include <xtensor/xview.hpp> #include <xtensor-r/rtensor.hpp> #include <opencv2/core/core.hpp> #include <opencv2/highgui/highgui.hpp> #include <opencv2/imgproc/imgproc.hpp> #include <Rcpp.h> #include <RcppThread.h> // using RcppThread::parallelFor; using json = nlohmann::json; using points = xt::xtensor<double,2>; // JSON using strokes = std::vector<points>; // JSON using xtensor3d = xt::xtensor<double, 3>; // using xtensor4d = xt::xtensor<double, 4>; // using rtensor3d = xt::rtensor<double, 3>; // R using rtensor4d = xt::rtensor<double, 4>; // R // // const static int SIZE = 256; // // . https://en.wikipedia.org/wiki/Pixel_connectivity#2-dimensional const static int LINE_TYPE = cv::LINE_4; // const static int LINE_WIDTH = 3; // // https://docs.opencv.org/3.1.0/da/d54/group__imgproc__transform.html#ga5bb5a1fea74ea38e1a5445ca803ff121 const static int RESIZE_TYPE = cv::INTER_LINEAR; // OpenCV- template <typename T, int NCH, typename XT=xt::xtensor<T,3,xt::layout_type::column_major>> XT to_xt(const cv::Mat_<cv::Vec<T, NCH>>& src) { // std::vector<int> shape = {src.rows, src.cols, NCH}; // size_t size = src.total() * NCH; // cv::Mat xt::xtensor XT res = xt::adapt((T*) src.data, size, xt::no_ownership(), shape); return res; } // JSON strokes parse_json(const std::string& x) { auto j = json::parse(x); // if (!j.is_array()) { throw std::runtime_error("'x' must be JSON array."); } strokes res; res.reserve(j.size()); for (const auto& a: j) { // 2- if (!a.is_array() || a.size() != 2) { throw std::runtime_error("'x' must include only 2d arrays."); } // auto p = a.get<points>(); res.push_back(p); } return res; } // // HSV cv::Mat ocv_draw_lines(const strokes& x, bool color = true) { // auto stype = color ? CV_8UC3 : CV_8UC1; // auto dtype = color ? CV_32FC3 : CV_32FC1; auto bg = color ? cv::Scalar(0, 0, 255) : cv::Scalar(255); auto col = color ? cv::Scalar(0, 255, 220) : cv::Scalar(0); cv::Mat img = cv::Mat(SIZE, SIZE, stype, bg); // size_t n = x.size(); for (const auto& s: x) { // size_t n_points = s.shape()[1]; for (size_t i = 0; i < n_points - 1; ++i) { // cv::Point from(s(0, i), s(1, i)); // cv::Point to(s(0, i + 1), s(1, i + 1)); // cv::line(img, from, to, col, LINE_WIDTH, LINE_TYPE); } if (color) { // col[0] += 180 / n; } } if (color) { // RGB cv::cvtColor(img, img, cv::COLOR_HSV2RGB); } // float32 [0, 1] img.convertTo(img, dtype, 1 / 255.0); return img; } // JSON xtensor3d process(const std::string& x, double scale = 1.0, bool color = true) { auto p = parse_json(x); auto img = ocv_draw_lines(p, color); if (scale != 1) { cv::Mat out; cv::resize(img, out, cv::Size(), scale, scale, RESIZE_TYPE); cv::swap(img, out); out.release(); } xtensor3d arr = color ? to_xt<double,3>(img) : to_xt<double,1>(img); return arr; } // [[Rcpp::export]] rtensor3d cpp_process_json_str(const std::string& x, double scale = 1.0, bool color = true) { xtensor3d res = process(x, scale, color); return res; } // [[Rcpp::export]] rtensor4d cpp_process_json_vector(const std::vector<std::string>& x, double scale = 1.0, bool color = false) { size_t n = x.size(); size_t dim = floor(SIZE * scale); size_t channels = color ? 3 : 1; xtensor4d res({n, dim, dim, channels}); parallelFor(0, n, [&x, &res, scale, color](int i) { xtensor3d tmp = process(x[i], scale, color); auto view = xt::view(res, i, xt::all(), xt::all(), xt::all()); view = tmp; }); return res; }
Este código debe colocarse en el src/cv_xt.cpp y compilarse con el comando Rcpp::sourceCpp(file = "src/cv_xt.cpp", env = .GlobalEnv) ; También necesitará nlohmann/json.hpp del repositorio para trabajar . El código se divide en varias funciones:
to_xt : una función de plantilla para convertir la matriz de imagen ( cv::Mat ) en el tensor xt::xtensor ;parse_json : la función analiza una cadena JSON, extrae las coordenadas de los puntos y los empaqueta en un vector;ocv_draw_lines - ocv_draw_lines líneas multicolores del vector de puntos recibido;process : combina las funciones anteriores y también agrega la capacidad de escalar la imagen resultante;cpp_process_json_str : un contenedor sobre la función de process , que exporta el resultado a un objeto R (matriz multidimensional);cpp_process_json_vector : un contenedor sobre la función cpp_process_json_str , que le permite procesar un vector de cadena en modo de subprocesos múltiples.
Para dibujar líneas multicolores, se utilizó el modelo de color HSV, seguido de la conversión a RGB. Prueba el resultado:
arr <- cpp_process_json_str(tmp_data[4, drawing]) dim(arr) # [1] 256 256 3 plot(magick::image_read(arr))

Comparación de la velocidad de implementaciones en R y C ++ res_bench <- bench::mark( r_process_json_str(tmp_data[4, drawing], scale = 0.5), cpp_process_json_str(tmp_data[4, drawing], scale = 0.5), check = FALSE, min_iterations = 100 ) # cols <- c("expression", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # expression min median max `itr/sec` total_time n_itr # <chr> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 r_process_json_str 3.49ms 3.55ms 4.47ms 273. 490ms 134 # 2 cpp_process_json_str 1.94ms 2.02ms 5.32ms 489. 497ms 243 library(ggplot2) # res_bench <- bench::press( batch_size = 2^(4:10), { .data <- tmp_data[sample(seq_len(.N), batch_size), drawing] bench::mark( r_process_json_vector(.data, scale = 0.5), cpp_process_json_vector(.data, scale = 0.5), min_iterations = 50, check = FALSE ) } ) res_bench[, cols] # expression batch_size min median max `itr/sec` total_time n_itr # <chr> <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 r 16 50.61ms 53.34ms 54.82ms 19.1 471.13ms 9 # 2 cpp 16 4.46ms 5.39ms 7.78ms 192. 474.09ms 91 # 3 r 32 105.7ms 109.74ms 212.26ms 7.69 6.5s 50 # 4 cpp 32 7.76ms 10.97ms 15.23ms 95.6 522.78ms 50 # 5 r 64 211.41ms 226.18ms 332.65ms 3.85 12.99s 50 # 6 cpp 64 25.09ms 27.34ms 32.04ms 36.0 1.39s 50 # 7 r 128 534.5ms 627.92ms 659.08ms 1.61 31.03s 50 # 8 cpp 128 56.37ms 58.46ms 66.03ms 16.9 2.95s 50 # 9 r 256 1.15s 1.18s 1.29s 0.851 58.78s 50 # 10 cpp 256 114.97ms 117.39ms 130.09ms 8.45 5.92s 50 # 11 r 512 2.09s 2.15s 2.32s 0.463 1.8m 50 # 12 cpp 512 230.81ms 235.6ms 261.99ms 4.18 11.97s 50 # 13 r 1024 4s 4.22s 4.4s 0.238 3.5m 50 # 14 cpp 1024 410.48ms 431.43ms 462.44ms 2.33 21.45s 50 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = expression, color = expression)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() + scale_color_discrete(name = "", labels = c("cpp", "r")) + theme(legend.position = "bottom")
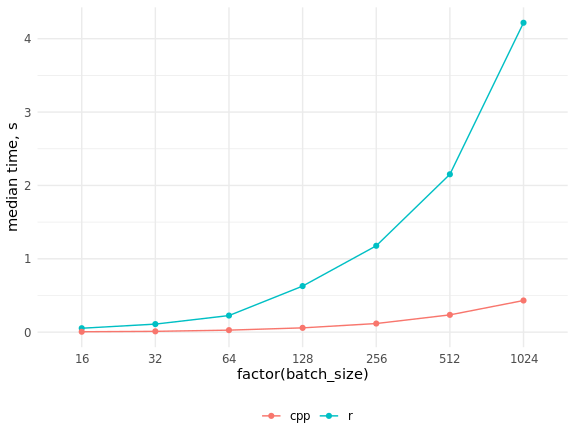
Como puede ver, el aumento de velocidad resultó ser muy significativo, y no es posible ponerse al día con el código C ++ paralelizando el código R.
3. Iteradores para descargar lotes de la base de datos
R tiene una reputación bien merecida como lenguaje para procesar datos ubicados en RAM, mientras que el procesamiento iterativo de datos es más típico de Python, lo que facilita y facilita la implementación de cálculos fuera del núcleo (cálculos utilizando memoria externa). Clásico y relevante para nosotros en el contexto del problema descrito, un ejemplo de tales cálculos son las redes neuronales profundas, entrenadas por el método de descenso de gradiente con la aproximación del gradiente en cada paso en una pequeña porción de observaciones, o un mini lote.
Los marcos de aprendizaje profundo escritos en Python tienen clases especiales que implementan iteradores basados en datos: tablas, imágenes en carpetas, formatos binarios, etc. Puede usar opciones ya hechas o escribir las suyas para tareas específicas. En R, podemos aprovechar al máximo la biblioteca Keras Python con sus diversos backends utilizando el paquete del mismo nombre, que a su vez funciona en la parte superior del paquete reticulado . Este último merece un artículo grande separado; no solo le permite ejecutar código Python desde R, sino que también proporciona la transferencia de objetos entre sesiones R y Python, realizando automáticamente todas las conversiones de tipo necesarias.
Nos deshicimos de la necesidad de almacenar todos los datos en la RAM debido al uso de MonetDBLite, todo el trabajo de la "red neuronal" se realizará mediante el código original de Python, solo tenemos que escribir un iterador basado en los datos, ya que no hay listo para tal situación en R o Python. : ( R ). R numpy-, keras .
:
train_generator <- function(db_connection = con, samples_index, num_classes = 340, batch_size = 32, scale = 1, color = FALSE, imagenet_preproc = FALSE) { # checkmate::assert_class(con, "DBIConnection") checkmate::assert_integerish(samples_index) checkmate::assert_count(num_classes) checkmate::assert_count(batch_size) checkmate::assert_number(scale, lower = 0.001, upper = 5) checkmate::assert_flag(color) checkmate::assert_flag(imagenet_preproc) # , dt <- data.table::data.table(id = sample(samples_index)) # dt[, batch := (.I - 1L) %/% batch_size + 1L] # dt <- dt[, if (.N == batch_size) .SD, keyby = batch] # i <- 1 # max_i <- dt[, max(batch)] # sql <- sprintf( "PREPARE SELECT drawing, label_int FROM doodles WHERE id IN (%s)", paste(rep("?", batch_size), collapse = ",") ) res <- DBI::dbSendQuery(con, sql) # keras::to_categorical to_categorical <- function(x, num) { n <- length(x) m <- numeric(n * num) m[x * n + seq_len(n)] <- 1 dim(m) <- c(n, num) return(m) } # function() { # if (i > max_i) { dt[, id := sample(id)] data.table::setkey(dt, batch) # i <<- 1 max_i <<- dt[, max(batch)] } # ID batch_ind <- dt[batch == i, id] # batch <- DBI::dbFetch(DBI::dbBind(res, as.list(batch_ind)), n = -1) # i <<- i + 1 # JSON batch_x <- cpp_process_json_vector(batch$drawing, scale = scale, color = color) if (imagenet_preproc) { # c [0, 1] [-1, 1] batch_x <- (batch_x - 0.5) * 2 } batch_y <- to_categorical(batch$label_int, num_classes) result <- list(batch_x, batch_y) return(result) } }
, , , , ( scale = 1 256256 , scale = 0.5 — 128128 ), ( color = FALSE , color = TRUE ) , imagenet-. , [0, 1] [-1, 1], keras .
, data.table samples_index , , SQL- . keras::to_categorical() . , , steps_per_epoch keras::fit_generator() , if (i > max_i) .
, , JSON- ( cpp_process_json_vector() , C++) , . one-hot , , . data.table — "" data.table - R.
Core i5 :
library(Rcpp) library(keras) library(ggplot2) source("utils/rcpp.R") source("utils/keras_iterator.R") con <- DBI::dbConnect(drv = MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR")) ind <- seq_len(DBI::dbGetQuery(con, "SELECT count(*) FROM doodles")[[1L]]) num_classes <- DBI::dbGetQuery(con, "SELECT max(label_int) + 1 FROM doodles")[[1L]] # train_ind <- sample(ind, floor(length(ind) * 0.995)) # val_ind <- ind[-train_ind] rm(ind) # scale <- 0.5 # res_bench <- bench::press( batch_size = 2^(4:10), { it1 <- train_generator( db_connection = con, samples_index = train_ind, num_classes = num_classes, batch_size = batch_size, scale = scale ) bench::mark( it1(), min_iterations = 50L ) } ) # cols <- c("batch_size", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # batch_size min median max `itr/sec` total_time n_itr # <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 16 25ms 64.36ms 92.2ms 15.9 3.09s 49 # 2 32 48.4ms 118.13ms 197.24ms 8.17 5.88s 48 # 3 64 69.3ms 117.93ms 181.14ms 8.57 5.83s 50 # 4 128 157.2ms 240.74ms 503.87ms 3.85 12.71s 49 # 5 256 359.3ms 613.52ms 988.73ms 1.54 30.5s 47 # 6 512 884.7ms 1.53s 2.07s 0.674 1.11m 45 # 7 1024 2.7s 3.83s 5.47s 0.261 2.81m 44 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = 1)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() DBI::dbDisconnect(con, shutdown = TRUE)

, ( 32 ). /dev/shm , . , /etc/fstab , tmpfs /dev/shm tmpfs defaults,size=25g 0 0 . , df -h .
, :
test_generator <- function(dt, batch_size = 32, scale = 1, color = FALSE, imagenet_preproc = FALSE) { # checkmate::assert_data_table(dt) checkmate::assert_count(batch_size) checkmate::assert_number(scale, lower = 0.001, upper = 5) checkmate::assert_flag(color) checkmate::assert_flag(imagenet_preproc) # dt[, batch := (.I - 1L) %/% batch_size + 1L] data.table::setkey(dt, batch) i <- 1 max_i <- dt[, max(batch)] # function() { batch_x <- cpp_process_json_vector(dt[batch == i, drawing], scale = scale, color = color) if (imagenet_preproc) { # c [0, 1] [-1, 1] batch_x <- (batch_x - 0.5) * 2 } result <- list(batch_x) i <<- i + 1 return(result) } }
4.
mobilenet v1 , . keras , , R. : (batch, height, width, 3) , . Python , , ( , keras- ):
mobilenet v1 library(keras) top_3_categorical_accuracy <- custom_metric( name = "top_3_categorical_accuracy", metric_fn = function(y_true, y_pred) { metric_top_k_categorical_accuracy(y_true, y_pred, k = 3) } ) layer_sep_conv_bn <- function(object, filters, alpha = 1, depth_multiplier = 1, strides = c(2, 2)) { # NB! depth_multiplier != resolution multiplier # https://github.com/keras-team/keras/issues/10349 layer_depthwise_conv_2d( object = object, kernel_size = c(3, 3), strides = strides, padding = "same", depth_multiplier = depth_multiplier ) %>% layer_batch_normalization() %>% layer_activation_relu() %>% layer_conv_2d( filters = filters * alpha, kernel_size = c(1, 1), strides = c(1, 1) ) %>% layer_batch_normalization() %>% layer_activation_relu() } get_mobilenet_v1 <- function(input_shape = c(224, 224, 1), num_classes = 340, alpha = 1, depth_multiplier = 1, optimizer = optimizer_adam(lr = 0.002), loss = "categorical_crossentropy", metrics = c("categorical_crossentropy", top_3_categorical_accuracy)) { inputs <- layer_input(shape = input_shape) outputs <- inputs %>% layer_conv_2d(filters = 32, kernel_size = c(3, 3), strides = c(2, 2), padding = "same") %>% layer_batch_normalization() %>% layer_activation_relu() %>% layer_sep_conv_bn(filters = 64, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 128, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 128, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 256, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 256, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 1024, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 1024, strides = c(1, 1)) %>% layer_global_average_pooling_2d() %>% layer_dense(units = num_classes) %>% layer_activation_softmax() model <- keras_model( inputs = inputs, outputs = outputs ) model %>% compile( optimizer = optimizer, loss = loss, metrics = metrics ) return(model) }
. , , , . , imagenet-. , . get_config() ( base_model_conf$layers — R- ), from_config() :
base_model_conf <- get_config(base_model) base_model_conf$layers[[1]]$config$batch_input_shape[[4]] <- 1L base_model <- from_config(base_model_conf)
keras imagenet- :
get_model <- function(name = "mobilenet_v2", input_shape = NULL, weights = "imagenet", pooling = "avg", num_classes = NULL, optimizer = keras::optimizer_adam(lr = 0.002), loss = "categorical_crossentropy", metrics = NULL, color = TRUE, compile = FALSE) { # checkmate::assert_string(name) checkmate::assert_integerish(input_shape, lower = 1, upper = 256, len = 3) checkmate::assert_count(num_classes) checkmate::assert_flag(color) checkmate::assert_flag(compile) # keras model_fun <- get0(paste0("application_", name), envir = asNamespace("keras")) # if (is.null(model_fun)) { stop("Model ", shQuote(name), " not found.", call. = FALSE) } base_model <- model_fun( input_shape = input_shape, include_top = FALSE, weights = weights, pooling = pooling ) # , if (!color) { base_model_conf <- keras::get_config(base_model) base_model_conf$layers[[1]]$config$batch_input_shape[[4]] <- 1L base_model <- keras::from_config(base_model_conf) } predictions <- keras::get_layer(base_model, "global_average_pooling2d_1")$output predictions <- keras::layer_dense(predictions, units = num_classes, activation = "softmax") model <- keras::keras_model( inputs = base_model$input, outputs = predictions ) if (compile) { keras::compile( object = model, optimizer = optimizer, loss = loss, metrics = metrics ) } return(model) }
. : get_weights() R- , ( - ), set_weights() . , , .
mobilenet 1 2, resnet34. , SE-ResNeXt. , , ( ).
5.
, docopt :
doc <- ' Usage: train_nn.R --help train_nn.R --list-models train_nn.R [options] Options: -h --help Show this message. -l --list-models List available models. -m --model=<model> Neural network model name [default: mobilenet_v2]. -b --batch-size=<size> Batch size [default: 32]. -s --scale-factor=<ratio> Scale factor [default: 0.5]. -c --color Use color lines [default: FALSE]. -d --db-dir=<path> Path to database directory [default: Sys.getenv("db_dir")]. -r --validate-ratio=<ratio> Validate sample ratio [default: 0.995]. -n --n-gpu=<number> Number of GPUs [default: 1]. ' args <- docopt::docopt(doc)
docopt http://docopt.org/ R. Rscript bin/train_nn.R -m resnet50 -c -d /home/andrey/doodle_db ./bin/train_nn.R -m resnet50 -c -d /home/andrey/doodle_db , train_nn.R ( resnet50 128128 , /home/andrey/doodle_db ). , . , mobilenet_v2 keras R - R- — , .
RStudio ( tfruns ). , RStudio.
6.
. R- .
« », . , NVIDIA, CUDA+cuDNN — , tensorflow/tensorflow:1.12.0-gpu , R-.
- :
Dockerfile FROM tensorflow/tensorflow:1.12.0-gpu MAINTAINER Artem Klevtsov <aaklevtsov@gmail.com> SHELL ["/bin/bash", "-c"] ARG LOCALE="en_US.UTF-8" ARG APT_PKG="libopencv-dev r-base r-base-dev littler" ARG R_BIN_PKG="futile.logger checkmate data.table rcpp rapidjsonr dbi keras jsonlite curl digest remotes" ARG R_SRC_PKG="xtensor RcppThread docopt MonetDBLite" ARG PY_PIP_PKG="keras" ARG DIRS="/db /app /app/data /app/models /app/logs" RUN source /etc/os-release && \ echo "deb https://cloud.r-project.org/bin/linux/ubuntu ${UBUNTU_CODENAME}-cran35/" > /etc/apt/sources.list.d/cran35.list && \ apt-key adv --keyserver keyserver.ubuntu.com --recv-keys E084DAB9 && \ add-apt-repository -y ppa:marutter/c2d4u3.5 && \ add-apt-repository -y ppa:timsc/opencv-3.4 && \ apt-get update && \ apt-get install -y locales && \ locale-gen ${LOCALE} && \ apt-get install -y --no-install-recommends ${APT_PKG} && \ ln -s /usr/lib/R/site-library/littler/examples/install.r /usr/local/bin/install.r && \ ln -s /usr/lib/R/site-library/littler/examples/install2.r /usr/local/bin/install2.r && \ ln -s /usr/lib/R/site-library/littler/examples/installGithub.r /usr/local/bin/installGithub.r && \ echo 'options(Ncpus = parallel::detectCores())' >> /etc/R/Rprofile.site && \ echo 'options(repos = c(CRAN = "https://cloud.r-project.org"))' >> /etc/R/Rprofile.site && \ apt-get install -y $(printf "r-cran-%s " ${R_BIN_PKG}) && \ install.r ${R_SRC_PKG} && \ pip install ${PY_PIP_PKG} && \ mkdir -p ${DIRS} && \ chmod 777 ${DIRS} && \ rm -rf /tmp/downloaded_packages/ /tmp/*.rds && \ rm -rf /var/lib/apt/lists/* COPY utils /app/utils COPY src /app/src COPY tests /app/tests COPY bin/*.R /app/ ENV DBDIR="/db" ENV CUDA_HOME="/usr/local/cuda" ENV PATH="/app:${PATH}" WORKDIR /app VOLUME /db VOLUME /app CMD bash
; . /bin/bash /etc/os-release . .
-, . , , , :
#!/bin/sh DBDIR=${PWD}/db LOGSDIR=${PWD}/logs MODELDIR=${PWD}/models DATADIR=${PWD}/data ARGS="--runtime=nvidia --rm -v ${DBDIR}:/db -v ${LOGSDIR}:/app/logs -v ${MODELDIR}:/app/models -v ${DATADIR}:/app/data" if [ -z "$1" ]; then CMD="Rscript /app/train_nn.R" elif [ "$1" = "bash" ]; then ARGS="${ARGS} -ti" else CMD="Rscript /app/train_nn.R $@" fi docker run ${ARGS} doodles-tf ${CMD}
- , train_nn.R ; — "bash", . : CMD="Rscript /app/train_nn.R $@" .
, , , .
7. GPU Google Cloud
(. , @Leigh.plt ODS-). , 1 GPU GPU . GoogleCloud ( ) - , $300. 4V100 SSD , . , . K80. — SSD c, dev/shm .
, GPU. CPU , :
with(tensorflow::tf$device("/cpu:0"), { model_cpu <- get_model( name = model_name, input_shape = input_shape, weights = weights, metrics =(top_3_categorical_accuracy, compile = FALSE ) })
( ) GPU, :
model <- keras::multi_gpu_model(model_cpu, gpus = n_gpu) keras::compile( object = model, optimizer = keras::optimizer_adam(lr = 0.0004), loss = "categorical_crossentropy", metrics = c(top_3_categorical_accuracy) )
, , , GPU .
tensorboard , :
# log_file_tmpl <- file.path("logs", sprintf( "%s_%d_%dch_%s.csv", model_name, dim_size, channels, format(Sys.time(), "%Y%m%d%H%M%OS") )) # model_file_tmpl <- file.path("models", sprintf( "%s_%d_%dch_{epoch:02d}_{val_loss:.2f}.h5", model_name, dim_size, channels )) callbacks_list <- list( keras::callback_csv_logger( filename = log_file_tmpl ), keras::callback_early_stopping( monitor = "val_loss", min_delta = 1e-4, patience = 8, verbose = 1, mode = "min" ), keras::callback_reduce_lr_on_plateau( monitor = "val_loss", factor = 0.5, # lr 2 patience = 4, verbose = 1, min_delta = 1e-4, mode = "min" ), keras::callback_model_checkpoint( filepath = model_file_tmpl, monitor = "val_loss", save_best_only = FALSE, save_weights_only = FALSE, mode = "min" ) )
8.
, , :
- keras (
lr_finder fast.ai ); , R , , ; - , GPU;
- , imagenet-;
- one cycle policy discriminative learning rates (osine annealing , skeydan ).
:
- ( ) . data.table in-place , , . .
- R C++ Rcpp . RcppThread RcppParallel , , R .
- Rcpp C++, . xtensor CRAN, , R C++. — ++ RStudio.
- docopt . , .. . RStudio , IDE .
- , . .
- Google Cloud — , .
- , R C++, bench — .
, .