 Protocolo experimental e implementación de un algoritmo de clasificación en una computadora de ADN programable
Protocolo experimental e implementación de un algoritmo de clasificación en una computadora de ADN programableLos científicos han estado experimentando durante mucho tiempo con el almacenamiento de información en el ADN y el procesamiento de esta información. Por ejemplo, científicos de la Universidad de Washington y Microsoft construyeron recientemente el
"primer Winchester de ADN del mundo" (
foto ). Este diseño puede por primera vez proporcionar el registro y la lectura de información en el almacenamiento de ADN sin intervención humana. Un logro muy significativo, dado que el ADN puede registrar información con una densidad de
2.2 petabytes por gramo . El ADN es un contenedor compacto con una densidad de grabación miles de veces mayor que los portadores existentes.
Sin embargo, todos los sistemas de ADN existentes tienen un problema: todos estos son desarrollos patentados únicos que carecen por completo de flexibilidad. Si lo comparamos con la tecnología de silicio, entonces cada grupo de investigadores desarrolla una nueva arquitectura de computadora, para la cual necesita escribir un nuevo software. Pero las cosas pueden cambiar gracias a la
primera computadora de ADN programable desarrollada en UC Davis, el Instituto de Tecnología de California y la Universidad de Maynooth.
La primera computadora programable de ADN se describe en un
artículo científico publicado el 20 de marzo de 2019 en la revista Nature. Los autores demostraron que con la ayuda de un simple desencadenante, el mismo conjunto básico de moléculas de ADN puede implementar muchos algoritmos diferentes. Aunque el estudio es un experimento puramente de laboratorio, los algoritmos moleculares programables en el futuro pueden usarse, por ejemplo, para programar robots de ADN que ya
entregan con éxito
medicamentos a las células cancerosas .
"Este es uno de los trabajos emblemáticos en esta área",
dice Torsten-Lars Schmidt, profesor asistente de biofísica experimental en la Universidad de Kent, que no participó en la investigación. "Solían demostrar el autoensamblaje algorítmico, pero no a ese grado de complejidad".
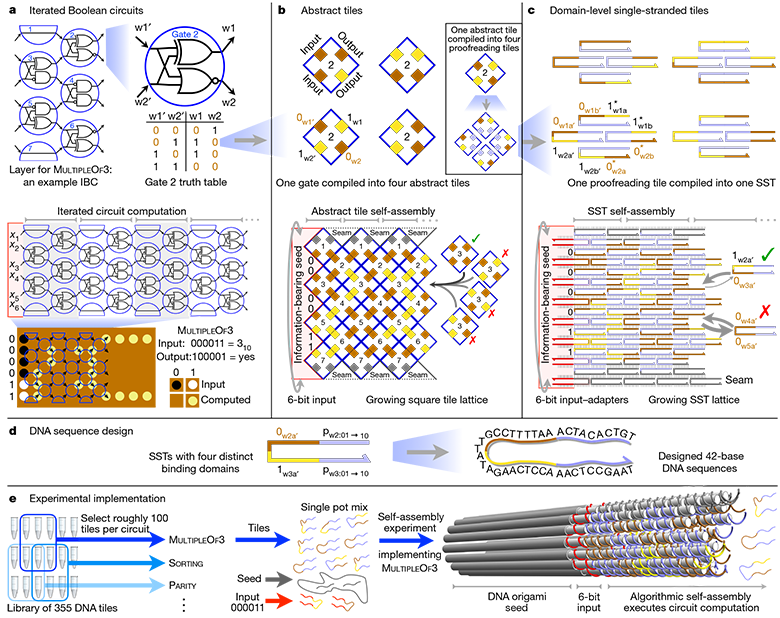
En las computadoras electrónicas, los bits son unidades binarias de información. Representan el estado físico discreto del equipo básico, por ejemplo, la presencia o ausencia de una corriente eléctrica. Estos bits, o más bien señales eléctricas, pasan a través de circuitos que consisten en elementos lógicos que realizan una operación en uno o más bits de entrada y producen un bit como salida.
Combinando estos simples bloques de construcción una y otra vez, las computadoras pueden ejecutar programas increíblemente complejos. La idea de la computación del ADN es reemplazar las señales eléctricas con enlaces químicos y el silicio con ácidos nucleicos para crear un software biomolecular.
 Jerarquía abstracta de la arquitectura e implementación práctica del circuito lógico IBC completo de 6 bits (circuito booleano iterado)
Jerarquía abstracta de la arquitectura e implementación práctica del circuito lógico IBC completo de 6 bits (circuito booleano iterado)Según Eric Winfrey, científico del Instituto de Tecnología de California y coautor del artículo, los algoritmos moleculares utilizan las posibilidades naturales de procesar información en el ADN, pero en lugar de permitir que la naturaleza tome las riendas en sus propias manos, los cálculos en el ADN se realizan de acuerdo con un programa escrito por el hombre.
En los últimos 20 años, se han llevado a cabo varios experimentos exitosos con algoritmos moleculares, por ejemplo, para jugar al tic-tac-toe o ensamblar moléculas de varias formas. En cada caso, se requirió un desarrollo cuidadoso de la secuencia de ADN para ejecutar un algoritmo específico que generaría la estructura del ADN. En este caso, la diferencia es que los investigadores desarrollaron un sistema en el que se pueden ordenar los mismos fragmentos básicos de ADN para crear
algoritmos completamente diferentes y, por lo tanto, para obtener resultados completamente diferentes.
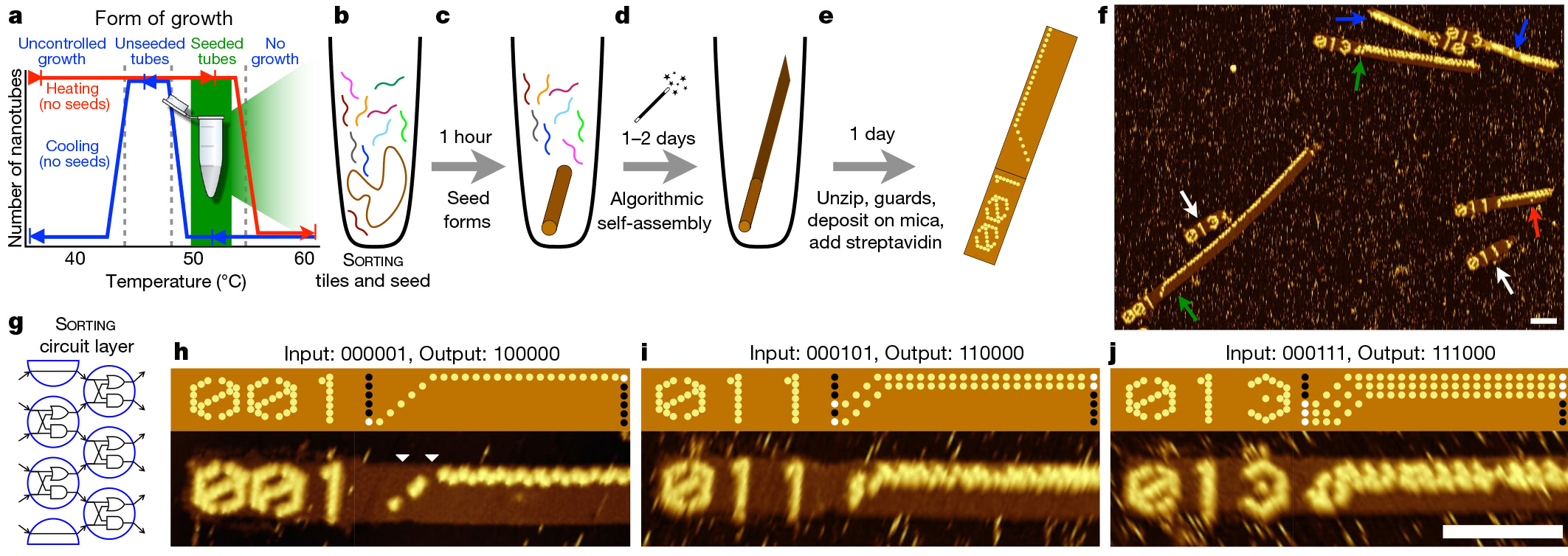
El proceso comienza con la técnica de origami de ADN, es decir, doblar una larga cadena de ADN en la forma deseada. Este segmento plegado funciona como una semilla, que ejecuta una línea de ensamblaje algorítmico. La semilla permanece prácticamente sin cambios, independientemente del algoritmo. Para cada experimento, solo se realizan pequeños cambios en varias secuencias.
 Reprogramación del circuito lógico.
Reprogramación del circuito lógico.Después de crear la "semilla", se agrega a la solución con cientos de otras cadenas de ADN, conocidas como mosaicos de ADN. Los científicos han desarrollado 355 de estos azulejos. Cada uno tiene una disposición única de bases nitrogenadas. En consecuencia, para cada algoritmo, los investigadores simplemente eligen un conjunto diferente de mosaicos iniciales. Dado que estos fragmentos de ADN están conectados durante el proceso de ensamblaje, forman un circuito que implementa el algoritmo molecular seleccionado en los bits de entrada proporcionados por la "semilla".
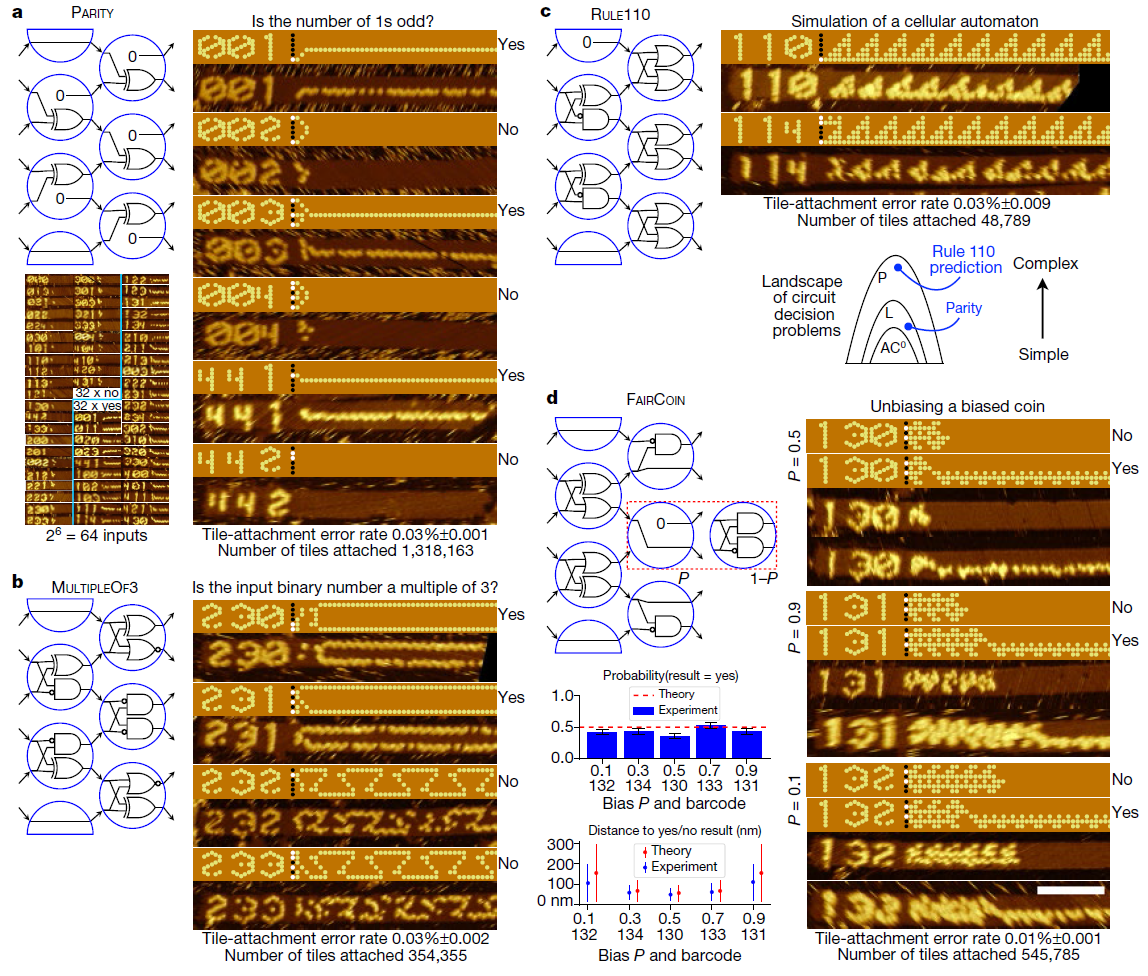
Usando este sistema, los investigadores desarrollaron y probaron 21 algoritmos para realizar tareas como reconocer una división entre tres,
elegir un líder , generar patrones y contar de 0 a 63. Todos estos algoritmos se implementan usando diferentes combinaciones de las mismas 355 fichas de ADN.
Por supuesto, no es fácil escribir código colocando fragmentos de ADN en un tubo de ensayo, pero si el proceso está automatizado, los futuros programadores moleculares ni siquiera tienen que pensar en la biomecánica, ya que los programadores de hoy no necesitan comprender la física de los transistores para escribir buenos programas.