Este artículo discute un caso para acelerar una aplicación de navegador reemplazando JavaScript con WebAssembly.
WebAssembly: ¿qué es?
En resumen, este es un formato de instrucciones binarias para una máquina virtual apilada. Wasm (nombre corto) a menudo se llama un lenguaje de programación, pero no lo es. El formato de instrucciones se ejecuta en el navegador junto con JavaScript.
Es importante que WebAssembly se pueda obtener compilando fuentes en lenguajes como C / C ++, Rust, Go. Utiliza la escritura estática y el llamado modelo de memoria plana. El código, como se mencionó anteriormente, se almacena en un formato binario compacto, por lo que se ejecuta casi tan rápido como si la aplicación se iniciara utilizando la línea de comandos. Estas características han llevado a la creciente popularidad de WebAssembly.
Le recordamos: para todos los lectores de "Habr": un descuento de 10.000 rublos al registrarse en cualquier curso de Skillbox con el código de promoción "Habr".
Skillbox recomienda: Curso práctico "Mobile Developer PRO" .
Wasm se usa actualmente en muchas aplicaciones, desde juegos como Doom 3 hasta aplicaciones web como Autocad y Figma. Wasm también se usa en áreas como la informática sin servidor.
Este artículo proporciona un ejemplo del uso de Wasm para acelerar un servicio web analítico. Para mayor claridad, tomamos una aplicación de trabajo escrita en C, que se compila en WebAssembly. El resultado se utilizará para reemplazar secciones de JS de baja productividad.
Transformación de la aplicación
El ejemplo utilizará el servicio de navegador fastq.bio, que está destinado a genetistas. La herramienta le permite evaluar la calidad de la secuenciación de ADN (decodificación).
Aquí hay una aplicación de ejemplo en el trabajo:

No se deben proporcionar los detalles del proceso, ya que son bastante complicados para los no especialistas, pero en resumen, los científicos de las infografías anteriores pueden entender si el proceso de secuenciación del ADN se realizó sin problemas y qué problemas surgieron.
Este servicio tiene alternativas, programas de escritorio. Pero fastq.bio puede acelerar las cosas visualizando datos. En la mayoría de los otros casos, debe poder trabajar con la línea de comando, pero no todos los genetistas tienen la experiencia necesaria.
Todo funciona de manera simple. En la entrada: datos presentados en forma de archivo de texto. Este archivo es generado por herramientas especializadas de secuenciación. El archivo contiene una lista de secuencias de ADN y una evaluación de calidad para cada nucleótido. El formato del archivo es .fastq, por lo que el servicio obtuvo su nombre.
Implementación de JavaScript
El primer paso del usuario cuando trabaja con fastq.bio es seleccionar el archivo apropiado. Usando el objeto File, la aplicación lee una selección aleatoria de datos del archivo y procesa este paquete. La tarea de JavaScript aquí es realizar operaciones de cadena simples e indicadores de conteo. Uno de ellos es el número de nucleótidos A, C, G y T en diferentes fragmentos de ADN.
Después de calcular los indicadores necesarios, se visualizan usando Plotly.js, y el servicio comienza a trabajar con una nueva selección de datos. La fragmentación se realiza para mejorar la calidad de UX. Si trabaja con todos los datos a la vez, el proceso se congelará por un tiempo, ya que los archivos con resultados de secuencia ocupan cientos de gigabytes de espacio de archivos. El servicio toma datos de 0.5 a 1 Mb de tamaño y trabaja con ellos paso a paso, construyendo datos gráficos.
Así es como funciona:

El rectángulo rojo contiene el algoritmo de conversión de cadenas para renderizar. Esta es la parte más cargada del servicio en términos de computación. Vale la pena intentar reemplazarlo con Wasm.
Prueba de WebAssembly
Para evaluar la posibilidad de usar Wasm, el equipo del proyecto comenzó a buscar soluciones listas para crear métricas de CC (CC - control de calidad) basadas en archivos fastq. La búsqueda se realizó entre las herramientas escritas en C, C ++ o Rust, de modo que fue posible portar el código a WebAssembly. Además, la herramienta no debe ser "en bruto", requiere un servicio ya verificado por los científicos.
Como resultado, la elección se hizo a favor de
seqtk . La aplicación es bastante popular, es de código abierto, el idioma de origen es C.
Antes de convertir a Wasm, debe observar el principio de compilar seqtk para el escritorio. Según el Makefile, esto es lo que necesita:
# Compile to binary $ gcc seqtk.c \ -o seqtk \ -O2 \ -lm \ -lz
Básicamente, puede compilar seqtk con Emscripten. Si no está allí,
recorra la imagen de Docker .
$ docker pull robertaboukhalil/emsdk:1.38.26 $ docker run -dt --name wasm-seqtk robertaboukhalil/emsdk:1.38.26
Si lo desea,
puede recogerlo usted mismo , pero lleva tiempo.
Dentro del contenedor, puede tomar fácilmente emcc como alternativa a gcc:
# Compile to WebAssembly $ emcc seqtk.c \ -o seqtk.js \ -O2 \ -lm \ -s USE_ZLIB=1 \ -s FORCE_FILESYSTEM=1
Cambios mínimos:
En lugar de enviar al archivo binario Emscripten, se utilizan .wasm y .js para generar los archivos, que se utilizan para iniciar el módulo WebAssemby.
Para admitir la biblioteca zlib, se usa el indicador USE_ZLIB. La biblioteca se distribuye y se transfiere a WebAssembly, y Emscripten la incluye en el proyecto.
El sistema de archivos virtual Emscrippten está activado. Este es un
FS similar a POSIX que opera en RAM dentro del navegador. Cuando la página se actualiza, la memoria se borra.
Para comprender por qué se necesita un sistema de archivos virtual, vale la pena comparar la forma de iniciar seqtk desde la línea de comandos con la forma de ejecutar el módulo compilado WebAssembly.
# On the command line $ ./seqtk fqchk data.fastq # In the browser console > Module.callMain(["fqchk", "data.fastq"])
Es necesario obtener acceso al sistema de archivos virtual para no sobrescribir seqtk con cadena en lugar de entrada de archivo. En este caso, el fragmento de datos se muestra como el archivo data.fastq en el FS virtual con el main () seqtk invocado en él.
Aquí está la nueva arquitectura:
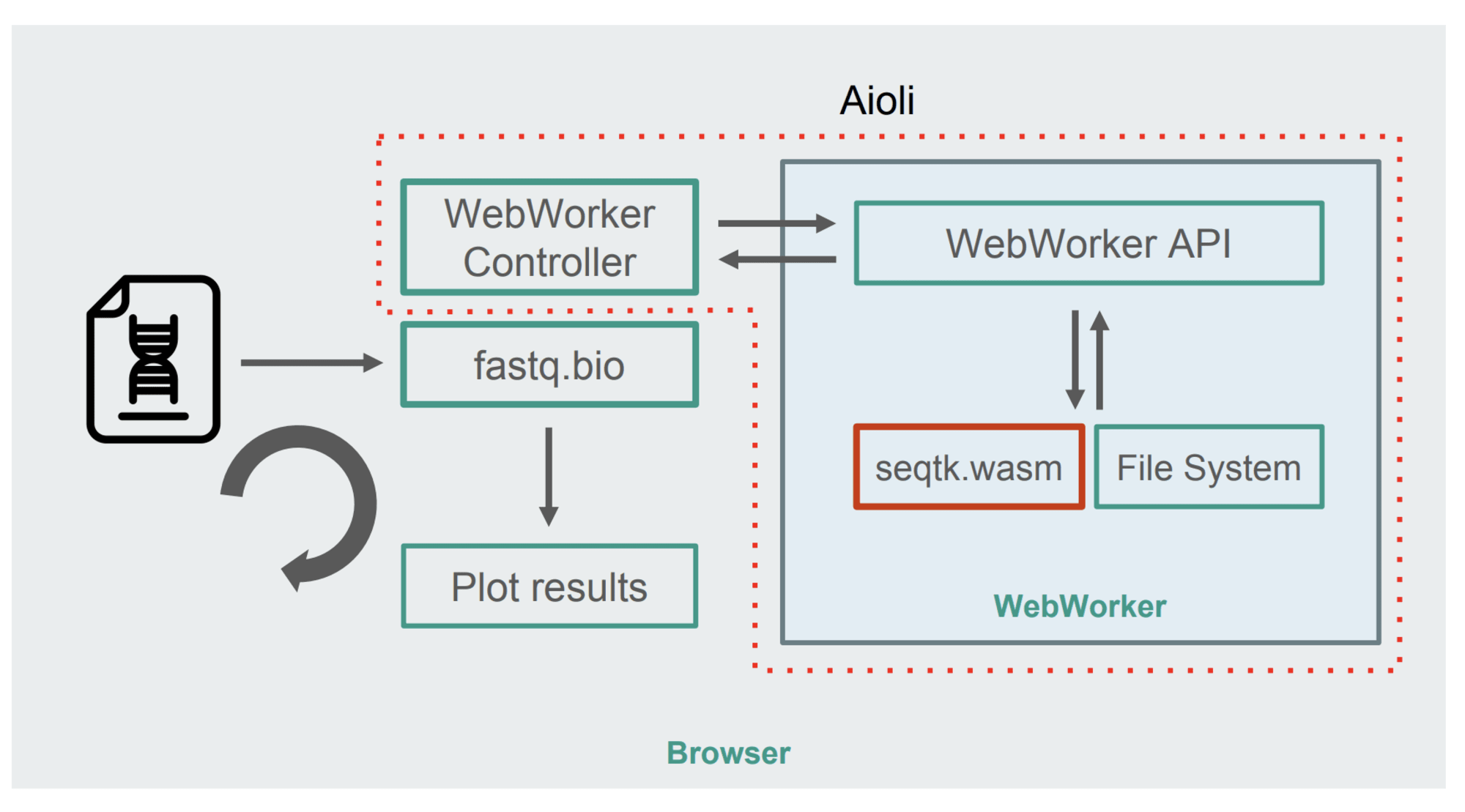
La figura muestra que, en lugar de la informática, la secuencia principal del navegador utiliza
WebWorkers . Este método permite realizar cálculos en el hilo de fondo sin afectar la capacidad de respuesta del navegador. Bueno, el controlador WebWorker lanza Worker, controlando su interacción con el hilo principal.
El comando seqtk se inicia utilizando Worker en el archivo montado. Después de completar el trabajo, Trabajador muestra el resultado en forma de Promesa. Cuando el hilo principal recibe un mensaje, el resultado se utiliza para actualizar las programaciones. Y así, en algunas iteraciones.
¿Qué pasa con el rendimiento de WebAssembly?
Para evaluar el cambio en el rendimiento, el equipo del proyecto utilizó el parámetro del número de operaciones de lectura por segundo. El tiempo de gráficos interactivos no se tiene en cuenta, ya que ambas implementaciones usan JavaScript.
Al usar la solución lista para usar, la ganancia de rendimiento fue nueve veces.

Este es un resultado excelente, pero resultó que existe la oportunidad de optimizarlo. El hecho es que seqtk no utiliza una gran cantidad de resultados de análisis de CC, por lo que puede eliminarlos. Si hace esto, el resultado en comparación con JS se mejora 13 veces.
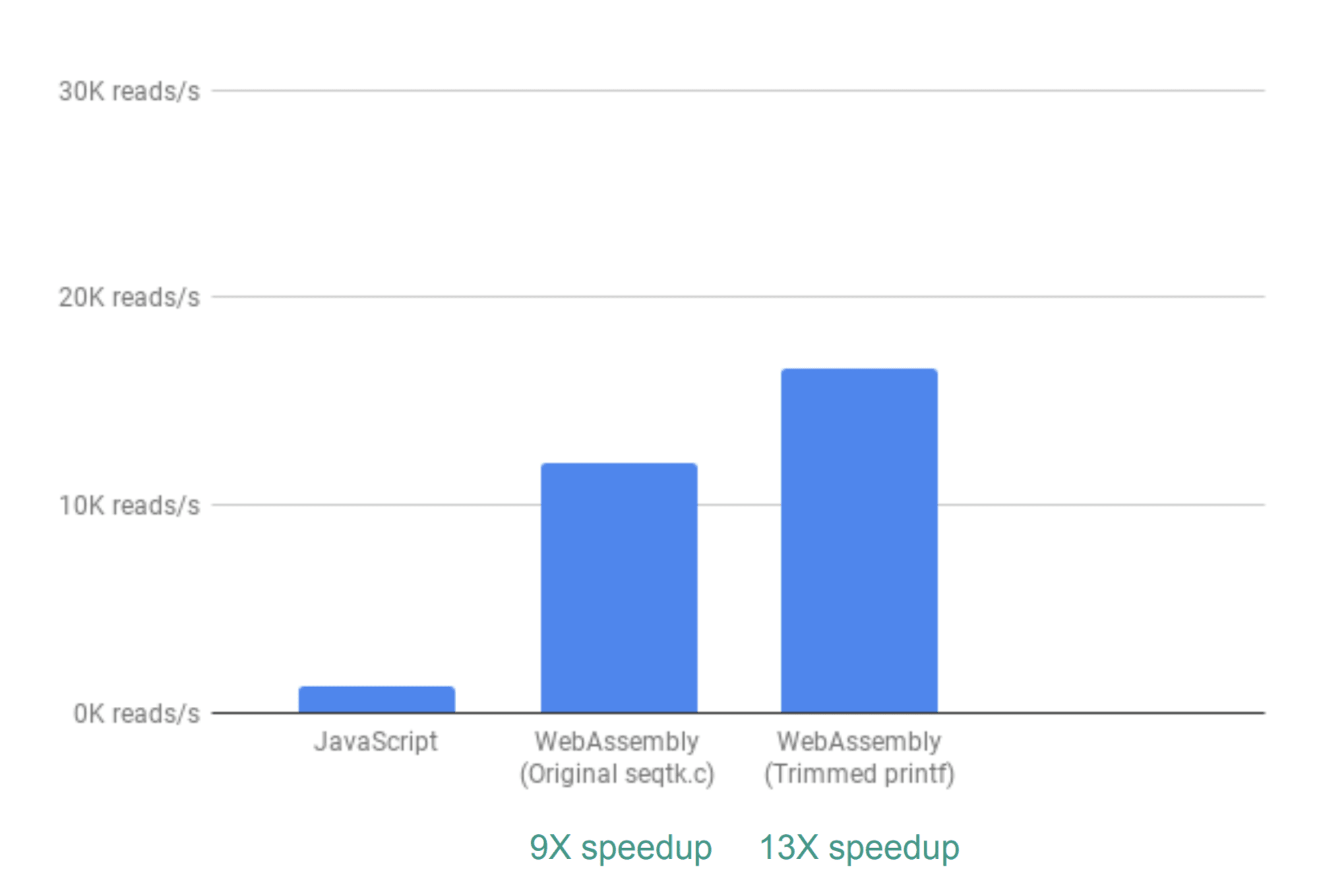
Fue posible lograrlo simplemente comentando los comandos printf ().
Pero eso no es todo. El hecho es que en esta etapa fastq.bio recibe los resultados del análisis llamando a diferentes funciones de C. Cualquiera de ellos calcula su propio conjunto de características, de modo que cada fragmento del archivo se lee dos veces.
Para solucionar este problema, se decidió combinar las dos funciones en una sola. Como resultado, la productividad aumentó en 20 veces.

Vale la pena señalar que un resultado tan sobresaliente no siempre se puede lograr. En algunos casos, el rendimiento disminuye, por lo que vale la pena evaluar cada caso.
Como conclusión, podemos decir que Wasm realmente hace posible mejorar el rendimiento de la aplicación, pero debe usarlo sabiamente.
Skillbox recomienda: