Hola de nuevo Estamos compartiendo una publicación, cuya traducción fue preparada específicamente para estudiantes del curso
"Redes neuronales en Python" .

Hoy hablaremos sobre el primer evento importante en la historia de DeepMind, para mostrar cómo la investigación que usa inteligencia artificial puede estimular la aparición de descubrimientos científicos. Debido a la naturaleza interdisciplinaria de nuestro trabajo, DeepMind reunió a expertos de los campos de la biología estructural, la física y el aprendizaje automático para utilizar métodos avanzados para predecir la estructura tridimensional de una proteína basada únicamente en su secuencia genética.
El sistema AlphaFold en el que hemos estado trabajando durante los últimos dos años se basa en muchos años de experiencia en investigación utilizando amplios datos del genoma para predecir la estructura de la proteína. Los modelos de proteínas tridimensionales que genera AlphaFold son mucho más precisos que los obtenidos anteriormente. Esto marcó un progreso significativo en una de las principales tareas de la biología.
¿Cuál es el problema del plegamiento de proteínas?
Las proteínas son moléculas grandes y complejas necesarias para mantener la vida. Casi todas las funciones de nuestro cuerpo, ya sea la contracción muscular, la percepción de la luz o la conversión de los alimentos en energía, se pueden rastrear a una o más proteínas y cómo se mueven y cambian. Las recetas para estas proteínas, llamadas genes, están codificadas en nuestro ADN.
Las propiedades de una proteína dependen de su estructura tridimensional única. Por ejemplo, las proteínas de anticuerpos que forman nuestro sistema inmunológico tienen "forma de Y" y parecen ganchos especiales. Al aferrarse a los virus y las bacterias, las proteínas de los anticuerpos pueden detectar y etiquetar los patógenos para su posterior destrucción. Del mismo modo, las proteínas de colágeno están en forma de cordones que transmiten tensión entre cartílago, ligamentos, huesos y piel. Otros tipos de proteínas incluyen Cas9, que, guiados por secuencias CRISPR, actúan como tijeras que cortan el ADN e insertan nuevos sitios. Proteínas anticongelantes, cuya estructura tridimensional les permite unirse a los cristales de hielo y evitar la congelación de organismos; y ribosomas, que actúan como un transportador programado que participa en la construcción de proteínas.
Determinar la estructura tridimensional de una proteína únicamente a partir de su secuencia genética es una tarea difícil con la que los científicos han estado luchando durante décadas. El problema es que el ADN contiene solo información sobre la secuencia de bloques de construcción de una proteína llamada residuos de aminoácidos que forman cadenas largas. Predecir cómo estas cadenas formarán una estructura compleja de proteínas 3D se conoce como el "problema de plegamiento de proteínas".
Cuanto más grande es la proteína, más difícil es modelarla, ya que se forman más enlaces entre los aminoácidos que deben tenerse en cuenta. Como se deduce de
la paradoja de Levintal , para enumerar todas las configuraciones posibles de una proteína ordinaria, antes de que se logre su estructura tridimensional correcta, tomará más tiempo del que existe el Universo.

¿Por qué es importante el plegamiento de proteínas?
La capacidad de predecir la forma de la proteína es extremadamente útil porque es fundamental para comprender el papel de la proteína en el cuerpo, así como el diagnóstico y el tratamiento de enfermedades como el Alzheimer, el
Parkinson , la
enfermedad de Huntington y la
fibrosis quística , que los médicos creen que son causadas por proteínas mal plegadas.
Nos complace especialmente que la capacidad de predecir la forma de una proteína pueda mejorar nuestra comprensión de cómo funcionan nuestros cuerpos, y esto nos permitirá desarrollar nuevos medicamentos de manera eficiente. A medida que obtenemos más información sobre las formas de las proteínas y cómo funcionan a través del modelado, se abren nuevas posibilidades para crear medicamentos y disminuye el costo de los experimentos. Finalmente, estos descubrimientos mejorarán la calidad de vida de millones de pacientes en todo el mundo.
Comprender el proceso de plegamiento de proteínas también puede ayudar a desarrollar un tipo de proteína que contribuya significativamente a la realidad circundante. Por ejemplo, los avances logrados mediante el desarrollo de proteínas en el campo de las enzimas biodegradables pueden ayudar a lidiar con contaminantes como el plástico y el aceite, ayudando a descomponer los desechos sin dañar el medio ambiente. De hecho, los investigadores ya han comenzado a
diseñar bacterias que secretan proteínas que hacen que los desechos sean biodegradables y que sean más fáciles de manejar.
Con el fin de estimular la investigación y evaluar el progreso en el campo de los últimos métodos para mejorar la precisión del pronóstico, en 1994 se
lanzó una competencia a gran escala de dos años llamada
Experimento comunitario sobre evaluación crítica de los métodos de predicción de la estructura de la proteína (CASP), que se convirtió en el estándar de oro en los métodos de evaluación.
¿Cómo hará AI la diferencia?
Durante las últimas cinco décadas, los científicos han podido reconocer las formas de proteínas en el laboratorio utilizando métodos experimentales como
la microscopía de crioelectrones ,
la resonancia magnética nuclear o
la difracción de rayos X , pero cada método ha sido deducido por muchos ensayos y errores que tomaron años y costaron decenas de miles de dólares. Es por eso que los biólogos ahora están recurriendo a los métodos de IA como una alternativa al largo y laborioso proceso de investigación de proteínas complejas.
Afortunadamente, el campo de la genómica tiene suficientes datos debido a la rápida reducción en el costo de la secuenciación genética. Como resultado, en los últimos años, los
enfoques para el problema del pronóstico mediante el aprendizaje profundo y basados en datos del genoma se han vuelto cada vez más populares. El trabajo de DeepMind en este tema llevó a la aparición de AlphaFold, que presentamos a CASP este año. Estamos orgullosos de ser parte del progreso que los expertos de CASP llamaron "progreso sin precedentes en la capacidad de los métodos computacionales para predecir la estructura de una proteína". Como resultado, tomamos el
primer lugar en el ranking de equipos (somos A7D).
Nuestro equipo se centró precisamente en la tarea de modelar formas objetivo desde cero, sin utilizar proteínas previamente resueltas como plantillas. Logramos un alto grado de precisión en la predicción de las propiedades físicas de la estructura de la proteína, y luego usamos dos métodos diferentes para predecir las estructuras completas de la proteína.
Usar redes neuronales para predecir propiedades físicas
Ambos métodos utilizaron redes neuronales profundas que están entrenadas para predecir las propiedades de una proteína por su secuencia genética. Las propiedades que predice la red son: (a) la distancia entre pares de aminoácidos y (b) los ángulos entre los enlaces químicos que conectan estos aminoácidos. El primer desarrollo fue un avance real en el uso de métodos populares que determinan si los pares de aminoácidos están uno al lado del otro.
Entrenamos la red neuronal para predecir una distribución separada de distancias entre cada par de residuos de proteínas. Estas probabilidades se combinaron en una estimación que muestra qué tan bien diseñada está la estructura de la proteína. También capacitamos a otra red neuronal que usa todas las distancias en total para evaluar qué tan cerca está la estructura propuesta de la respuesta correcta.

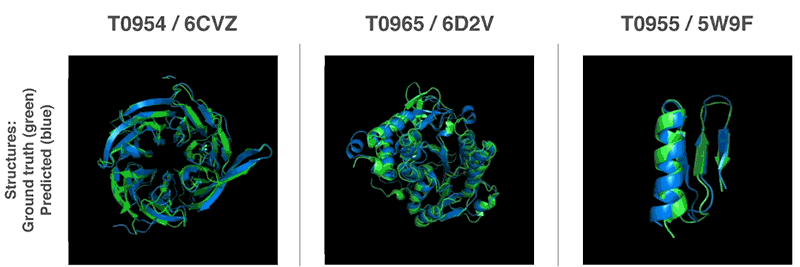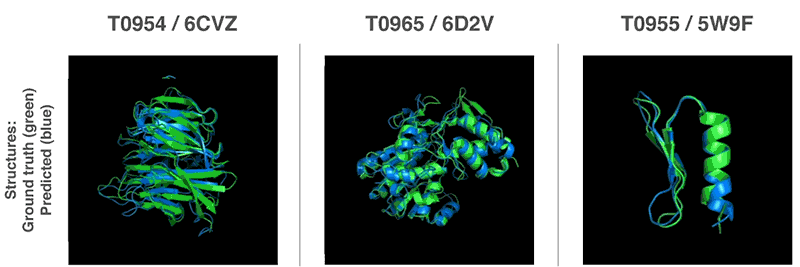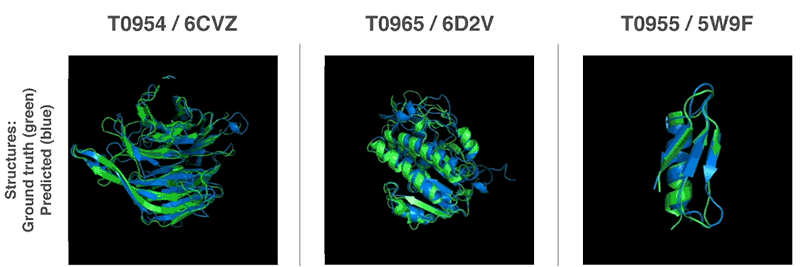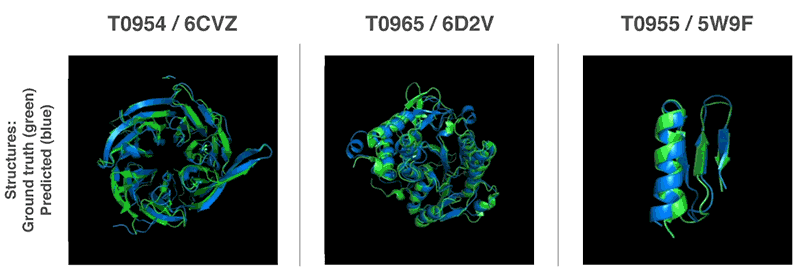
Nuevos métodos para predecir estructuras de proteínas.
Usando estas funciones de valoración, pudimos encontrar estructuras que se ajustan a nuestros pronósticos. Nuestro primer método se basa en métodos ampliamente utilizados en biología estructural; ha reemplazado repetidamente partes de la estructura de la proteína con nuevos fragmentos. Capacitamos a la red neuronal generativa competitiva para proponer nuevos fragmentos que se utilizan para mejorar continuamente la evaluación de la estructura proteica propuesta.

El segundo método optimizó las calificaciones usando el descenso de gradiente (un método matemático comúnmente utilizado en el aprendizaje automático para pequeñas mejoras incrementales), lo que condujo a una alta precisión de las estructuras. Este método se aplicó a cadenas proteicas completas, y no a piezas que deben apilarse por separado antes del ensamblaje, lo que reduce la complejidad del proceso de predicción.
Que sigue
El éxito de nuestra prueba de la pluma de coagulación de proteínas muestra que los sistemas de aprendizaje automático pueden integrar múltiples fuentes de información para ayudar a los científicos a desarrollar rápidamente soluciones creativas a problemas complejos. Ya hemos visto cómo la IA ayuda a las personas a dominar juegos complejos a través de sistemas como
AlphaGo y
AlphaZero , también esperamos que una vez que el avance de la IA ayude a la humanidad a resolver problemas científicos fundamentales.
Es interesante ver el primer progreso en el plegamiento de proteínas, lo que demuestra la utilidad de la IA en los descubrimientos científicos. Aunque todavía tenemos mucho por hacer, entendemos claramente que podremos contribuir a la búsqueda de tratamiento de diversas enfermedades, ayudar al medio ambiente y mucho más, porque de hecho, el potencial es enorme. Con un equipo dedicado enfocado en explorar cómo el aprendizaje automático puede avanzar en el mundo de la ciencia, exploraremos las diversas formas y métodos por los cuales nuestra tecnología puede afectar el mundo que nos rodea.