Me encontré con
un artículo en el blog de la empresa School of Data y decidí comprobar de qué es capaz la biblioteca Fast.ai en el mismo conjunto de datos que se menciona en el artículo. Aquí no encontrará argumentos sobre la importancia de diagnosticar la neumonía de manera oportuna y correcta, si se necesitarán radiólogos en las condiciones de desarrollo tecnológico, si la predicción de una red neuronal puede considerarse un diagnóstico médico, etc. El objetivo principal es mostrar que el aprendizaje automático en las bibliotecas modernas puede ser bastante simple (literalmente requiere unas pocas líneas de código) y ofrece excelentes resultados. Recordemos el resultado del artículo (precisión = 0.84, recuerdo = 0.96) y veamos qué pasa con nosotros.
Tomamos los datos para el entrenamiento
desde aquí . Los datos son 5856 rayos X distribuidos en dos clases, con o sin signos de neumonía. La tarea de la red neuronal es darnos un clasificador binario de alta calidad de imágenes de rayos X para determinar los signos de neumonía.
Comenzamos importando las bibliotecas y algunas configuraciones estándar:
%reload_ext autoreload %autoreload 2 %matplotlib inline from fastai.vision import * from fastai.metrics import error_rate import os
Luego, determine el tamaño del lote. Al aprender en la GPU, es importante elegirlo de tal manera que su memoria no esté llena. Si es necesario, se puede reducir a la mitad.
bs = 64
Actualización importante:Como se señala correctamente en los comentarios a continuación, es importante monitorear claramente los datos sobre los cuales se capacitará el modelo y sobre los cuales probaremos su efectividad. Entrenaremos el modelo en las imágenes en las carpetas train y val, y validaremos en las imágenes en la carpeta de prueba, similar a lo que se hizo
aquí .
Determinamos las rutas a nuestros datos.
path = Path('storage/chest_xray') path.ls()
y verifique que todas las carpetas estén en su lugar (la carpeta val se ha movido para entrenar):
Out: [PosixPath('storage/chest_xray/train'), PosixPath('storage/chest_xray/test')]
Estamos preparando nuestros datos para "cargarlos" en la red neuronal. Es importante tener en cuenta que en Fast.ai hay varios métodos para hacer coincidir la etiqueta de la imagen. El método from_folder nos dice que las etiquetas deben tomarse del nombre de la carpeta en la que se encuentra la imagen.
El parámetro de tamaño significa que redimensionamos todas las imágenes a un tamaño de 299x299 (nuestros algoritmos funcionan con imágenes cuadradas). La función get_transforms nos brinda un aumento de imagen para aumentar la cantidad de datos de entrenamiento (dejamos aquí la configuración predeterminada).
np.random.seed(5) data = ImageDataBunch.from_folder(path, train = 'train', valid = 'test', size=299, bs=bs, ds_tfms=get_transforms()).normalize(imagenet_stats)
Veamos los datos:
data.show_batch(rows=3, figsize=(6,6))
Para verificar, observamos qué clases obtuvimos y qué distribución cuantitativa de imágenes entre el tren y la validación:
data.classes, data.c, len(data.train_ds), len(data.valid_ds)
Out: (['NORMAL', 'PNEUMONIA'], 2, 5232, 624)
Definimos un modelo de entrenamiento basado en la arquitectura Resnet50:
learn = cnn_learner(data, models.resnet50, metrics=error_rate)
y comience a aprender en 8 eras según la
Política de un ciclo :
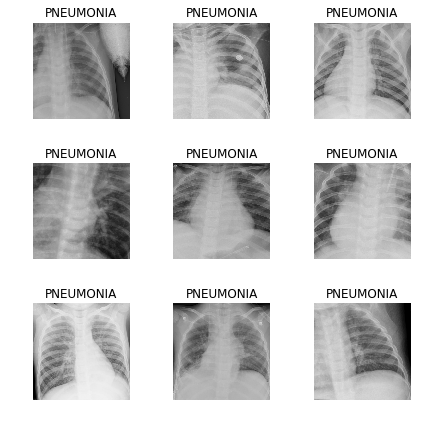
learn.fit_one_cycle(8)
Vemos que ya hemos obtenido una precisión del 89% en la muestra de validación. Anotaremos los pesos de nuestro modelo por ahora e intentaremos mejorar el resultado.
learn.save('step-1-50')
"Descongela" todo el modelo, porque antes de eso, entrenamos el modelo solo en el último grupo de capas, y los pesos del resto se tomaron del modelo pre-entrenado en Imagenet y "congelado":
learn.unfreeze()
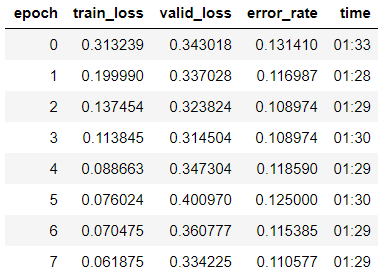
Estamos buscando la tasa de aprendizaje óptima para continuar aprendiendo:
learn.lr_find() learn.recorder.plot()
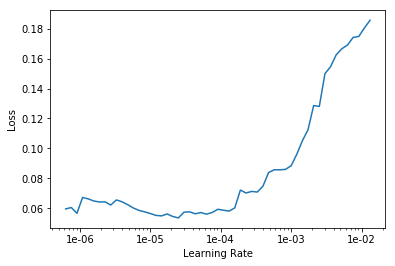
Comenzamos a entrenar durante 10 eras con diferentes tasas de aprendizaje para cada grupo de capas.
learn.fit_one_cycle(10, max_lr=slice(1e-6, 1e-4))
Vemos que la precisión de nuestro modelo aumentó ligeramente a 89.4% en la muestra de validación.
Anotamos los pesos.
learn.save('step-2-50')
Construir matriz de confusión:
interp = ClassificationInterpretation.from_learner(learn) interp.plot_confusion_matrix()
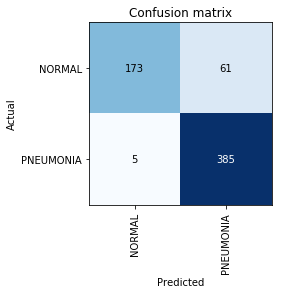
En este punto, recordamos que el parámetro de precisión por sí solo es insuficiente, especialmente para las clases desequilibradas. Por ejemplo, si en la vida real la neumonía ocurre solo en el 0.1% de los que se someten a un examen de rayos X, el sistema simplemente puede detectar la ausencia de neumonía en todos los casos y su precisión estará en el nivel del 99.9% con una utilidad absolutamente cero.
Aquí es donde entran en juego las métricas de precisión y recuperación:
- TP - predicción positiva verdadera;
- TN - predicción negativa verdadera;
- FP - predicción falsa positiva;
- FN - Predicción falsa negativa.
Precisión=TP/(TP+FP)=385/446=0.863
Recall=TP/(TP+FN)=385/390=$0.98
Vemos que el resultado que obtuvimos es incluso un poco más alto que el mencionado en el artículo. En el trabajo adicional en la tarea, vale la pena recordar que Recall es un parámetro extremadamente importante en problemas médicos, porque Los falsos errores negativos son los más peligrosos desde el punto de vista del diagnóstico (lo que significa que simplemente podemos "pasar por alto" un diagnóstico peligroso).