
عند كتابة مقال عن تطوير كاشف الشذوذ ، قمت بتطبيق إحدى الخوارزميات تسمى الغاز العصبي المتزايد المتزايد.
في الأدب السوفيتي الجزء الروسي من الإنترنت ، يتم تغطية هذا الموضوع بشكل سيئ إلى حد ما ، وكان هناك مقال واحد فقط ، وحتى ذلك الحين مع تطبيق هذه الخوارزمية.
إذن ما هي بالضبط خوارزمية الغاز العصبي المتزايد المتزايدة؟
مقدمة
IGNG ، مثل GNG ، هو خوارزمية تجميعية تكيفية.
تم وصف الخوارزمية نفسها في مقالة كتبها Prudent و Ennadji لعام 2005 .
مثل GNG ، هناك العديد من ناقلات البيانات X أو توليد وظيفة f(t) ، الذي يوفر ناقلات من البيانات الموزعة بشكل عشوائي (المعلمة t - الوقت أو رقم العينة في العينة).
لا تفرض الخوارزمية قيودًا إضافية على هذه البيانات.
لكن في الداخل يختلف تمامًا عن GNG.
هذه الخوارزمية مثيرة للاهتمام أيضًا لأنها أكثر دقة قليلاً من تكوين الخلايا العصبية لنماذج GNG.
وصف الخوارزمية
تقسم الخوارزمية الكثير من البيانات إلى مجموعات.
بالمقارنة مع GNG ، ميزته هي معدل تقارب أعلى.
الأفكار التي تستند إليها الخوارزمية:
- نظرية الرنين التكيفي : أولاً ، يتم البحث عن أقرب خلية عصبية ، وإذا لم يتجاوز الفرق العتبة ("معلمة اليقظة") ، يتم تعديل الأوزان ، أو يتم تغيير إحداثيات الخلايا العصبية في مساحة البيانات. إذا لم يتم تجاوز العتبة ، يتم إنشاء الخلايا العصبية الجديدة التي تقارب بشكل أفضل قيمة عينة البيانات.
- تحتوي كل من الاتصالات والخلايا العصبية على معلمة عمر (لدى GNG اتصالات فقط) ، وهي في البداية صفر ، ولكنها تزداد كلما تعلمت.
- لا يظهر العصبون على الفور: أولاً يظهر جنين (أو العصبون الجرثومي) ، ويزداد سنه مع كل تكرار ، حتى ينضج. بعد التدريب ، تشارك فقط الخلايا العصبية الناضجة في التصنيف .
الدورة الرئيسية
يبدأ العمل برسم بياني فارغ. معلمة سيجما تم التهيئة من خلال الانحراف المعياري لعينة التدريب:
sigma= sqrt frac1N sum limitsNi=1 left(xi− barx right)2
أين: barx - المتوسط بين إحداثيات العينة.
تقلل الحلقة الرئيسية في كل خطوة من القيمة سيجما ، وهو حد القرب ، ويحسب الفرق بين المستوى السابق لجودة التجميع والمستوى الذي تم الحصول عليه بعد التجميع بواسطة إجراء IGNG .
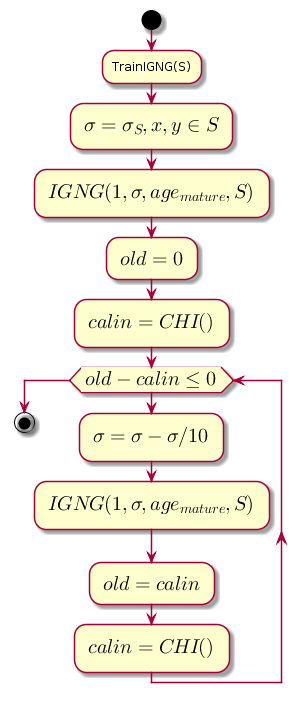
رمز المخطط.@startuml start :TrainIGNG(S); :<latex>\sigma = \sigma_S,x,y \in S</latex>; :<latex>IGNG(1, \sigma, age_{mature}, S)</latex>; :<latex>old = 0</latex>; :<latex>calin = CHI()</latex>; while (<latex>old - calin \leq 0</latex>) :<latex>\sigma=\sigma - \sigma / 10</latex>; :<latex>IGNG(1, \sigma, age_{mature}, S)</latex>; :<latex>old = calin</latex>; :<latex>calin = CHI()</latex>; endwhile stop @enduml
CHI هو مؤشر Kalinsky-Kharabaz الذي يوضح جودة التكتل:
CHI= fracB/(c−1)W/(n−c)
أين:
- n - عدد عينات البيانات.
- c - عدد العناقيد (في هذه الحالة عدد الخلايا العصبية).
- B - مصفوفة التشتت الداخلي (مجموع المسافات المربعة بين إحداثيات الخلايا العصبية ومتوسط جميع البيانات).
- W - مصفوفة التشتت الخارجي (مجموع المسافات المربعة بين جميع البيانات وأقرب خلية عصبية).
كلما كانت قيمة المؤشر أكبر ، كلما كان ذلك أفضل ، لأنه إذا كان الفرق بين المؤشرات بعد التجميع وقبله سلبيًا ، فمن الممكن أن نفترض أن المؤشر أصبح إيجابيًا وتجاوز المؤشر السابق ، أي تم التجميع بنجاح.
إجراء IGNG
هذا هو الإجراء الأساسي للخوارزمية.
وهي مقسمة إلى ثلاث مراحل متنافية:
- لم يتم العثور على الخلايا العصبية.
- تم العثور على خلية عصبية مرضية.
- وجدت اثنتين تفيان بظروف العصبون.
في حالة مرور أحد الشروط ، لا يتم تنفيذ الخطوات الأخرى.
أولاً ، يتم البحث عن الخلايا العصبية للحصول على أفضل عينة بيانات تقريبية:
c1=min(dist( xi، omegac))
هنا dist(x omega،x xi) - دالة حساب المسافة ، والتي عادة ما تكون مقياسًا إقليديًا .
إذا لم يتم العثور على العصبون ، أو أنه بعيد جدًا عن البيانات ، أي لا يفي بمعيار القرب dist( xi، omegac) leq sigma ، يتم إنشاء عصبون جنيني جديد بإحداثيات تساوي إحداثيات العينة في مساحة البيانات.

إذا اجتاز فحص القرب ، يتم البحث عن خلية عصبية ثانية بنفس الطريقة ويتم التحقق من القرب من عينة البيانات.
إذا لم يتم العثور على العصبون الثاني ، يتم إنشاؤه.

إذا تم العثور على اثنين من الخلايا العصبية التي تلبي حالة القرب من عينة البيانات ، يتم تصحيح إحداثياتهم وفقًا للصيغة التالية:
epsilon(t)hc،ci= startcases epsilonb، if\،c=ci epsilonn، if\،هناك ،الاتصال ،بين ،c=ci0،\،in\،other\،case endcases
Delta omegac= epsilon(t)hc،c1\allel xi− omegac\allel omegac= omegac+ Delta omegac
أين:
- epsilon(t) - خطوة التكيف.
- ci هو عدد الخلايا العصبية.
- hc،c1 - وظيفة جوار العصبون c مع الخلايا العصبية الفائزة (في هذه الحالة ، ستُرجع 1 للجيران المباشرين ، 0 خلاف ذلك ، لأن خطوة التكيف ، للحساب omega ستكون غير صفرية فقط للجيران المباشرين).
وبعبارة أخرى ، يتم تغيير إحداثية (وزن) العصبون الفائز إلى epsilonb∗ Delta omegai ، وجميع جيرانها المباشرين (أولئك المرتبطين بها من حافة الرسم البياني) على epsilonn∗ Delta omegai أين omegai - إحداثيات الخلايا العصبية المقابلة قبل التغيير.
ثم يتم إنشاء اتصال بين الخلايا العصبية الفائزة ، وإذا تم إنشاؤه بالفعل ، فسيتم إعادة تعيين عمره.
عمر جميع العلاقات الأخرى آخذ في الازدياد.
جميع الاتصالات التي تجاوز سنها الثابت agemax يتم حذفها.
بعد ذلك ، تتم إزالة جميع الخلايا العصبية الناضجة (تلك التي ليس لها اتصال مع الآخرين).
يتزايد عمر الخلايا العصبية المباشرة المجاورة للخلايا العصبية الفائزة.
إذا تجاوز عمر أي من الخلايا العصبية الجرثومية agemature يصبح عصبون ناضج.
يحتوي الرسم البياني النهائي فقط على الخلايا العصبية الناضجة.
يمكن اعتبار شرط إكمال إجراء IGNG أدناه عددًا ثابتًا من الدورات.
تظهر الخوارزمية أدناه (الصورة قابلة للنقر):
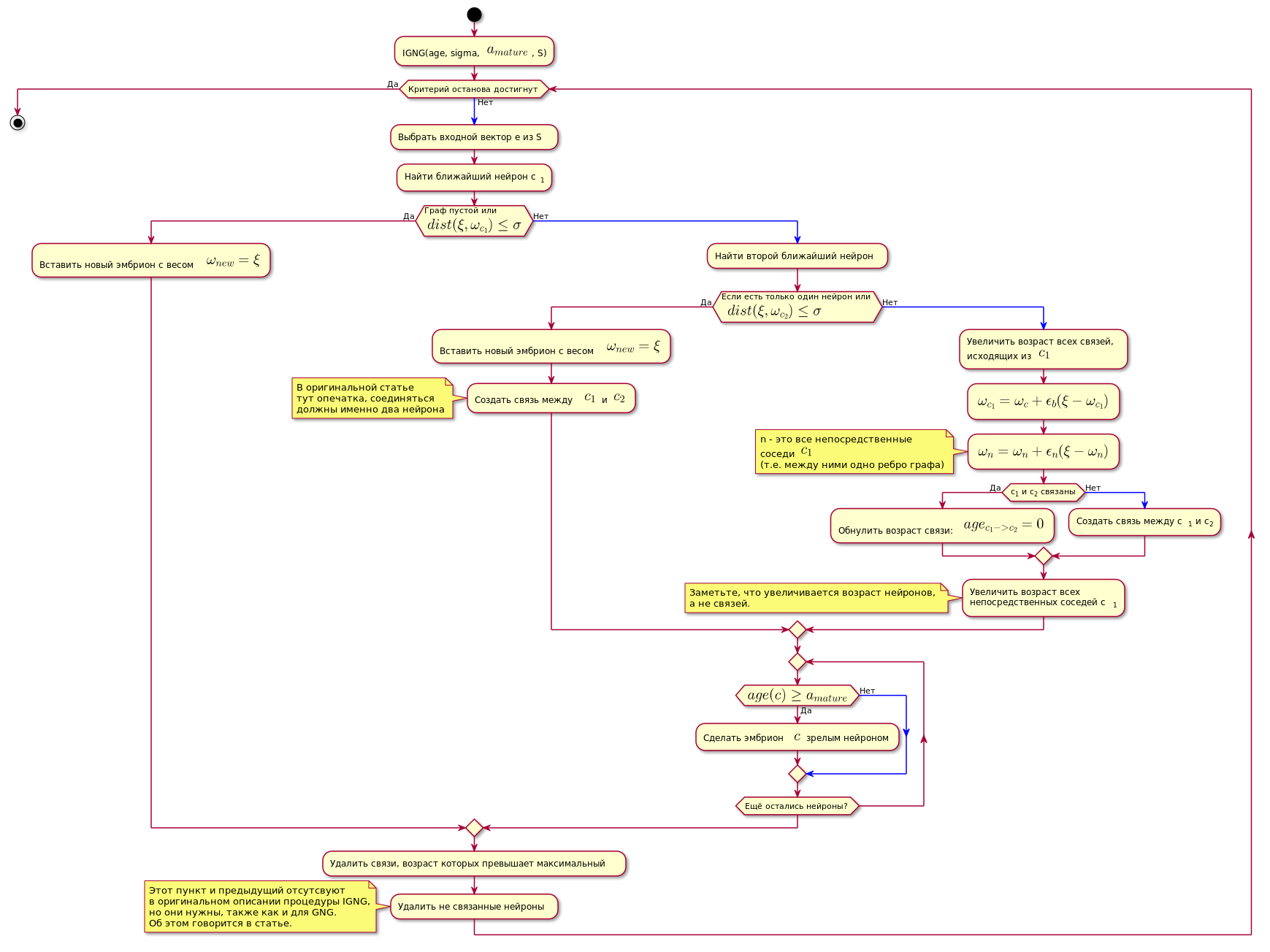
رمز المخطط. @startuml skinparam nodesep 10 skinparam ranksep 20 start :IGNG(age, sigma, <latex>a_{mature}</latex>, S); while ( ) is () -[#blue]-> : e S; : c<sub>1</sub>; if ( \n<latex>dist(\xi, \omega_{c_1}) \leq \sigma</latex>) then () : <latex>\omega_{new} = \xi</latex>; else () -[#blue]-> : ; if ( \n <latex>dist(\xi, \omega_{c_2}) \leq \sigma</latex>) then () : <latex>\omega_{new} = \xi</latex>; : <latex>c_1</latex> <latex>c_2</latex>; note , end note else () -[#blue]-> : ,\n <latex>c_1</latex>; :<latex>\omega_{c_1} = \omega_c + \epsilon_b(\xi - \omega_{c_1})</latex>; :<latex>\omega_n = \omega_n + \epsilon_n(\xi - \omega_n)</latex>; note n - <latex>c_1</latex> (.. ) end note if (c<sub>1</sub> c<sub>2</sub> ) then () : : <latex>age_{c_1 -> c_2} = 0</latex>; else () -[#blue]-> : c<sub>1</sub> c<sub>2</sub>; endif : \n c<sub>1</sub>; note , , . end note endif repeat if (<latex>age(c) \geq a_{mature}</latex>) then () : $$ ; else () -[#blue]-> endif repeat while ( ?) endif : , ; : ; note IGNG, , GNG. . endnote endwhile () stop @enduml
التنفيذ
تم تنفيذ الشبكة في Python باستخدام مكتبة الرسم البياني NetworkX . يتم إعطاء قطع الرمز من النموذج الأولي في المقالة السابقة أدناه. هناك أيضًا تفسيرات موجزة للرمز.
إذا كان أي شخص مهتمًا بالكود الكامل ، فإليك رابطًا إلى المستودع .
مثال على الخوارزمية:

الجزء الأكبر من التعليمات البرمجية class NeuralGas(): __metaclass__ = ABCMeta def __init__(self, data, surface_graph=None, output_images_dir='images'): self._graph = nx.Graph() self._data = data self._surface_graph = surface_graph