
مرحبا يا هبر!
في الخريف الماضي في Kaggle ، تم إجراء مسابقة لتصنيف صور Quick Draw Doodle Recognition مرسومة باليد ، شارك فيها فريق من R-schiks يتكون من
Artem Klevtsov و
Philip Upravitelev و
Andrey Ogurtsov . لن نصف المسابقة بالتفصيل ، لقد تم ذلك بالفعل في
منشور حديث .
هذه المرة لم تكن هناك ميداليات في المزرعة ، ولكن اكتسبت خبرة قيمة كبيرة ، لذلك أود أن أخبر المجتمع عن عدد من أكثر الأشياء إثارة للاهتمام والمفيدة في Kagl وفي العمل اليومي. من بين الموضوعات التي تم تناولها: الحياة الصعبة بدون
OpenCV ، تحليل JSONs (توضح هذه الأمثلة تكامل كود C ++ في البرامج النصية أو الحزم في R باستخدام
Rcpp ) ، تحديد معلمات البرامج النصية وإرساء الحل النهائي. كل رمز من الرسالة في شكل مناسب للإطلاق متاح في
المستودع .
المحتويات:
- تحميل البيانات الفعال من CSV إلى قاعدة بيانات MonetDB
- إعداد الدفعات
- مكررون لتفريغ الدفعات من قاعدة البيانات
- اختيار نموذج العمارة
- معلمة البرنامج النصي
- الإرساء مخطوطات
- استخدام وحدات معالجة الرسومات المتعددة في Google Cloud
- بدلا من الاستنتاج
1. فعالية تحميل البيانات من CSV إلى قاعدة بيانات MonetDB
لا يتم توفير البيانات في هذه المسابقة في صورة صور جاهزة ، ولكن في شكل ملفات CSV 340 (ملف واحد لكل فئة) تحتوي على JSONs مع إحداثيات نقطة. عند ربط هذه النقاط بالخطوط ، نحصل على الصورة النهائية بحجم 256 × 256 بكسل. وأيضًا ، بالنسبة لكل سجل ، يتم تقديم تسمية ما إذا كان قد تم التعرف على الصورة بشكل صحيح من قبل المصنف المستخدم في وقت جمع مجموعة البيانات ، والكود المكون من حرفين في بلد إقامة المؤلف ، ومعرف فريد ، وختم زمني واسم فئة يطابق اسم الملف. يزن الإصدار المبسط للبيانات المصدر 7.4 غيغابايت في الأرشيف ونحو 20 غيغابايت بعد التفريغ ، أما البيانات الكاملة بعد التفريغ فتستغرق 240 غيغابايت. يضمن المنظمون أن كلا الإصدارين يعاد إنتاج نفس الرسومات ، أي أن النسخة الكاملة زائدة عن الحاجة. على أي حال ، فإن تخزين 50 مليون صورة في ملفات الرسومات أو في المصفوفات كان يعتبر على الفور غير مربح ، وقررنا دمج جميع ملفات CSV من أرشيف train_simplified.zip في قاعدة بيانات مع الجيل التالي من الصور بالحجم المناسب أثناء التنقل لكل دفعة .
تم اختيار MonetDB الراسخ باعتباره DBMS ، أي تنفيذ R في شكل حزمة MonetDBLite . تتضمن الحزمة إصدارًا مضمنًا من خادم قاعدة البيانات ويسمح لك برفع الخادم مباشرةً من جلسة العمل R والعمل معه هناك. يتم إنشاء قاعدة بيانات والاتصال بها بواسطة أمر واحد:
con <- DBI::dbConnect(drv = MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR"))
سنحتاج إلى إنشاء جدولين: أحدهما لكل البيانات والآخر للمعلومات العامة حول الملفات التي تم تنزيلها (مفيد في حالة حدوث خطأ ويجب استئناف العملية بعد تحميل عدة ملفات):
إنشاء الجداول if (!DBI::dbExistsTable(con, "doodles")) { DBI::dbCreateTable( con = con, name = "doodles", fields = c( "countrycode" = "char(2)", "drawing" = "text", "key_id" = "bigint", "recognized" = "bool", "timestamp" = "timestamp", "word" = "text" ) ) } if (!DBI::dbExistsTable(con, "upload_log")) { DBI::dbCreateTable( con = con, name = "upload_log", fields = c( "id" = "serial", "file_name" = "text UNIQUE", "uploaded" = "bool DEFAULT false" ) ) }
كانت أسرع طريقة لتحميل البيانات في قاعدة البيانات هي نسخ ملفات CSV مباشرة باستخدام SQL - الأمر COPY OFFSET 2 INTO tablename FROM path USING DELIMITERS ',','\\n','\"' NULL AS '' BEST EFFORT يستخدم COPY OFFSET 2 INTO tablename FROM path USING DELIMITERS ',','\\n','\"' NULL AS '' BEST EFFORT ، حيث tablename هو اسم الجدول path هو المسار إلى الملف ، فيما بعد ، تم اكتشاف طريقة أخرى لزيادة السرعة: فقط BEST EFFORT بـ LOCKED BEST EFFORT . عند العمل مع الأرشيف ، اتضح أن تطبيق unzip المدمج في R لا يعمل بشكل صحيح مع عدد من الملفات من الأرشيف ، لذلك استخدمنا نظام unzip (باستخدام getOption("unzip") ).
وظيفة للكتابة إلى قاعدة البيانات #' @title #' #' @description #' CSV- ZIP- #' #' @param con ( `MonetDBEmbeddedConnection`). #' @param tablename . #' @oaram zipfile ZIP-. #' @oaram filename ZIP-. #' @param preprocess , . #' `data` ( `data.table`). #' #' @return `TRUE`. #' upload_file <- function(con, tablename, zipfile, filename, preprocess = NULL) { # checkmate::assert_class(con, "MonetDBEmbeddedConnection") checkmate::assert_string(tablename) checkmate::assert_string(filename) checkmate::assert_true(DBI::dbExistsTable(con, tablename)) checkmate::assert_file_exists(zipfile, access = "r", extension = "zip") checkmate::assert_function(preprocess, args = c("data"), null.ok = TRUE) # path <- file.path(tempdir(), filename) unzip(zipfile, files = filename, exdir = tempdir(), junkpaths = TRUE, unzip = getOption("unzip")) on.exit(unlink(file.path(path))) # if (!is.null(preprocess)) { .data <- data.table::fread(file = path) .data <- preprocess(data = .data) data.table::fwrite(x = .data, file = path, append = FALSE) rm(.data) } # CSV sql <- sprintf( "COPY OFFSET 2 INTO %s FROM '%s' USING DELIMITERS ',','\\n','\"' NULL AS '' BEST EFFORT", tablename, path ) # DBI::dbExecute(con, sql) # DBI::dbExecute(con, sprintf("INSERT INTO upload_log(file_name, uploaded) VALUES('%s', true)", filename)) return(invisible(TRUE)) }
في حال كنت بحاجة إلى تحويل الجدول قبل الكتابة إلى قاعدة البيانات ، يكفي تمرير الوظيفة التي ستحول البيانات إلى وسيطة preprocess .
رمز للتحميل التسلسلي للبيانات في قاعدة البيانات:
كتابة البيانات إلى قاعدة البيانات # files <- unzip(zipfile, list = TRUE)$Name # , to_skip <- DBI::dbGetQuery(con, "SELECT file_name FROM upload_log")[[1L]] files <- setdiff(files, to_skip) if (length(files) > 0L) { # tictoc::tic() # pb <- txtProgressBar(min = 0L, max = length(files), style = 3) for (i in seq_along(files)) { upload_file(con = con, tablename = "doodles", zipfile = zipfile, filename = files[i]) setTxtProgressBar(pb, i) } close(pb) # tictoc::toc() } # 526.141 sec elapsed - SSD->SSD # 558.879 sec elapsed - USB->SSD
قد يختلف وقت تحميل البيانات حسب خصائص سرعة محرك الأقراص المستخدم. في حالتنا ، تستغرق القراءة والكتابة داخل SSD نفسه أو من محرك أقراص فلاش USB (ملف مصدر) إلى SSD (قاعدة بيانات) أقل من 10 دقائق.
يستغرق الأمر بضع ثوانٍ إضافية لإنشاء عمود مع تسمية فئة عدد صحيح وعمود فهرس ( ORDERED INDEX ) مع أرقام الأسطر ، والتي سيتم استخدامها لتحديد الحالات عند إنشاء دفعات:
إنشاء أعمدة إضافية وفهرس message("Generate lables") invisible(DBI::dbExecute(con, "ALTER TABLE doodles ADD label_int int")) invisible(DBI::dbExecute(con, "UPDATE doodles SET label_int = dense_rank() OVER (ORDER BY word) - 1")) message("Generate row numbers") invisible(DBI::dbExecute(con, "ALTER TABLE doodles ADD id serial")) invisible(DBI::dbExecute(con, "CREATE ORDERED INDEX doodles_id_ord_idx ON doodles(id)"))
لحل مشكلة إنشاء دفعة "سريعًا" ، احتجنا إلى تحقيق أقصى سرعة لاستخراج سلاسل عشوائية من جدول doodles . لهذا استخدمنا 3 الحيل. الأول هو تقليل بُعد النوع الذي يتم تخزين معرف الملاحظة فيه. في مجموعة البيانات الأصلية ، يكون نوع bigint مطلوبًا لتخزين المعرف ، لكن عدد المشاهدات يسمح بتركيب معرفاتها مساوية للرقم التسلسلي في النوع int . البحث أسرع بكثير. الخدعة الثانية كانت استخدام ORDERED INDEX - تم اتخاذ هذا القرار بشكل تجريبي ، حيث تم فرز جميع الخيارات المتاحة. والثالث هو استخدام استعلامات ذات معلمات. يكمن جوهر الأسلوب في تنفيذ أمر PREPARE مرة واحدة ثم استخدام التعبير المُعد لإنشاء كومة من نفس النوع من الاستعلامات ، ولكن في الواقع فإن الكسب مقارنةً بـ SELECT البسيط SELECT في منطقة الخطأ الإحصائي.
تستهلك عملية ملء البيانات أكثر من 450 ميغابايت من ذاكرة الوصول العشوائي. بمعنى أن الطريقة الموضحة تسمح لك بتدوير مجموعات البيانات التي تزن عشرات الجيجابايت على أي أجهزة ميزانية تقريبًا ، بما في ذلك بعض أجهزة الكمبيوتر ذات اللوحة الواحدة ، والتي تعتبر رائعة جدًا.
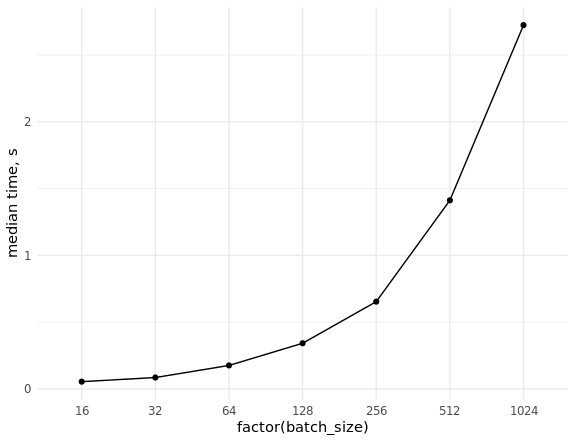
يبقى أن نأخذ قياسات معدل استخراج البيانات (العشوائية) وتقييم القياس عند أخذ عينات من أحجام مختلفة:
قاعدة بيانات المعيار library(ggplot2) set.seed(0) # con <- DBI::dbConnect(MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR")) # prep_sql <- function(batch_size) { sql <- sprintf("PREPARE SELECT id FROM doodles WHERE id IN (%s)", paste(rep("?", batch_size), collapse = ",")) res <- DBI::dbSendQuery(con, sql) return(res) } # fetch_data <- function(rs, batch_size) { ids <- sample(seq_len(n), batch_size) res <- DBI::dbFetch(DBI::dbBind(rs, as.list(ids))) return(res) } # res_bench <- bench::press( batch_size = 2^(4:10), { rs <- prep_sql(batch_size) bench::mark( fetch_data(rs, batch_size), min_iterations = 50L ) } ) # cols <- c("batch_size", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # batch_size min median max `itr/sec` total_time n_itr # <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 16 23.6ms 54.02ms 93.43ms 18.8 2.6s 49 # 2 32 38ms 84.83ms 151.55ms 11.4 4.29s 49 # 3 64 63.3ms 175.54ms 248.94ms 5.85 8.54s 50 # 4 128 83.2ms 341.52ms 496.24ms 3.00 16.69s 50 # 5 256 232.8ms 653.21ms 847.44ms 1.58 31.66s 50 # 6 512 784.6ms 1.41s 1.98s 0.740 1.1m 49 # 7 1024 681.7ms 2.72s 4.06s 0.377 2.16m 49 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = 1)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() DBI::dbDisconnect(con, shutdown = TRUE)
2. إعداد دفعات
تتكون العملية الكاملة لإعداد الدفعات من الخطوات التالية:
- تحليل JSONs متعددة تحتوي على متجهات خط مع إحداثيات نقطة.
- رسم خطوط ملونة بإحداثيات النقاط في الصورة بالحجم المطلوب (على سبيل المثال ، 256 × 256 أو 128 × 128).
- تحويل الصور الناتجة إلى موتر.
في إطار المنافسة بين النواة في بيثون ، تم حل المشكلة بشكل رئيسي عن طريق OpenCV . أحد النظير الأكثر بساطة والأكثر وضوحًا على R سيبدو كما يلي:
تطبيق JSON لتحويل الموتر على R r_process_json_str <- function(json, line.width = 3, color = TRUE, scale = 1) { # JSON coords <- jsonlite::fromJSON(json, simplifyMatrix = FALSE) tmp <- tempfile() # on.exit(unlink(tmp)) png(filename = tmp, width = 256 * scale, height = 256 * scale, pointsize = 1) # plot.new() # plot.window(xlim = c(256 * scale, 0), ylim = c(256 * scale, 0)) # cols <- if (color) rainbow(length(coords)) else "#000000" for (i in seq_along(coords)) { lines(x = coords[[i]][[1]] * scale, y = coords[[i]][[2]] * scale, col = cols[i], lwd = line.width) } dev.off() # 3- res <- png::readPNG(tmp) return(res) } r_process_json_vector <- function(x, ...) { res <- lapply(x, r_process_json_str, ...) # 3- 4- res <- do.call(abind::abind, c(res, along = 0)) return(res) }
يتم تنفيذ الرسم باستخدام أدوات R القياسية وحفظها في PNG مؤقت المخزنة في ذاكرة الوصول العشوائي (على لينكس ، توجد أدلة R المؤقتة في دليل /tmp المثبتة في RAM). ثم يتم قراءة هذا الملف في شكل صفيف ثلاثي الأبعاد مع وجود أرقام في النطاق من 0 إلى 1. هذا مهم لأنه سيتم قراءة BMP الأكثر شيوعًا في صفيف خام به رموز ألوان سداسية عشرية.
اختبر النتيجة:
zip_file <- file.path("data", "train_simplified.zip") csv_file <- "cat.csv" unzip(zip_file, files = csv_file, exdir = tempdir(), junkpaths = TRUE, unzip = getOption("unzip")) tmp_data <- data.table::fread(file.path(tempdir(), csv_file), sep = ",", select = "drawing", nrows = 10000) arr <- r_process_json_str(tmp_data[4, drawing]) dim(arr) # [1] 256 256 3 plot(magick::image_read(arr))

سيتم تشكيل الدفعة نفسها على النحو التالي:
res <- r_process_json_vector(tmp_data[1:4, drawing], scale = 0.5) str(res) # num [1:4, 1:128, 1:128, 1:3] 1 1 1 1 1 1 1 1 1 1 ... # - attr(*, "dimnames")=List of 4 # ..$ : NULL # ..$ : NULL # ..$ : NULL # ..$ : NULL
بدا لنا أن هذا التطبيق غير مثالي ، لأن تكوين مجموعات كبيرة يستغرق وقتًا غير لائق كثيرًا ، وقررنا استخدام تجربة زملائنا باستخدام مكتبة OpenCV القوية. في ذلك الوقت ، لم يكن هناك حزمة جاهزة لـ R (لا يوجد حتى الآن) ، لذلك تمت كتابة الحد الأدنى من تنفيذ الوظيفة المطلوبة في C ++ مع التكامل في كود R باستخدام Rcpp .
لحل المشكلة ، تم استخدام الحزم والمكتبات التالية:
- OpenCV للتصوير ورسم الخط. استخدمنا مكتبات النظام المثبتة مسبقًا وملفات الرأس ، بالإضافة إلى الارتباط الديناميكي.
- xtensor للعمل مع المصفوفات متعددة الأبعاد والتنسورات. استخدمنا ملفات الرأس المضمنة في حزمة R التي تحمل نفس الاسم. تتيح لك المكتبة العمل مع المصفوفات متعددة الأبعاد ، سواء في الصف الرئيسي أو ترتيب الأعمدة الرئيسية.
- ndjson لتحليل JSON. يتم استخدام هذه المكتبة في xtensor تلقائيًا عندما تكون متوفرة في المشروع.
- RcppThread لتنظيم معالجة متعددة الخيوط لمتجه من JSON. استخدم ملفات الرأس المقدمة من هذه الحزمة. تختلف الحزمة عن RcppParallel الأكثر شعبية من بين أمور أخرى من خلال آلية المقاطعة المضمنة.
تجدر الإشارة إلى أن xtensor اتضح أنها مجرد اكتشاف: بالإضافة إلى وجود وظائف واسعة وأداء عالٍ ، تحول مطوروها إلى استجابة سريعة وفورية وأجابوا بالتفصيل على الأسئلة التي نشأت. من خلال مساعدتهم ، كان من الممكن تنفيذ تحويل مصفوفات OpenCV إلى التنسورات xtensor ، بالإضافة إلى طريقة لدمج التنسورات ثلاثية الأبعاد للصورة في موتر ثلاثي الأبعاد للبعد الصحيح (في الواقع الدُفعة).
دراسة المواد ل Rcpp ، xtensor ، و RcppThread لتجميع الملفات باستخدام ملفات النظام والربط الديناميكي مع المكتبات المثبتة في النظام ، استخدمنا آلية المكونات الإضافية المطبقة في حزمة Rcpp . للعثور على المسارات والأعلام تلقائيًا ، استخدمنا أداة التشغيل الشهيرة pkg-config linux.
تطبيق البرنامج المساعد Rcpp لاستخدام مكتبة OpenCV Rcpp::registerPlugin("opencv", function() { # pkg_config_name <- c("opencv", "opencv4") # pkg-config pkg_config_bin <- Sys.which("pkg-config") # checkmate::assert_file_exists(pkg_config_bin, access = "x") # OpenCV pkg-config check <- sapply(pkg_config_name, function(pkg) system(paste(pkg_config_bin, pkg))) if (all(check != 0)) { stop("OpenCV config for the pkg-config not found", call. = FALSE) } pkg_config_name <- pkg_config_name[check == 0] list(env = list( PKG_CXXFLAGS = system(paste(pkg_config_bin, "--cflags", pkg_config_name), intern = TRUE), PKG_LIBS = system(paste(pkg_config_bin, "--libs", pkg_config_name), intern = TRUE) )) })
نتيجة للمكون الإضافي ، أثناء التحويل البرمجي ، سيتم استبدال القيم التالية:
Rcpp:::.plugins$opencv()$env # $PKG_CXXFLAGS # [1] "-I/usr/include/opencv" # # $PKG_LIBS # [1] "-lopencv_shape -lopencv_stitching -lopencv_superres -lopencv_videostab -lopencv_aruco -lopencv_bgsegm -lopencv_bioinspired -lopencv_ccalib -lopencv_datasets -lopencv_dpm -lopencv_face -lopencv_freetype -lopencv_fuzzy -lopencv_hdf -lopencv_line_descriptor -lopencv_optflow -lopencv_video -lopencv_plot -lopencv_reg -lopencv_saliency -lopencv_stereo -lopencv_structured_light -lopencv_phase_unwrapping -lopencv_rgbd -lopencv_viz -lopencv_surface_matching -lopencv_text -lopencv_ximgproc -lopencv_calib3d -lopencv_features2d -lopencv_flann -lopencv_xobjdetect -lopencv_objdetect -lopencv_ml -lopencv_xphoto -lopencv_highgui -lopencv_videoio -lopencv_imgcodecs -lopencv_photo -lopencv_imgproc -lopencv_core"
يتم إعطاء رمز تنفيذ تحليل JSON وإنشاء مجموعة لنقله إلى النموذج أسفل المفسد. أولاً ، أضف دليل المشروع المحلي للبحث عن ملفات الرأس (اللازمة لـ ndjson):
Sys.setenv("PKG_CXXFLAGS" = paste0("-I", normalizePath(file.path("src"))))
تطبيق JSON لتحويل الموتر في C ++ // [[Rcpp::plugins(cpp14)]] // [[Rcpp::plugins(opencv)]] // [[Rcpp::depends(xtensor)]] // [[Rcpp::depends(RcppThread)]] #include <xtensor/xjson.hpp> #include <xtensor/xadapt.hpp> #include <xtensor/xview.hpp> #include <xtensor-r/rtensor.hpp> #include <opencv2/core/core.hpp> #include <opencv2/highgui/highgui.hpp> #include <opencv2/imgproc/imgproc.hpp> #include <Rcpp.h> #include <RcppThread.h> // using RcppThread::parallelFor; using json = nlohmann::json; using points = xt::xtensor<double,2>; // JSON using strokes = std::vector<points>; // JSON using xtensor3d = xt::xtensor<double, 3>; // using xtensor4d = xt::xtensor<double, 4>; // using rtensor3d = xt::rtensor<double, 3>; // R using rtensor4d = xt::rtensor<double, 4>; // R // // const static int SIZE = 256; // // . https://en.wikipedia.org/wiki/Pixel_connectivity#2-dimensional const static int LINE_TYPE = cv::LINE_4; // const static int LINE_WIDTH = 3; // // https://docs.opencv.org/3.1.0/da/d54/group__imgproc__transform.html#ga5bb5a1fea74ea38e1a5445ca803ff121 const static int RESIZE_TYPE = cv::INTER_LINEAR; // OpenCV- template <typename T, int NCH, typename XT=xt::xtensor<T,3,xt::layout_type::column_major>> XT to_xt(const cv::Mat_<cv::Vec<T, NCH>>& src) { // std::vector<int> shape = {src.rows, src.cols, NCH}; // size_t size = src.total() * NCH; // cv::Mat xt::xtensor XT res = xt::adapt((T*) src.data, size, xt::no_ownership(), shape); return res; } // JSON strokes parse_json(const std::string& x) { auto j = json::parse(x); // if (!j.is_array()) { throw std::runtime_error("'x' must be JSON array."); } strokes res; res.reserve(j.size()); for (const auto& a: j) { // 2- if (!a.is_array() || a.size() != 2) { throw std::runtime_error("'x' must include only 2d arrays."); } // auto p = a.get<points>(); res.push_back(p); } return res; } // // HSV cv::Mat ocv_draw_lines(const strokes& x, bool color = true) { // auto stype = color ? CV_8UC3 : CV_8UC1; // auto dtype = color ? CV_32FC3 : CV_32FC1; auto bg = color ? cv::Scalar(0, 0, 255) : cv::Scalar(255); auto col = color ? cv::Scalar(0, 255, 220) : cv::Scalar(0); cv::Mat img = cv::Mat(SIZE, SIZE, stype, bg); // size_t n = x.size(); for (const auto& s: x) { // size_t n_points = s.shape()[1]; for (size_t i = 0; i < n_points - 1; ++i) { // cv::Point from(s(0, i), s(1, i)); // cv::Point to(s(0, i + 1), s(1, i + 1)); // cv::line(img, from, to, col, LINE_WIDTH, LINE_TYPE); } if (color) { // col[0] += 180 / n; } } if (color) { // RGB cv::cvtColor(img, img, cv::COLOR_HSV2RGB); } // float32 [0, 1] img.convertTo(img, dtype, 1 / 255.0); return img; } // JSON xtensor3d process(const std::string& x, double scale = 1.0, bool color = true) { auto p = parse_json(x); auto img = ocv_draw_lines(p, color); if (scale != 1) { cv::Mat out; cv::resize(img, out, cv::Size(), scale, scale, RESIZE_TYPE); cv::swap(img, out); out.release(); } xtensor3d arr = color ? to_xt<double,3>(img) : to_xt<double,1>(img); return arr; } // [[Rcpp::export]] rtensor3d cpp_process_json_str(const std::string& x, double scale = 1.0, bool color = true) { xtensor3d res = process(x, scale, color); return res; } // [[Rcpp::export]] rtensor4d cpp_process_json_vector(const std::vector<std::string>& x, double scale = 1.0, bool color = false) { size_t n = x.size(); size_t dim = floor(SIZE * scale); size_t channels = color ? 3 : 1; xtensor4d res({n, dim, dim, channels}); parallelFor(0, n, [&x, &res, scale, color](int i) { xtensor3d tmp = process(x[i], scale, color); auto view = xt::view(res, i, xt::all(), xt::all(), xt::all()); view = tmp; }); return res; }
يجب وضع هذا الرمز في src/cv_xt.cpp باستخدام الأمر Rcpp::sourceCpp(file = "src/cv_xt.cpp", env = .GlobalEnv) ؛ ستحتاج أيضًا إلى nlohmann/json.hpp من المستودع إلى العمل . ينقسم الرمز إلى عدة وظائف:
to_xt - دالة قالب لتحويل مصفوفة الصورة ( cv::Mat ) إلى tensor xt::xtensor ؛parse_json - تقوم الدالة بتوزيع سلسلة JSON واستخراج إحداثيات النقاط parse_json في ناقل.ocv_draw_lines - ocv_draw_lines خطوطًا متعددة الألوان من متجه النقاط المستلم ؛process - يجمع بين الوظائف المذكورة أعلاه ، ويضيف أيضًا القدرة على تغيير حجم الصورة الناتجة ؛cpp_process_json_str - غلاف على دالة process ، والذي يصدر النتيجة إلى كائن R (مجموعة متعددة الأبعاد) ؛cpp_process_json_vector - برنامج التفاف على دالة cpp_process_json_str ، والذي يسمح لك بمعالجة متجه السلسلة في الوضع متعدد مؤشرات الترابط.
لرسم خطوط متعددة الألوان ، تم استخدام نموذج ألوان HSV ، ثم التحويل إلى RGB. اختبر النتيجة:
arr <- cpp_process_json_str(tmp_data[4, drawing]) dim(arr) # [1] 256 256 3 plot(magick::image_read(arr))

مقارنة سرعة التطبيقات في R و C ++ res_bench <- bench::mark( r_process_json_str(tmp_data[4, drawing], scale = 0.5), cpp_process_json_str(tmp_data[4, drawing], scale = 0.5), check = FALSE, min_iterations = 100 ) # cols <- c("expression", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # expression min median max `itr/sec` total_time n_itr # <chr> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 r_process_json_str 3.49ms 3.55ms 4.47ms 273. 490ms 134 # 2 cpp_process_json_str 1.94ms 2.02ms 5.32ms 489. 497ms 243 library(ggplot2) # res_bench <- bench::press( batch_size = 2^(4:10), { .data <- tmp_data[sample(seq_len(.N), batch_size), drawing] bench::mark( r_process_json_vector(.data, scale = 0.5), cpp_process_json_vector(.data, scale = 0.5), min_iterations = 50, check = FALSE ) } ) res_bench[, cols] # expression batch_size min median max `itr/sec` total_time n_itr # <chr> <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 r 16 50.61ms 53.34ms 54.82ms 19.1 471.13ms 9 # 2 cpp 16 4.46ms 5.39ms 7.78ms 192. 474.09ms 91 # 3 r 32 105.7ms 109.74ms 212.26ms 7.69 6.5s 50 # 4 cpp 32 7.76ms 10.97ms 15.23ms 95.6 522.78ms 50 # 5 r 64 211.41ms 226.18ms 332.65ms 3.85 12.99s 50 # 6 cpp 64 25.09ms 27.34ms 32.04ms 36.0 1.39s 50 # 7 r 128 534.5ms 627.92ms 659.08ms 1.61 31.03s 50 # 8 cpp 128 56.37ms 58.46ms 66.03ms 16.9 2.95s 50 # 9 r 256 1.15s 1.18s 1.29s 0.851 58.78s 50 # 10 cpp 256 114.97ms 117.39ms 130.09ms 8.45 5.92s 50 # 11 r 512 2.09s 2.15s 2.32s 0.463 1.8m 50 # 12 cpp 512 230.81ms 235.6ms 261.99ms 4.18 11.97s 50 # 13 r 1024 4s 4.22s 4.4s 0.238 3.5m 50 # 14 cpp 1024 410.48ms 431.43ms 462.44ms 2.33 21.45s 50 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = expression, color = expression)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() + scale_color_discrete(name = "", labels = c("cpp", "r")) + theme(legend.position = "bottom")
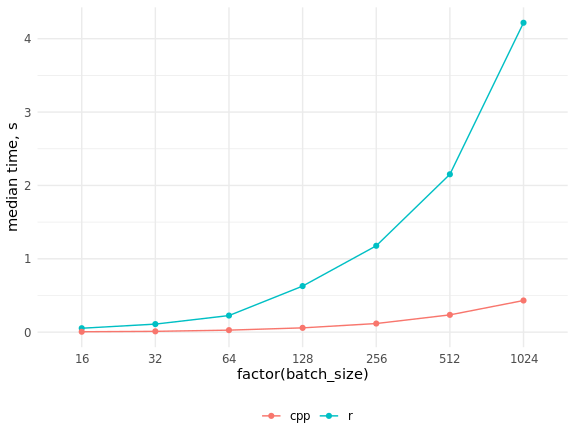
كما ترون ، اتضح أن الزيادة في السرعة كانت مهمة للغاية ، ولا يمكن اللحاق برمز C ++ من خلال موازاة رمز R.
3. التكرار لتفريغ الدفعات من قاعدة البيانات
تتمتع R بسمعة مستحقة باعتبارها لغة لمعالجة البيانات الموجودة في ذاكرة الوصول العشوائي ، بينما يتميز Python بمعالجة البيانات التكرارية ، مما يجعل من السهل والسهل تنفيذ العمليات الحسابية الأساسية (العمليات الحسابية باستخدام الذاكرة الخارجية). الكلاسيكية وذات الصلة بالنسبة لنا في سياق المشكلة الموصوفة ، مثال على هذه الحسابات هي الشبكات العصبية العميقة ، المدربة على طريقة نزول التدرج مع تقريب التدرج في كل خطوة بواسطة جزء صغير من الملاحظات ، أو دفعة صغيرة.
تحتوي أطر التعلّم العميق المكتوبة في Python على فصول خاصة تقوم بتنفيذ التكرارات استنادًا إلى البيانات: الجداول ، والصور في المجلدات ، والتنسيقات الثنائية ، وما إلى ذلك. في R ، يمكننا الاستفادة الكاملة من مكتبة Keras Python مع مختلف جوانبها الخلفية باستخدام الحزمة التي تحمل الاسم نفسه ، والتي تعمل بدورها أعلى حزمة شبكية . هذا الأخير يستحق مادة كبيرة منفصلة. لا يسمح لك فقط بتشغيل شفرة Python من R ، بل يوفر أيضًا نقل الكائنات بين جلسات R- و Python ، مع إجراء جميع تحويلات الأنواع الضرورية تلقائيًا.
لقد تخلصنا من الحاجة إلى تخزين جميع البيانات في ذاكرة الوصول العشوائي بسبب استخدام MonetDBLite ، وسيتم تنفيذ جميع أعمال "الشبكة العصبية" بواسطة رمز بيثون الأصلي ، وعلينا فقط كتابة مكرر بناءً على البيانات ، لأنه لا يوجد استعداد لمثل هذا الموقف في كل من R أو Python. : ( R ). R numpy-, keras .
:
train_generator <- function(db_connection = con, samples_index, num_classes = 340, batch_size = 32, scale = 1, color = FALSE, imagenet_preproc = FALSE) { # checkmate::assert_class(con, "DBIConnection") checkmate::assert_integerish(samples_index) checkmate::assert_count(num_classes) checkmate::assert_count(batch_size) checkmate::assert_number(scale, lower = 0.001, upper = 5) checkmate::assert_flag(color) checkmate::assert_flag(imagenet_preproc) # , dt <- data.table::data.table(id = sample(samples_index)) # dt[, batch := (.I - 1L) %/% batch_size + 1L] # dt <- dt[, if (.N == batch_size) .SD, keyby = batch] # i <- 1 # max_i <- dt[, max(batch)] # sql <- sprintf( "PREPARE SELECT drawing, label_int FROM doodles WHERE id IN (%s)", paste(rep("?", batch_size), collapse = ",") ) res <- DBI::dbSendQuery(con, sql) # keras::to_categorical to_categorical <- function(x, num) { n <- length(x) m <- numeric(n * num) m[x * n + seq_len(n)] <- 1 dim(m) <- c(n, num) return(m) } # function() { # if (i > max_i) { dt[, id := sample(id)] data.table::setkey(dt, batch) # i <<- 1 max_i <<- dt[, max(batch)] } # ID batch_ind <- dt[batch == i, id] # batch <- DBI::dbFetch(DBI::dbBind(res, as.list(batch_ind)), n = -1) # i <<- i + 1 # JSON batch_x <- cpp_process_json_vector(batch$drawing, scale = scale, color = color) if (imagenet_preproc) { # c [0, 1] [-1, 1] batch_x <- (batch_x - 0.5) * 2 } batch_y <- to_categorical(batch$label_int, num_classes) result <- list(batch_x, batch_y) return(result) } }
, , , , ( scale = 1 256256 , scale = 0.5 — 128128 ), ( color = FALSE , color = TRUE ) , imagenet-. , [0, 1] [-1, 1], keras .
, data.table samples_index , , SQL- . keras::to_categorical() . , , steps_per_epoch keras::fit_generator() , if (i > max_i) .
, , JSON- ( cpp_process_json_vector() , C++) , . one-hot , , . data.table — "" data.table - R.
Core i5 :
library(Rcpp) library(keras) library(ggplot2) source("utils/rcpp.R") source("utils/keras_iterator.R") con <- DBI::dbConnect(drv = MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR")) ind <- seq_len(DBI::dbGetQuery(con, "SELECT count(*) FROM doodles")[[1L]]) num_classes <- DBI::dbGetQuery(con, "SELECT max(label_int) + 1 FROM doodles")[[1L]] # train_ind <- sample(ind, floor(length(ind) * 0.995)) # val_ind <- ind[-train_ind] rm(ind) # scale <- 0.5 # res_bench <- bench::press( batch_size = 2^(4:10), { it1 <- train_generator( db_connection = con, samples_index = train_ind, num_classes = num_classes, batch_size = batch_size, scale = scale ) bench::mark( it1(), min_iterations = 50L ) } ) # cols <- c("batch_size", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # batch_size min median max `itr/sec` total_time n_itr # <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 16 25ms 64.36ms 92.2ms 15.9 3.09s 49 # 2 32 48.4ms 118.13ms 197.24ms 8.17 5.88s 48 # 3 64 69.3ms 117.93ms 181.14ms 8.57 5.83s 50 # 4 128 157.2ms 240.74ms 503.87ms 3.85 12.71s 49 # 5 256 359.3ms 613.52ms 988.73ms 1.54 30.5s 47 # 6 512 884.7ms 1.53s 2.07s 0.674 1.11m 45 # 7 1024 2.7s 3.83s 5.47s 0.261 2.81m 44 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = 1)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() DBI::dbDisconnect(con, shutdown = TRUE)

, ( 32 ). /dev/shm , . , /etc/fstab , tmpfs /dev/shm tmpfs defaults,size=25g 0 0 . , df -h .
, :
test_generator <- function(dt, batch_size = 32, scale = 1, color = FALSE, imagenet_preproc = FALSE) { # checkmate::assert_data_table(dt) checkmate::assert_count(batch_size) checkmate::assert_number(scale, lower = 0.001, upper = 5) checkmate::assert_flag(color) checkmate::assert_flag(imagenet_preproc) # dt[, batch := (.I - 1L) %/% batch_size + 1L] data.table::setkey(dt, batch) i <- 1 max_i <- dt[, max(batch)] # function() { batch_x <- cpp_process_json_vector(dt[batch == i, drawing], scale = scale, color = color) if (imagenet_preproc) { # c [0, 1] [-1, 1] batch_x <- (batch_x - 0.5) * 2 } result <- list(batch_x) i <<- i + 1 return(result) } }
4.
mobilenet v1 , . keras , , R. : (batch, height, width, 3) , . Python , , ( , keras- ):
mobilenet v1 library(keras) top_3_categorical_accuracy <- custom_metric( name = "top_3_categorical_accuracy", metric_fn = function(y_true, y_pred) { metric_top_k_categorical_accuracy(y_true, y_pred, k = 3) } ) layer_sep_conv_bn <- function(object, filters, alpha = 1, depth_multiplier = 1, strides = c(2, 2)) { # NB! depth_multiplier != resolution multiplier # https://github.com/keras-team/keras/issues/10349 layer_depthwise_conv_2d( object = object, kernel_size = c(3, 3), strides = strides, padding = "same", depth_multiplier = depth_multiplier ) %>% layer_batch_normalization() %>% layer_activation_relu() %>% layer_conv_2d( filters = filters * alpha, kernel_size = c(1, 1), strides = c(1, 1) ) %>% layer_batch_normalization() %>% layer_activation_relu() } get_mobilenet_v1 <- function(input_shape = c(224, 224, 1), num_classes = 340, alpha = 1, depth_multiplier = 1, optimizer = optimizer_adam(lr = 0.002), loss = "categorical_crossentropy", metrics = c("categorical_crossentropy", top_3_categorical_accuracy)) { inputs <- layer_input(shape = input_shape) outputs <- inputs %>% layer_conv_2d(filters = 32, kernel_size = c(3, 3), strides = c(2, 2), padding = "same") %>% layer_batch_normalization() %>% layer_activation_relu() %>% layer_sep_conv_bn(filters = 64, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 128, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 128, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 256, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 256, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 1024, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 1024, strides = c(1, 1)) %>% layer_global_average_pooling_2d() %>% layer_dense(units = num_classes) %>% layer_activation_softmax() model <- keras_model( inputs = inputs, outputs = outputs ) model %>% compile( optimizer = optimizer, loss = loss, metrics = metrics ) return(model) }
. , , , . , imagenet-. , . get_config() ( base_model_conf$layers — R- ), from_config() :
base_model_conf <- get_config(base_model) base_model_conf$layers[[1]]$config$batch_input_shape[[4]] <- 1L base_model <- from_config(base_model_conf)
keras imagenet- :
get_model <- function(name = "mobilenet_v2", input_shape = NULL, weights = "imagenet", pooling = "avg", num_classes = NULL, optimizer = keras::optimizer_adam(lr = 0.002), loss = "categorical_crossentropy", metrics = NULL, color = TRUE, compile = FALSE) { # checkmate::assert_string(name) checkmate::assert_integerish(input_shape, lower = 1, upper = 256, len = 3) checkmate::assert_count(num_classes) checkmate::assert_flag(color) checkmate::assert_flag(compile) # keras model_fun <- get0(paste0("application_", name), envir = asNamespace("keras")) # if (is.null(model_fun)) { stop("Model ", shQuote(name), " not found.", call. = FALSE) } base_model <- model_fun( input_shape = input_shape, include_top = FALSE, weights = weights, pooling = pooling ) # , if (!color) { base_model_conf <- keras::get_config(base_model) base_model_conf$layers[[1]]$config$batch_input_shape[[4]] <- 1L base_model <- keras::from_config(base_model_conf) } predictions <- keras::get_layer(base_model, "global_average_pooling2d_1")$output predictions <- keras::layer_dense(predictions, units = num_classes, activation = "softmax") model <- keras::keras_model( inputs = base_model$input, outputs = predictions ) if (compile) { keras::compile( object = model, optimizer = optimizer, loss = loss, metrics = metrics ) } return(model) }
. : get_weights() R- , ( - ), set_weights() . , , .
mobilenet 1 2, resnet34. , SE-ResNeXt. , , ( ).
5.
, docopt :
doc <- ' Usage: train_nn.R --help train_nn.R --list-models train_nn.R [options] Options: -h --help Show this message. -l --list-models List available models. -m --model=<model> Neural network model name [default: mobilenet_v2]. -b --batch-size=<size> Batch size [default: 32]. -s --scale-factor=<ratio> Scale factor [default: 0.5]. -c --color Use color lines [default: FALSE]. -d --db-dir=<path> Path to database directory [default: Sys.getenv("db_dir")]. -r --validate-ratio=<ratio> Validate sample ratio [default: 0.995]. -n --n-gpu=<number> Number of GPUs [default: 1]. ' args <- docopt::docopt(doc)
docopt http://docopt.org/ R. Rscript bin/train_nn.R -m resnet50 -c -d /home/andrey/doodle_db ./bin/train_nn.R -m resnet50 -c -d /home/andrey/doodle_db , train_nn.R ( resnet50 128128 , /home/andrey/doodle_db ). , . , mobilenet_v2 keras R - R- — , .
RStudio ( tfruns ). , RStudio.
6.
. R- .
« », . , NVIDIA, CUDA+cuDNN — , tensorflow/tensorflow:1.12.0-gpu , R-.
- :
Dockerfile FROM tensorflow/tensorflow:1.12.0-gpu MAINTAINER Artem Klevtsov <aaklevtsov@gmail.com> SHELL ["/bin/bash", "-c"] ARG LOCALE="en_US.UTF-8" ARG APT_PKG="libopencv-dev r-base r-base-dev littler" ARG R_BIN_PKG="futile.logger checkmate data.table rcpp rapidjsonr dbi keras jsonlite curl digest remotes" ARG R_SRC_PKG="xtensor RcppThread docopt MonetDBLite" ARG PY_PIP_PKG="keras" ARG DIRS="/db /app /app/data /app/models /app/logs" RUN source /etc/os-release && \ echo "deb https://cloud.r-project.org/bin/linux/ubuntu ${UBUNTU_CODENAME}-cran35/" > /etc/apt/sources.list.d/cran35.list && \ apt-key adv --keyserver keyserver.ubuntu.com --recv-keys E084DAB9 && \ add-apt-repository -y ppa:marutter/c2d4u3.5 && \ add-apt-repository -y ppa:timsc/opencv-3.4 && \ apt-get update && \ apt-get install -y locales && \ locale-gen ${LOCALE} && \ apt-get install -y --no-install-recommends ${APT_PKG} && \ ln -s /usr/lib/R/site-library/littler/examples/install.r /usr/local/bin/install.r && \ ln -s /usr/lib/R/site-library/littler/examples/install2.r /usr/local/bin/install2.r && \ ln -s /usr/lib/R/site-library/littler/examples/installGithub.r /usr/local/bin/installGithub.r && \ echo 'options(Ncpus = parallel::detectCores())' >> /etc/R/Rprofile.site && \ echo 'options(repos = c(CRAN = "https://cloud.r-project.org"))' >> /etc/R/Rprofile.site && \ apt-get install -y $(printf "r-cran-%s " ${R_BIN_PKG}) && \ install.r ${R_SRC_PKG} && \ pip install ${PY_PIP_PKG} && \ mkdir -p ${DIRS} && \ chmod 777 ${DIRS} && \ rm -rf /tmp/downloaded_packages/ /tmp/*.rds && \ rm -rf /var/lib/apt/lists/* COPY utils /app/utils COPY src /app/src COPY tests /app/tests COPY bin/*.R /app/ ENV DBDIR="/db" ENV CUDA_HOME="/usr/local/cuda" ENV PATH="/app:${PATH}" WORKDIR /app VOLUME /db VOLUME /app CMD bash
; . /bin/bash /etc/os-release . .
-, . , , , :
#!/bin/sh DBDIR=${PWD}/db LOGSDIR=${PWD}/logs MODELDIR=${PWD}/models DATADIR=${PWD}/data ARGS="--runtime=nvidia --rm -v ${DBDIR}:/db -v ${LOGSDIR}:/app/logs -v ${MODELDIR}:/app/models -v ${DATADIR}:/app/data" if [ -z "$1" ]; then CMD="Rscript /app/train_nn.R" elif [ "$1" = "bash" ]; then ARGS="${ARGS} -ti" else CMD="Rscript /app/train_nn.R $@" fi docker run ${ARGS} doodles-tf ${CMD}
- , train_nn.R ; — "bash", . : CMD="Rscript /app/train_nn.R $@" .
, , , .
7. GPU Google Cloud
(. , @Leigh.plt ODS-). , 1 GPU GPU . GoogleCloud ( ) - , $300. 4V100 SSD , . , . K80. — SSD c, dev/shm .
, GPU. CPU , :
with(tensorflow::tf$device("/cpu:0"), { model_cpu <- get_model( name = model_name, input_shape = input_shape, weights = weights, metrics =(top_3_categorical_accuracy, compile = FALSE ) })
( ) GPU, :
model <- keras::multi_gpu_model(model_cpu, gpus = n_gpu) keras::compile( object = model, optimizer = keras::optimizer_adam(lr = 0.0004), loss = "categorical_crossentropy", metrics = c(top_3_categorical_accuracy) )
, , , GPU .
tensorboard , :
# log_file_tmpl <- file.path("logs", sprintf( "%s_%d_%dch_%s.csv", model_name, dim_size, channels, format(Sys.time(), "%Y%m%d%H%M%OS") )) # model_file_tmpl <- file.path("models", sprintf( "%s_%d_%dch_{epoch:02d}_{val_loss:.2f}.h5", model_name, dim_size, channels )) callbacks_list <- list( keras::callback_csv_logger( filename = log_file_tmpl ), keras::callback_early_stopping( monitor = "val_loss", min_delta = 1e-4, patience = 8, verbose = 1, mode = "min" ), keras::callback_reduce_lr_on_plateau( monitor = "val_loss", factor = 0.5, # lr 2 patience = 4, verbose = 1, min_delta = 1e-4, mode = "min" ), keras::callback_model_checkpoint( filepath = model_file_tmpl, monitor = "val_loss", save_best_only = FALSE, save_weights_only = FALSE, mode = "min" ) )
8.
, , :
- keras (
lr_finder fast.ai ); , R , , ; - , GPU;
- , imagenet-;
- one cycle policy discriminative learning rates (osine annealing , skeydan ).
:
- ( ) . data.table in-place , , . .
- R C++ Rcpp . RcppThread RcppParallel , , R .
- Rcpp C++, . xtensor CRAN, , R C++. — ++ RStudio.
- docopt . , .. . RStudio , IDE .
- , . .
- Google Cloud — , .
- , R C++, bench — .
, .