كيف أقرأ هذا المقال : أعتذر عن حقيقة أن النص انتهى لفترة طويلة وفوضوي. لتوفير الوقت ، أبدأ كل فصل بمقدمة "ما تعلمته" ، والذي أشرح فيه جوهر الفصل في جملة أو جملتين.
"فقط أظهر الحل!" إذا كنت ترغب فقط في رؤية ما جئت إليه ، فانتقل إلى الفصل "كن أكثر إبداعًا" ، لكنني أعتقد أنه من الممتع والمفيد قراءة المزيد عن حالات الفشل.في الآونة الأخيرة ، تلقيت تعليمات بإعداد عملية لمعالجة حجم كبير من تسلسل الحمض النووي الأصلي (من الناحية الفنية ، هذه عبارة عن شريحة SNP). كان من الضروري الحصول بسرعة على البيانات حول موقع وراثي معين (يسمى SNP) للنمذجة اللاحقة وغيرها من المهام. بمساعدة R و AWK ، تمكنت من تنظيف البيانات وتنظيمها بطريقة طبيعية ، مما أدى إلى زيادة كبيرة في معالجة الطلبات. هذا لم يكن سهلا بالنسبة لي وتطلب العديد من التكرار. هذه المقالة سوف تساعدك على تجنب بعض أخطائي وإظهار ما فعلت في النهاية.
أولاً ، بعض التفسيرات التمهيدية.
معطيات
زودنا مركز معالجة المعلومات الوراثية بالجامعة لدينا ببيانات تبلغ 25 تيرابايت من TSV. حصلت عليها مقسمة إلى 5 حزم مضغوطة بواسطة Gzip ، كل منها يحتوي على حوالي 240 ملف بأربعة غيغابايت. كل صف يحتوي على بيانات عن SNP واحد لشخص واحد. في المجموع ، تم نقل البيانات حول ~ 2.5 مليون SNPs و ~ 60 ألف شخص. بالإضافة إلى معلومات SNP ، كان هناك العديد من الأعمدة في الملفات مع أرقام تعكس الخصائص المختلفة ، مثل شدة القراءة ، وتواتر الأليلات المختلفة ، إلخ. كان هناك حوالي 30 عمودًا بقيم فريدة.
هدف
كما هو الحال مع أي مشروع لإدارة البيانات ، كان الشيء الأكثر أهمية هو تحديد كيفية استخدام البيانات. في هذه الحالة ،
بالنسبة للجزء الأكبر ، سنختار النماذج وسير العمل ل SNP على أساس SNP . وهذا هو ، في الوقت نفسه سنحتاج إلى بيانات SNP واحد فقط. كان عليّ أن أتعلم كيف استخرج كل السجلات المتعلقة بأحد 2.5 مليون من SNPs بكل بساطة قدر الإمكان وأسرع وأرخص.
كيف لا تفعل ذلك
سوف أقتبس كليشيه مناسبة:
لم أفشل ألف مرة ، لقد اكتشفت ألف طريقة لعدم تحليل مجموعة من البيانات بتنسيق مناسب للاستعلامات.
المحاولة الأولى
ما تعلمته : لا توجد وسيلة رخيصة لتحليل 25 تيرابايت في وقت واحد.
بعد الاستماع إلى موضوع "أساليب معالجة البيانات الضخمة المتقدمة" في جامعة فاندربيلت ، كنت متأكدًا من أنها كانت قبعة. ربما يتطلب الأمر ساعة أو ساعتين لتكوين خادم Hive لتشغيل جميع البيانات والإبلاغ عن النتيجة. نظرًا لأنه يتم تخزين بياناتنا في AWS S3 ، فقد استخدمت خدمة
Athena ، التي تتيح لك تطبيق استعلامات Hive SQL على بيانات S3. لا حاجة لتكوين / رفع Hive-cluster ، وحتى دفع فقط للبيانات التي تبحث عنها.
بعد أن عرضت أثينا على بياناتي وتنسيقه ، أجريت بعض الاختبارات مع استفسارات مماثلة:
select * from intensityData limit 10;
وحصلت بسرعة على نتائج جيدة التنظيم. القيام به.
حتى حاولنا استخدام البيانات في العمل ...
لقد طُلب مني سحب جميع معلومات SNP من أجل اختبار النموذج الموجود عليها. قمت بتشغيل استعلام:
select * from intensityData where snp = 'rs123456';
... وانتظر. بعد ثماني دقائق وأكثر من 4 تيرابايت من البيانات المطلوبة ، حصلت على النتيجة. تفرض Athena رسومًا على كمية البيانات التي يتم العثور عليها ، بمبلغ 5 دولارات لكل تيرابايت. إذن كلف هذا الطلب الواحد 20 دولارًا وثماني دقائق من الانتظار. لتشغيل النموذج وفقًا لجميع البيانات ، كان من الضروري الانتظار لمدة 38 عامًا ودفع 50 مليون دولار ، ومن الواضح أن هذا لم يناسبنا.
كان من الضروري استخدام الباركيه ...
ما تعلمته : كن حذرًا في حجم ملفات الباركيه وتنظيمها.
في البداية حاولت تصحيح الموقف عن طريق تحويل جميع ملفات TSV إلى
ملفات الباركيه . إنها ملائمة للعمل مع مجموعات البيانات الكبيرة ، لأن المعلومات الموجودة بها يتم تخزينها في نموذج عمودي: يكمن كل عمود في مقطع الذاكرة / القرص الخاص به ، على عكس الملفات النصية التي تحتوي الأسطر على عناصر من كل عمود. وإذا كنت بحاجة إلى العثور على شيء ما ، فما عليك سوى قراءة العمود الضروري. بالإضافة إلى ذلك ، يتم تخزين مجموعة من القيم في كل ملف في عمود ، لذلك إذا لم تكن القيمة المطلوبة في نطاق العمود ، فلن يضيع Spark الوقت في فحص الملف بأكمله.
قمت بتشغيل مهمة
AWS Glue بسيطة لتحويل ملفات TSV الخاصة بنا إلى Parquet وأسقطت ملفات جديدة في Athena. استغرق الأمر حوالي 5 ساعات. ولكن عندما أطلقت الطلب ، استغرق الأمر حوالي الوقت نفسه وأقل قليلاً من المال لإكماله. والحقيقة هي أن سبارك ، في محاولة لتحسين المهمة ، ببساطة فك قطعة واحدة من TSV ووضعها في قطعة الباركيه الخاصة بها. ونظرًا لأن كل جزء كبير بما فيه الكفاية ويحتوي على السجلات الكاملة للعديد من الأشخاص ، تم تخزين جميع SNPs في كل ملف ، لذلك كان على Spark فتح جميع الملفات لاستخراج المعلومات اللازمة.
الغريب أن نوع الضغط الافتراضي (والموصى به) في الباركيه - snappy - ليس قابلاً للانقسام. لذلك ، تمسك كل منفّذ بمهمة تفريغ وتنزيل مجموعة البيانات الكاملة 3.5 جيجابايت.

نحن نفهم المشكلة
ما تعلمته : الفرز أمر صعب ، خاصة إذا تم توزيع البيانات.
بدا لي الآن أنني فهمت جوهر المشكلة. كل ما كان علي فعله هو فرز البيانات حسب عمود SNP ، وليس حسب الأشخاص. بعد ذلك ، سيتم تخزين عدة SNPs في مقطع منفصل للبيانات ، ومن ثم ستظهر وظيفة Parquet الذكية "مفتوحة فقط إذا كانت القيمة في النطاق" في كل مجدها. لسوء الحظ ، فإن فرز مليارات الصفوف المتناثرة عبر مجموعة قد أثبت أنه مهمة شاقة.
بالتأكيد لا ترغب AWS في إعادة الأموال بسبب "أنا طالب غائب عن التفكير". بعد أن بدأت الفرز على Amazon Glue ، عملت لمدة يومين وتحطمت.
ماذا عن التقسيم؟
ما تعلمته : يجب أن تكون الأقسام في Spark متوازنة.
ثم جاءت الفكرة لتقسيم البيانات على الكروموسومات. هناك 23 منهم (وعدد قليل آخر ، بالنظر إلى الحمض النووي للميتوكوندريا والمناطق غير المعينة).
سيتيح لك ذلك تقسيم البيانات إلى أجزاء أصغر. إذا قمت بإضافة سطر
partition_by = "chr" واحد فقط إلى وظيفة تصدير Spark في نص الغراء ، فيجب حينئذٍ تصنيف البيانات في مجموعات.
 يتكون الجينوم من شظايا عديدة تسمى الكروموسومات.
يتكون الجينوم من شظايا عديدة تسمى الكروموسومات.لسوء الحظ ، هذا لم ينجح. الكروموسومات لها أحجام مختلفة ، وبالتالي كمية مختلفة من المعلومات. هذا يعني أن المهام التي أرسلها سبارك للعمال لم تكن متوازنة وأداء ببطء ، لأن بعض العقد انتهت في وقت مبكر وكانت خاملة. ومع ذلك ، تم الانتهاء من المهام. ولكن عند طلب SNP واحد ، تسبب الخلل مرة أخرى في مشاكل. انخفضت تكلفة معالجة SNPs على الكروموسومات الأكبر (أي حيث نريد الحصول على البيانات) بنحو 10 مرات فقط. الكثير ، ولكن ليس بما فيه الكفاية.
وإذا كنت تنقسم إلى أقسام أصغر؟
ما تعلمته : لا تحاول أبدًا عمل 2.5 مليون قسم على الإطلاق.
قررت أن أمشي ونقسم كل SNP. هذا يضمن نفس حجم الأقسام.
كان سيئا فكرة . لقد استفدت من الغراء وأضفت
partition_by = 'snp' . بدأت المهمة وبدأت في الجري. بعد يوم واحد ، راجعت ورأيت أنه لم يتم كتابة أي شيء في S3 حتى الآن ، لذا فقد قتلت المهمة. يبدو أن الغراء كان يكتب ملفات وسيطة إلى مكان مخفي في S3 ، وكثير من الملفات ، ربما بضعة ملايين. ونتيجة لذلك ، تكلف خطأي أكثر من ألف دولار ولم يرضي مرشدي.
التقسيم + الفرز
ما تعلمته : الفرز لا يزال صعبًا ، وكذلك إعداد Spark.
كانت آخر محاولة للتقسيم هي أنني قسمت الكروموسومات ثم قمت بترتيب كل قسم. من الناحية النظرية ، فإن هذا من شأنه تسريع كل طلب ، لأن بيانات SNP المطلوبة يجب أن تكون ضمن العديد من قطع الباركيه ضمن نطاق معين. للأسف ، أثبت فرز البيانات حتى المقسمة أنه مهمة صعبة. كنتيجة لذلك ، انتقلت إلى EMR للحصول على مجموعة مخصصة واستخدمت ثماني حالات قوية (C5.4xl) و Sparklyr لإنشاء سير عمل أكثر مرونة ...
# Sparklyr snippet to partition by chr and sort w/in partition # Join the raw data with the snp bins raw_data group_by(chr) %>% arrange(Position) %>% Spark_write_Parquet( path = DUMP_LOC, mode = 'overwrite', partition_by = c('chr') )
... ومع ذلك ، فإن المهمة لم تكتمل بعد. قمت بضبطها بكل طريقة: قمت بزيادة تخصيص الذاكرة لكل من ينفذ الاستعلام ، واستخدمت العقد بكمية كبيرة من الذاكرة ، واستخدمت متغيرات البث ، ولكن في كل مرة اتضح أنها نصف التدابير ، وبدأ فناني الأداء بالتدريج ، حتى توقف كل شيء.
أنا أتلقى المزيد من الإبداع
ما تعلمته : في بعض الأحيان تتطلب البيانات الخاصة حلولًا خاصة.
كل SNP لها قيمة مركزية. هذا هو الرقم المقابل لعدد القواعد الموجودة على كروموسومها. هذه طريقة جيدة وطبيعية لتنظيم بياناتنا. في البداية كنت أرغب في التقسيم حسب منطقة كل كروموسوم. على سبيل المثال ، المواقف 1 - 2000 ، 2001 - 4000 ، إلخ. ولكن المشكلة هي أن تعدد الأشكال لا يتم توزيعه بشكل متساوٍ عبر الكروموسومات ، وهذا هو السبب في أن حجم المجموعات سوف يتغير بشكل كبير.
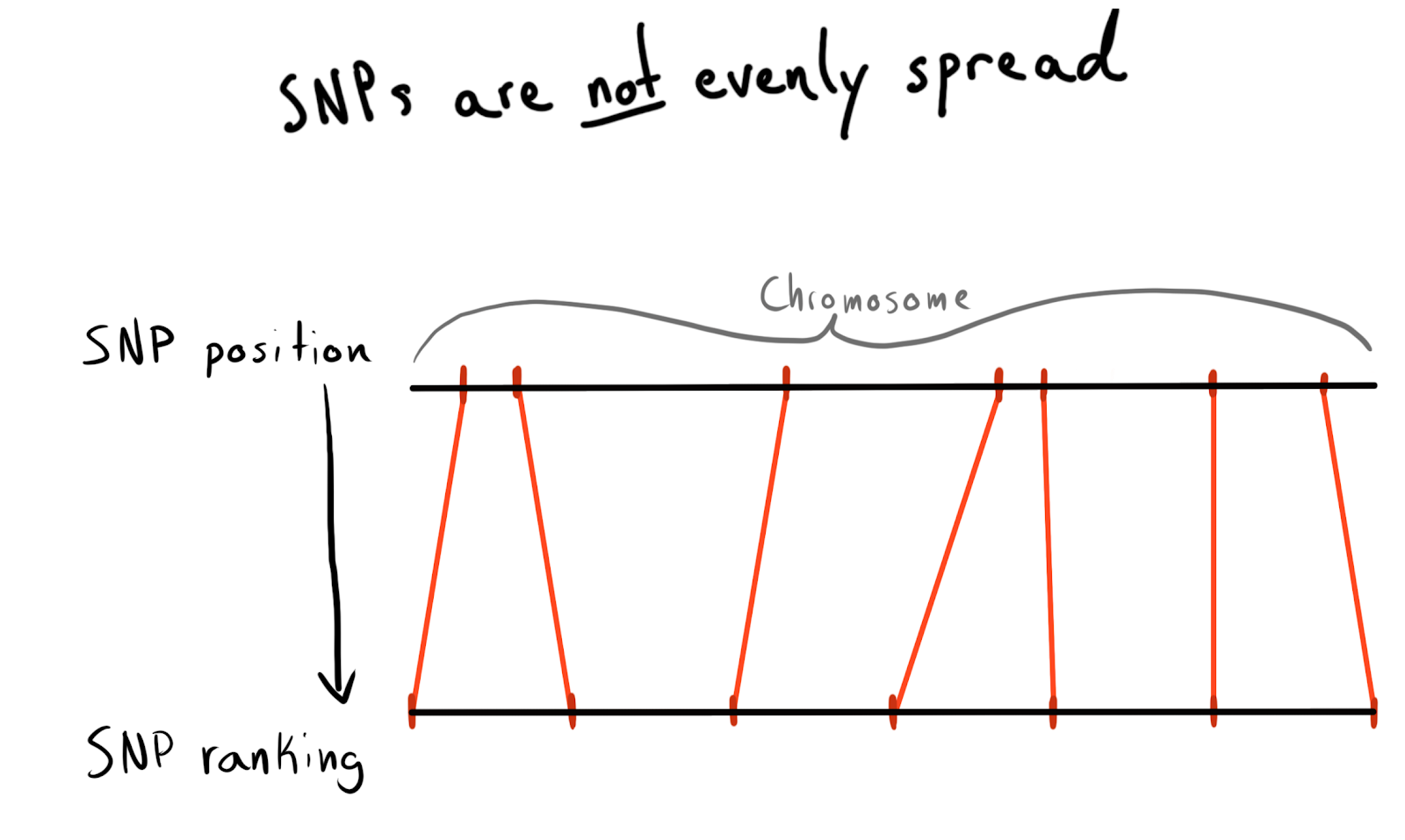
نتيجة لذلك ، أصبحت مقسمة إلى وظائف (رتبة). وفقًا للبيانات التي تم تنزيلها بالفعل ، قمت بتشغيل طلب للحصول على قائمة SNP فريدة ومواقعها وكروموسوماتها. ثم قام بفرز البيانات داخل كل كروموسوم وجمع SNP في مجموعات (بن) بحجم معين. قل 1000 SNP لكل منهما. هذا أعطاني علاقة SNP مع مجموعة في كروموسوم.
في النهاية ، قمت بإنشاء مجموعات (بن) على 75 SNP ، وسأشرح السبب أدناه.
snp_to_bin <- unique_snps %>% group_by(chr) %>% arrange(position) %>% mutate( rank = 1:n() bin = floor(rank/snps_per_bin) ) %>% ungroup()
المحاولة الأولى مع سبارك
ما تعلمته : تكامل Spark سريع ، لكن التقسيم لا يزال مكلفًا.
كنت أرغب في قراءة إطار البيانات الصغير (2.5 مليون خط) في Spark ، والجمع بينه وبين البيانات الخام ، ثم التقسيم حسب عمود الصندوق المُضاف حديثًا.
لقد استخدمت
sdf_broadcast() ، لذلك اكتشف Spark أنه يجب إرسال إطار بيانات إلى جميع العقد. هذا مفيد إذا كانت البيانات صغيرة ومطلوبة لجميع المهام. خلاف ذلك ، يحاول Spark أن يكون ذكيًا ويقوم بتوزيع البيانات حسب الحاجة ، مما قد يسبب الفرامل.
ومرة أخرى ، لم تنجح فكرتي: المهام التي عملت لفترة من الوقت ، أكملت الدمج ، وبعد ذلك ، مثل التنفيذيين الذين بدأوا بالتقسيم ، بدأوا في الفشل.
أضف AWK
ما تعلمته : لا تنام عندما تعلمك الأساسيات. بالتأكيد شخص ما حل مشكلتك بالفعل في الثمانينات.
حتى هذه النقطة ، كان سبب كل إخفاقاتي مع Spark هو تشويش البيانات في المجموعة. ربما يمكن تحسين الوضع عن طريق المعالجة المسبقة. قررت محاولة تقسيم بيانات النص الخام إلى أعمدة كروموسوم ، لذلك كنت آمل في تزويد Spark ببيانات "مُقسمة مسبقًا".
لقد بحثت في StackOverflow عن كيفية تحليل قيم الأعمدة ووجدت
إجابة رائعة. باستخدام AWK ، يمكنك تقسيم ملف نصي إلى قيم أعمدة بالكتابة إلى البرنامج النصي ، بدلاً من إرسال النتائج إلى
stdout .
للاختبار ، كتبت سيناريو Bash. لقد قمت بتنزيل أحد TSVs المحزوم ، ثم فككت الحزمة بواسطة
gzip وأرسلتها إلى
awk .
gzip -dc path/to/chunk/file.gz | awk -F '\t' \ '{print $1",..."$30">"chunked/"$chr"_chr"$15".csv"}'
لقد نجحت!
ملء الأساسية
ما تعلمته :
gnu parallel هو شيء سحري ، يجب على الجميع استخدامه.
كان الفصل بطيئًا إلى حد ما ، وعندما بدأت في
htop لاختبار استخدام مثيل EC2 قوي (وباهظ الثمن) ، اتضح أنني كنت أستخدم فقط نواة واحدة وحوالي 200 ميجابايت من الذاكرة. من أجل حل المشكلة وعدم خسارة الكثير من المال ، كان من الضروري معرفة كيفية موازاة العمل. لحسن الحظ ، في
علم البيانات المذهل لجيرون يانسنز
في كتاب
Command Line ، وجدت فصلاً عن التوازي. من ذلك ، تعلمت عن
gnu parallel ، وهي طريقة مرنة جدًا لتنفيذ multithreading على Unix.
عندما بدأت القسم باستخدام عملية جديدة ، كان كل شيء على ما يرام ، ولكن كان هناك عنق الزجاجة - لم يكن تنزيل كائنات S3 إلى القرص سريعًا جدًا ولم يكن متوازناً تمامًا. لإصلاح هذا ، قمت بهذا:
- اكتشفت أنه من الممكن تنفيذ خطوة التنزيل S3 مباشرة في خط الأنابيب ، مما يؤدي إلى التخلص التام من التخزين الوسيط على القرص. هذا يعني أنه يمكنني تجنب كتابة البيانات الخام على القرص واستخدام تخزين أصغر ، وبالتالي أرخص على AWS.
aws configure set default.s3.max_concurrent_requests 50 أوامر aws configure set default.s3.max_concurrent_requests 50 زادت بشكل كبير عدد مؤشرات الترابط التي يستخدمها AWS CLI (هناك 10 بشكل افتراضي).
- لقد تحولت إلى مثيل EC2 الأمثل لسرعة الشبكة ، مع حرف ن في الاسم. لقد وجدت أن فقد طاقة الحوسبة عند استخدام مثيلات n أكثر من تعويض بزيادة في سرعة التنزيل. بالنسبة لمعظم المهام ، كنت c5n.4xl.
- لقد غيرت
gzip إلى pigz ، فهذه أداة gzip يمكنها القيام بأشياء رائعة لموازنة المهمة التي لم يسبق لها مثيل المتمثلة في تفريغ الملفات (ساعد هذا على الأقل).
يتم دمج هذه الخطوات مع بعضها البعض بحيث يعمل كل شيء بسرعة كبيرة. بفضل سرعة التنزيل المتزايدة ورفض الكتابة على القرص ، يمكنني الآن معالجة حزمة سعة 5 تيرابايت في غضون ساعات قليلة.
كان من المفترض أن تذكر هذه التغريدة "TSV". للأسف.
باستخدام إعادة تحليل البيانات
ما تعلمته : يحب Spark البيانات غير المضغوطة ولا يحب الجمع بين الأقسام.
الآن كانت البيانات في S3 بتنسيق (تم قراءتها ومشاركتها) وشبه مرتبة ، ويمكنني العودة إلى Spark مرة أخرى. لقد انتظرتني مفاجأة: فشلت مرة أخرى في تحقيق المطلوب! كان من الصعب للغاية معرفة سبارك بالضبط كيف تم تقسيم البيانات. وحتى عندما فعلت هذا ، اتضح أن هناك الكثير من الأقسام (95 ألف) ، وعندما قمت بتقليل عددهم إلى حدود متماسكة مع التماسك ، فقد أدى ذلك إلى تدمير التقسيم. أنا متأكد من أنه يمكن إصلاح ذلك ، لكن في غضون يومين من البحث ، لم أتمكن من إيجاد حل. في النهاية ، أكملت جميع المهام في Spark ، على الرغم من أن الأمر استغرق بعض الوقت ، ولم تكن ملفات الباركيه المنقسمة صغيرة جدًا (~ 200 كيلوبايت). ومع ذلك ، كانت البيانات حيث كانت هناك حاجة إليها.
 صغيرة ومختلفة ، رائعة!
صغيرة ومختلفة ، رائعة!اختبار طلبات سبارك المحلية
ما تعلمته : لقد سبارك الكثير من النفقات العامة في حل المشاكل البسيطة.
عن طريق تنزيل البيانات بتنسيق ذكي ، تمكنت من اختبار السرعة. قمت بإعداد برنامج نصي على R لبدء تشغيل خادم Spark المحلي ، ثم قمت بتحميل إطار بيانات Spark من مستودع مجموعات Parquet المحدد (حاوية). حاولت تحميل جميع البيانات ، لكن لم أستطع دفع Sparklyr إلى التعرف على التقسيم.
sc <- Spark_connect(master = "local") desired_snp <- 'rs34771739' # Start a timer start_time <- Sys.time() # Load the desired bin into Spark intensity_data <- sc %>% Spark_read_Parquet( name = 'intensity_data', path = get_snp_location(desired_snp), memory = FALSE ) # Subset bin to snp and then collect to local test_subset <- intensity_data %>% filter(SNP_Name == desired_snp) %>% collect() print(Sys.time() - start_time)
استغرق التنفيذ 29.415 ثانية. أفضل بكثير ، لكن ليس جيدًا جدًا لاجراء اختبارات على أي شيء. بالإضافة إلى ذلك ، لم أستطع تسريع العمل باستخدام التخزين المؤقت ، لأنه عندما حاولت مؤقتًا تخزين إطار بيانات في الذاكرة ، تعطل Spark دائمًا ، حتى عندما قمت بتخصيص أكثر من 50 جيجابايت من الذاكرة لمجموعة بيانات وزنها أقل من 15.
العودة إلى AWK
ما تعلمته : المصفوفات الترابطية AWK فعالة للغاية.
فهمت أنه يمكنني تحقيق سرعة أعلى. تذكرت أنه في دليل
بروس بارنيت الممتاز AWK ، قرأت عن ميزة رائعة تسمى "
المصفوفات الترابطية ". في الواقع ، هذه أزواج ذات قيمة رئيسية ، والتي كانت تسمى لسبب مختلف في AWK ، وبالتالي أنا بطريقة ما لم أذكرها بشكل خاص.
ذكر رومان تشيبليكا أن مصطلح "المصفوفات الترابطية" أقدم بكثير من مصطلح "زوج القيمة الرئيسية". حتى إذا كنت
تبحث عن مفتاح القيمة في Google Ngram ، فلن ترى هذا المصطلح هناك ، ولكنك ستجد مصفوفات ترابطية! بالإضافة إلى ذلك ، غالبًا ما يرتبط الزوج ذو القيمة الرئيسية بقواعد البيانات ، لذلك فمن المنطقي مقارنة هذه المقارنة بالهاشماب. أدركت أنه يمكنني استخدام هذه المصفوفات الترابطية لتوصيل SNPs الخاصة بي بجدول الصناديق والبيانات الأولية دون استخدام Spark.
لهذا ، في البرنامج النصي AWK ، استخدمت كتلة
BEGIN . هذا جزء من التعليمات البرمجية التي يتم تنفيذها قبل نقل السطر الأول من البيانات إلى النص الرئيسي للبرنامج النصي.
join_data.awk BEGIN { FS=","; batch_num=substr(chunk,7,1); chunk_id=substr(chunk,15,2); while(getline < "snp_to_bin.csv") {bin[$1] = $2} } { print $0 > "chunked/chr_"chr"_bin_"bin[$1]"_"batch_num"_"chunk_id".csv" }
قام الأمر
while(getline...) بتحميل جميع الأسطر من مجموعة CSV (bin) ، واضبط العمود الأول (SNP name) كمفتاح لصفيف
bin والقيمة الثانية (group) كقيمة. بعد ذلك ، في الكتلة
{ } ، التي يتم تطبيقها على جميع أسطر الملف الرئيسي ، يتم إرسال كل سطر إلى ملف الإخراج ، والذي يحصل على اسم فريد اعتمادًا على مجموعته (bin):
..._bin_"bin[$1]"_...تتوافق
chunk_id و
chunk_id مع البيانات المقدمة بواسطة خط الأنابيب ، والتي تجنبت حالة السباق ، وكتب كل مؤشر ترابط تنفيذ تم تشغيله
parallel مع ملفه الفريد الخاص به.
نظرًا لأنني قمت بتوزيع جميع البيانات الأولية في مجلدات على الكروموسومات التي تركت بعد تجربتي السابقة مع AWK ، يمكنني الآن كتابة نص برمجي آخر للمعالجة على الكروموسوم في وقت واحد وإعطاء بيانات مقسمة أعمق إلى S3.
DESIRED_CHR='13'
يحتوي البرنامج على قسمين
parallel .
يقرأ القسم الأول البيانات من جميع الملفات التي تحتوي على معلومات عن الكروموسوم المرغوب فيه ، ثم يتم توزيع هذه البيانات عبر التدفقات التي تبعثر الملفات في المجموعات المقابلة (حاوية). لمنع حدوث حالات السباق عند كتابة تدفقات متعددة في ملف واحد ، تقوم AWK بنقل أسماء الملفات لكتابة البيانات إلى أماكن مختلفة ، على سبيل المثال ،
chr_10_bin_52_batch_2_aa.csv . نتيجة لذلك ، يتم إنشاء العديد من الملفات الصغيرة على القرص (لهذا فقد استخدمت وحدات تخزين تيرابايت EBS).
يمر خط الأنابيب من القسم
parallel الثاني عبر المجموعات (حاوية) ويجمع ملفاتهم الفردية في ملفات CSV مشتركة مع
cat ، ثم يرسلها للتصدير.
البث إلى R؟
ما تعلمته : يمكنك الوصول إلى
stdin و
stdout من برنامج نصي R ، وبالتالي استخدامه في خط الأنابيب.
في البرنامج النصي Bash ، قد تلاحظ هذا السطر:
...cat chunked/*_bin_{}_*.csv | ./upload_as_rds.R... ...cat chunked/*_bin_{}_*.csv | ./upload_as_rds.R... إنه يترجم كل ملفات المجموعة المتسلسلة (bin) إلى البرنامج النصي R أدناه.
{} هي تقنية
parallel خاصة تقوم بإدراج أي بيانات يتم إرسالها إليها في الدفق المحدد مباشرة في الأمر نفسه. يوفر خيار
{#} معرف مؤشر ترابط فريدًا ، ويمثل
{%} رقم فتحة المهمة (مكرر ، ولكن ليس أبدًا في نفس الوقت). يمكن الاطلاع على قائمة بجميع الخيارات في
الوثائق. #!/usr/bin/env Rscript library(readr) library(aws.s3) # Read first command line argument data_destination <- commandArgs(trailingOnly = TRUE)[1] data_cols <- list(SNP_Name = 'c', ...) s3saveRDS( read_csv( file("stdin"), col_names = names(data_cols), col_types = data_cols ), object = data_destination )
عند تمرير متغير
file("stdin") إلى
readr::read_csv ، يتم تحميل البيانات المترجمة إلى البرنامج النصي R في الإطار ، والذي يتم كتابته مباشرةً بعد ذلك إلى S3 كملف
.rds باستخدام
aws.s3 .
RDS يشبه إلى حد ما إصدار أصغر من النيابة العامة ، دون زخرفة زخرفة العمود.
بعد إكمال البرنامج النصي Bash ، تلقيت
.rds ملفات
.rds الموجودة في S3 ، مما سمح لي باستخدام أنواع الضغط المضمنة الفعالة.
على الرغم من استخدام الفرامل R ، كل شيء يعمل بسرعة كبيرة. ليس من المستغرب أن يتم تحسين الشظايا في R المسؤولة عن قراءة البيانات وكتابتها. بعد الاختبار على كروموسوم واحد متوسط الحجم ، تم الانتهاء من المهمة على مثيل C5n.4xl خلال ساعتين تقريبًا.
قيود S3
ما تعلمته : بفضل التطبيق الذكي للمسارات ، يمكن لـ S3 معالجة العديد من الملفات.
كنت قلقًا إذا كان بإمكان S3 التعامل مع الكثير من الملفات المنقولة إليها. يمكنني جعل أسماء الملفات ذات معنى ، ولكن كيف سيبحث S3 عنها؟
 S3 ,
S3 , / . FAQ- S3., S3 - . (bucket) , — .
Amazon, , «-----» . : get-, . , 20 . bin-. , , (, , ). .
?
: — .
: « ?» ( gzip CSV- 7 ) . , R Parquet ( Arrow) Spark. R, , , .
: , .
, .
EC2 , ( , Spark ). , , AWS- 10 .
R .
S3 , .
library(aws.s3) library(tidyverse) chr_sizes <- get_bucket_df( bucket = '...', prefix = '...', max = Inf ) %>% mutate(Size = as.numeric(Size)) %>% filter(Size != 0) %>% mutate(
, , ,
num_jobs , .
num_jobs <- 7
purrr .
1:1000 %>% map_df(shuffle_job) %>% filter(sd == min(sd)) %>% pull(data) %>% pluck(1)
, . Bash-
for . 10 . , . , .
for DESIRED_CHR in "16" "9" "7" "21" "MT" do
:
sudo shutdown -h now
… ! AWS CLI
user_data Bash- . , .
aws ec2 run-instances ...\ --tag-specifications "ResourceType=instance,Tags=[{Key=Name,Value=<<job_name>>}]" \ --user-data file://<<job_script_loc>>
!
: API .
- . , . API .
.rds Parquet-, , . R-.
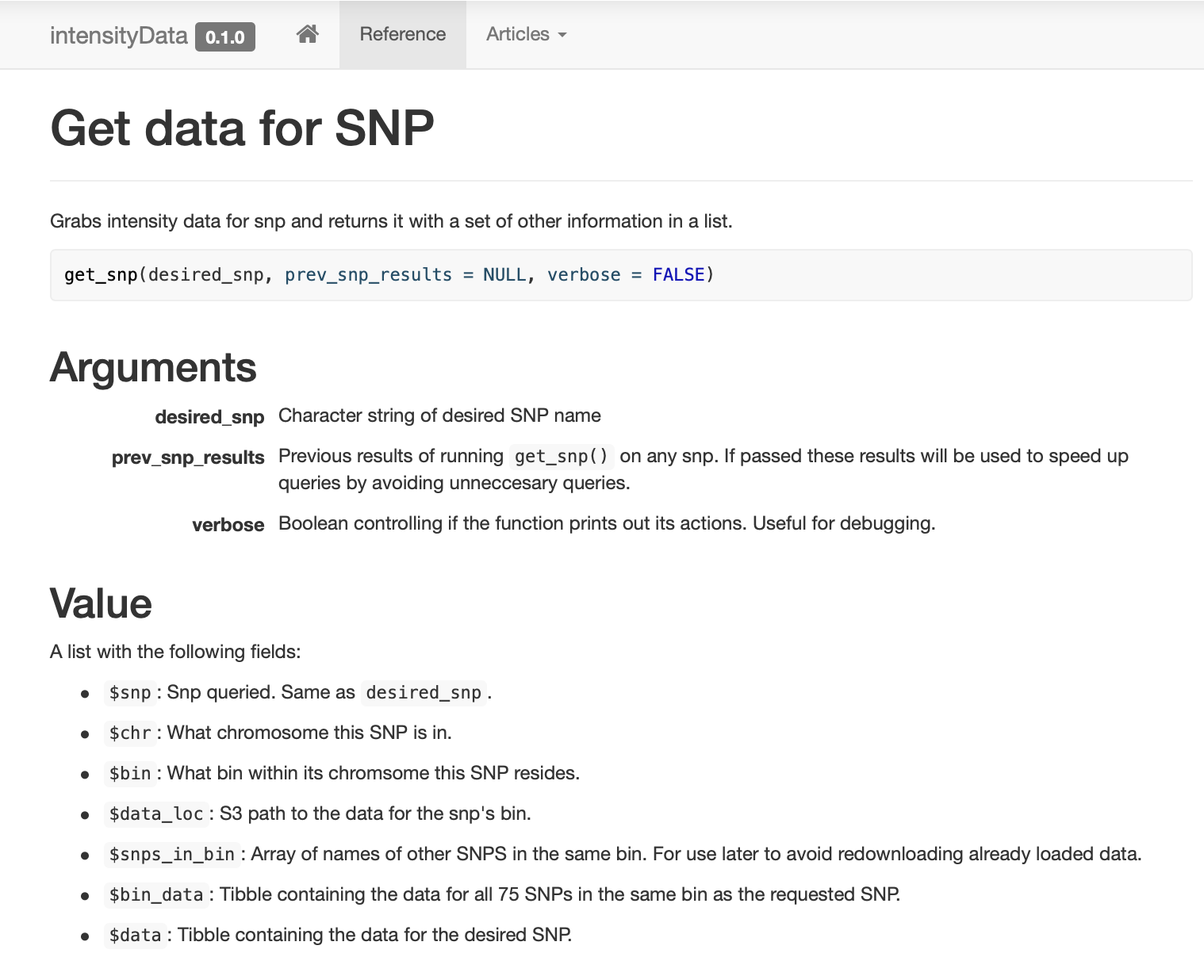
, ,
get_snp .
pkgdown , .
: , !
SNP , (binning) . SNP, (bin). ( ) .
, . , . ,
dplyr::filter , , .
,
prev_snp_results snps_in_bin . SNP (bin), , . SNP (bin) :
النتائج
( ) , . , . .
, , , …
. . ( ), , (bin) , SNP 0,1 , , S3 .
استنتاج
— . , . , . , , , . , , , , . , , , , - .
. , , «» , . .
:
- 25 ;
- Parquet- ;
- Spark ;
- 2,5 ;
- , Spark;
- ;
- Spark , ;
- , , - 1980-;
gnu parallel — , ;
- Spark ;
- Spark ;
- AWK ;
stdin stdout R-, ;
- S3 ;
- — ;
- , ;
- API ;
- , !