Zusammenbau von Schaltkreisen aus biologischen Schaltern
← Vorheriger Artikel im ZyklusDie Logik, Operons verschiedener Stoffwechselwege in E. coli einzubeziehen, liegt nahe an den Elementen AND und OR. Basierend auf diesen können Sie daher ein Genkonstrukt erstellen, das ein beliebiges logisches Schema implementiert (nicht zu kompliziert). Es gibt Programme, zum Beispiel Cello, die aus der Beschreibung der Logik in Verilog eine Sequenz eines Genkonstrukts erstellen, das diese Logik implementiert.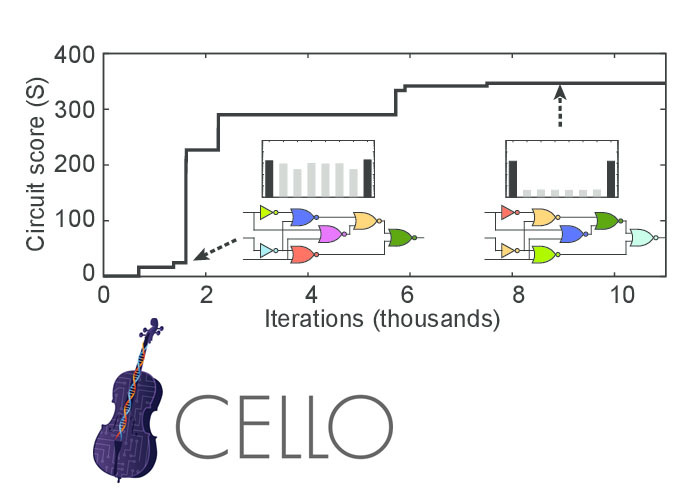
Dynamik des Genwechsels
Im letzten Artikel haben wir die Funktionsweise des Laktoseoperons mit einem einfachen Modell mit vier Parametern modelliert. Die Zeit war nicht Teil dieses Modells. Daher beschreibt es tatsächlich nur den Gleichgewichtszustand, wenn die Eingangssignalpegel für eine lange Zeit konstant bleiben. Um schnelle Prozesse beim Ein- und Ausschalten von Genen zu modellieren, müssen zusätzliche Faktoren berücksichtigt werden.Erstens gibt es eine Transkriptionsverzögerung: Vom Beginn der Arbeit der RNA-Polymerase bis zum Ende der Synthese von mRNA und dann von Proteinen vergeht eine bemerkenswerte Zeit, etwa 10 Minuten in Escherichia coli und ab einer Stunde oder mehr beim Menschen. Daher hängt die Geschwindigkeit des Auftretens neuer Proteinmoleküle nicht vom aktuellen Zustand des Promotors seines Gens ab, sondern vom Zustand des Promotors vor einigen Minuten.Zweitens wird jedes Protein nicht nur synthetisiert, sondern auch abgebaut. Im einfachsten Fall wird der Abbau des Proteins nicht kontrolliert und jede Minute wird ein fester Anteil der Moleküle dieses Proteins in der Zelle abgebaut. Je mehr sie sind, desto mehr trennen sie sich. Das heißt, das Protein hat eine Halbwertszeit, während der seine Konzentration um die Hälfte abnimmt.Weitere Details für die Neugierigsten— - . , — .
Um die Dynamik des Ein- und Ausschaltens des Gens zu beschreiben, müssen wir einen Diffur schreiben:
Dabei ist A die Genaktivität (dieselbe Eingabefunktion aus dem vorherigen Artikel), Tau die Transkriptionsverzögerungszeit und k die Proteinabbaurate. Wenn A zuerst gleich Null ist und dann abrupt ansteigt und auf einem konstanten Niveau bleibt, sehen Änderungen der Proteinmenge im Laufe der Zeit folgendermaßen aus:Motive in Gennetzwerken und ihre Funktionen
Das einfachste für Escherichia coli charakteristische Motiv - die Rückkopplungsschleife - besteht aus einem Gen, das sich selbst reguliert. Meistens ist das Gen ein Repressor für sich selbst, dh die Rückkopplung in der Schleife ist negativ. Wenn dies der wichtigste regulatorische Input ist, dient diese Rückkopplung normalerweise dazu, das Niveau des vom Gen codierten Proteins zu stabilisieren. Eine solche Rückkopplungsschleife hält beispielsweise ein konstantes Niveau des Laktoserepressors in der Zelle aufrecht. Wenn es neben negativer Rückkopplung noch andere regulatorische Eingaben gibt, beschleunigt die Rückkopplung die Genantwort auf ein externes Signal.(Gestrichelte Linien sind theoretische Kurven, durchgezogene Linien sind das Ergebnis des Experiments. Grüne Linien mit negativer Rückkopplung, blaue Linien ohne Rückkopplung)Positives Feedback (ein Protein aktiviert sein eigenes Gen) führt normalerweise zum Auslösen von Verhalten. Ein solches Gen hat zwei stabile Zustände: Entweder funktioniert es überhaupt nicht oder es funktioniert mit voller Kapazität. Ein starkes, aber kurzes externes Signal kann ein Gen von einem stabilen Zustand in einen anderen übertragen, in dem es bis zum nächsten Signal verbleibt. Ein solches Motiv kann als Erinnerungselement dienen. Wenn ein Gen Rauschen an einem regulatorischen Eingang „fängt“, führt eine positive Rückkopplung zu einer starken Ausbreitung der Genaktivität zwischen benachbarten Zellen, die unter denselben Bedingungen wachsen. Dies wird verwendet, um die Heterogenität der zusammenwachsenden Bakterien aufrechtzuerhalten, so dass plötzlicher Stress nicht jeden auf einmal tötet.Beispielsweise stören viele Antibiotika die Proteinsynthese. Wenn die Zelle nicht wächst und keine Proteine synthetisiert, kann sie die Zugabe von Antibiotika zum Medium leicht überleben und später zum Wachstum zurückkehren. In E. coli befinden sich selbst unter idealen Bedingungen bis zu 0,01% der Zellen im Falle einer plötzlichen Vergiftung der Umwelt in einem solchen Winterschlaf. Diese ruhenden Zellen (sie werden als "Persistoren" bezeichnet) unterscheiden sich nicht von den übrigen DNA-Zellen. Sie treten unter der Kontrolle eines Gens mit positiver Rückkopplung und Rauscheingang in den Winterschlaf ein und verlassen ihn.Ein weiteres häufiges Motiv von Gennetzwerken ist etwas komplizierter. Es wird als "Feed-Forward-Schleife" bezeichnet und besteht aus drei Genen: Das X-Gen reguliert die Y- und Z-Gene, das Y-Gen reguliert Z. Jede der drei Verbindungen in der Schleife kann aktiviert oder unterdrückt werden, daher sind im Prinzip 8 Typen möglich solche Schleifen. In der Realität werden normalerweise zwei Achtertypen gefunden: Entweder sind die X- und Y-Gene Aktivatoren (Typ 1-Kohärenzschleife) oder X ist der Y- und Z-Aktivator, Y ist der Z-Repressor (Typ 1-Inkohärenzschleife).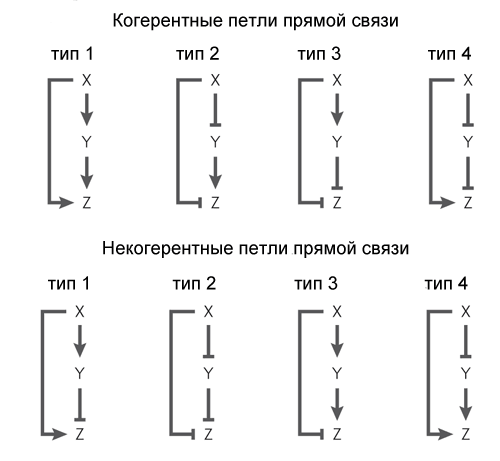
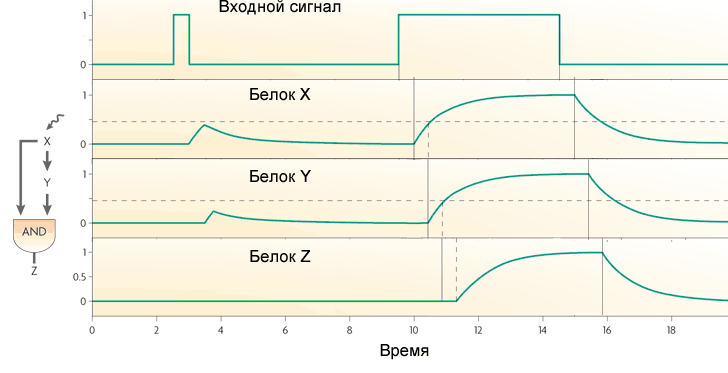 Wenn das Z-Gen wie OR funktioniert, funktioniert die Schleife wird das Gegenteil: Es leitet alle Impulse an den Ausgang weiter und verwandelt kurze in lange. Aber sie verpasst keine kurzen Pausen zwischen den Impulsen.Die Direktschleife mit Aktivator X und Repressor Y wirkt als Hochpassfilter. Es erzeugt Impulse kleiner fester Länge (gleich der Transkriptionsverzögerung des Y-Gens) am Ausgang als Reaktion auf die Vorderflanken der Eingangssignale.Das dritte häufige Motiv in den Gennetzwerken von Escherichia coli ist ein Single-Input-Modul. Es ist einfach eine Gruppe von Genen, die von demselben regulatorischen Gen angetrieben werden. Typischerweise haben Gene in dieser Gruppe unterschiedliche Aktivierungsschwellen, so dass ein sanfter Anstieg der Konzentration des Transkriptionsfaktors Gene in einer bestimmten Reihenfolge umfasst. Ein solches Schema wird beispielsweise bei der Verwaltung von Reaktionen auf Stress verwendet.
Wenn das Z-Gen wie OR funktioniert, funktioniert die Schleife wird das Gegenteil: Es leitet alle Impulse an den Ausgang weiter und verwandelt kurze in lange. Aber sie verpasst keine kurzen Pausen zwischen den Impulsen.Die Direktschleife mit Aktivator X und Repressor Y wirkt als Hochpassfilter. Es erzeugt Impulse kleiner fester Länge (gleich der Transkriptionsverzögerung des Y-Gens) am Ausgang als Reaktion auf die Vorderflanken der Eingangssignale.Das dritte häufige Motiv in den Gennetzwerken von Escherichia coli ist ein Single-Input-Modul. Es ist einfach eine Gruppe von Genen, die von demselben regulatorischen Gen angetrieben werden. Typischerweise haben Gene in dieser Gruppe unterschiedliche Aktivierungsschwellen, so dass ein sanfter Anstieg der Konzentration des Transkriptionsfaktors Gene in einer bestimmten Reihenfolge umfasst. Ein solches Schema wird beispielsweise bei der Verwaltung von Reaktionen auf Stress verwendet.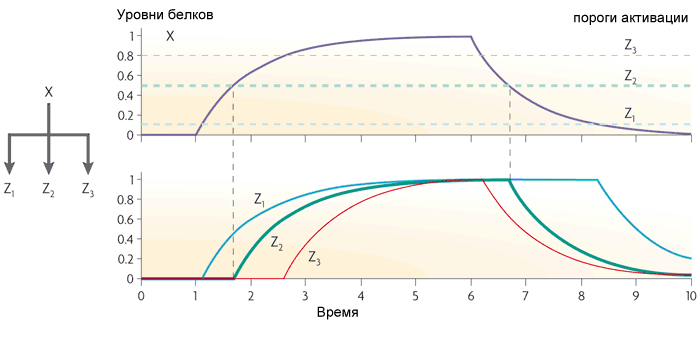 Das vierte, komplexeste Motiv ist das Multi-Input-Modul oder das dicht überlappende Regulon. Es besteht aus mehreren regulatorischen Genen, die mehrere "Output" -Gene steuern.Abhängig von der Logik der Regulation jedes Ausgangsgens kann dieses Motiv sehr unterschiedlich wirken. Es kann mit einer Anordnung von Logikelementen verglichen werden, die je nach Verbindung ein Decoder, ein Zähler oder etwas anderes sein können. Eine vollständige Beschreibung der Logik der Arbeit solcher Motive existiert noch nicht.Mit diesen vier Motiven ist die Struktur der Gennetzwerke von Escherichia coli oder Hefe praktisch erschöpft. Auf ihrer Grundlage werden bereits verschiedene künstliche Schemata erstellt. Zum Beispiel Bakterien mit einem Oszillationsgenerator, der sich auch in benachbarten Zellen synchronisiert, und die Ausgabe von Lumineszenzproteinen- Ein komplettes Analogon von "Blink a Glühbirne" für Mikrocontroller und "Hello World" für andere Programmierer. Durch Hinzufügen eines „Quorum-Gefühls“ zu diesem System (wenn Bakterien erkennen, wie viele Cousins sie umgeben) und Ändern des Lumineszenzausstoßes in Toxine haben Wissenschaftler Bakterien gesammelt, die in Krebstumoren kriechen und sich in großen Mengen ansammeln. Sie sterben zusammen, setzen Toxine frei und töten Tumor ( mehr Details hier ).Aber das sind Bakterien und Hefen. Tiergen-Netzwerke sind viel komplexer. Im nächsten Teil werden wir eines der gut untersuchten Tiergen-Netzwerke und die Probleme analysieren, die bei ihrer Untersuchung und Modellierung auftreten.
Das vierte, komplexeste Motiv ist das Multi-Input-Modul oder das dicht überlappende Regulon. Es besteht aus mehreren regulatorischen Genen, die mehrere "Output" -Gene steuern.Abhängig von der Logik der Regulation jedes Ausgangsgens kann dieses Motiv sehr unterschiedlich wirken. Es kann mit einer Anordnung von Logikelementen verglichen werden, die je nach Verbindung ein Decoder, ein Zähler oder etwas anderes sein können. Eine vollständige Beschreibung der Logik der Arbeit solcher Motive existiert noch nicht.Mit diesen vier Motiven ist die Struktur der Gennetzwerke von Escherichia coli oder Hefe praktisch erschöpft. Auf ihrer Grundlage werden bereits verschiedene künstliche Schemata erstellt. Zum Beispiel Bakterien mit einem Oszillationsgenerator, der sich auch in benachbarten Zellen synchronisiert, und die Ausgabe von Lumineszenzproteinen- Ein komplettes Analogon von "Blink a Glühbirne" für Mikrocontroller und "Hello World" für andere Programmierer. Durch Hinzufügen eines „Quorum-Gefühls“ zu diesem System (wenn Bakterien erkennen, wie viele Cousins sie umgeben) und Ändern des Lumineszenzausstoßes in Toxine haben Wissenschaftler Bakterien gesammelt, die in Krebstumoren kriechen und sich in großen Mengen ansammeln. Sie sterben zusammen, setzen Toxine frei und töten Tumor ( mehr Details hier ).Aber das sind Bakterien und Hefen. Tiergen-Netzwerke sind viel komplexer. Im nächsten Teil werden wir eines der gut untersuchten Tiergen-Netzwerke und die Probleme analysieren, die bei ihrer Untersuchung und Modellierung auftreten. Source: https://habr.com/ru/post/de400899/
All Articles