
Trotz der offensichtlichen Hindernisse und Schwierigkeiten, die manchmal der Entwicklung und Implementierung gentechnischer Produkte im Wege stehen, kann man sich das 21. Jahrhundert nicht ohne die Früchte dieser wichtigen und vielfältigen Technologie im Arsenal eines modernen Biologen vorstellen. Der am häufigsten verwendete Organismus bei GI sind Bakterien.
Was ist GI und warum brauchen wir es? Warum sind Bakterien bei Gentechnikern so beliebt? In welcher Form ist es am einfachsten, das gewünschte Gen in das Bakterium einzuführen? Welche Schwierigkeiten können bei der Arbeit mit diesen Organismen auftreten? Was ist vorher passiert: die Entstehung des ersten gentechnisch veränderten Bakteriums oder die Entdeckung der Struktur von DNA und Genom? Lesen Sie darüber und vieles mehr unter der Katze.
0. Kurzes Bildungsprogramm in Biologie
Dieser Absatz enthält eine kurze Beschreibung des sogenannten
zentralen Dogmas der Molekularbiologie . Wenn Sie über Grundkenntnisse in Molekularbiologie verfügen, können Sie mit Schritt 1 fortfahren.
 Das zentrale Dogma der Molekularbiologie in einem Bild
Das zentrale Dogma der Molekularbiologie in einem BildAlso fangen wir an. Alle Informationen über alle Entwicklungsstadien und die Eigenschaften eines Organismus, ob
Prokaryoten (Bakterien),
Archaeen oder
Eukaryoten (alle übrigen sind einfach und vielzellig), werden in genomischer DNA kodiert, einem Komplex aus zwei zueinander komplementären Polynukleotidketten, die eine Doppelhelix bilden ( komplementäre DNA-Nukleotide: AT und GC). Eukaryontische Chromosomen sind lineare doppelsträngige DNA-Moleküle, und prokaryotische Chromosomen sind geloopt. Oft machen Gene nur einen kleinen Teil des gesamten Genoms aus (beim Menschen - etwa 1,5%).
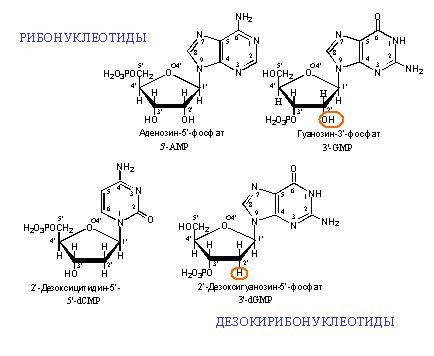 Beispiele für DNA- und RNA-Monomere. "Deoxy" im DNA-Namen bedeutet das Fehlen eines Sauerstoffatoms in Position 2 '(in der Abbildung ist Position 2' rot eingekreist).
Beispiele für DNA- und RNA-Monomere. "Deoxy" im DNA-Namen bedeutet das Fehlen eines Sauerstoffatoms in Position 2 '(in der Abbildung ist Position 2' rot eingekreist).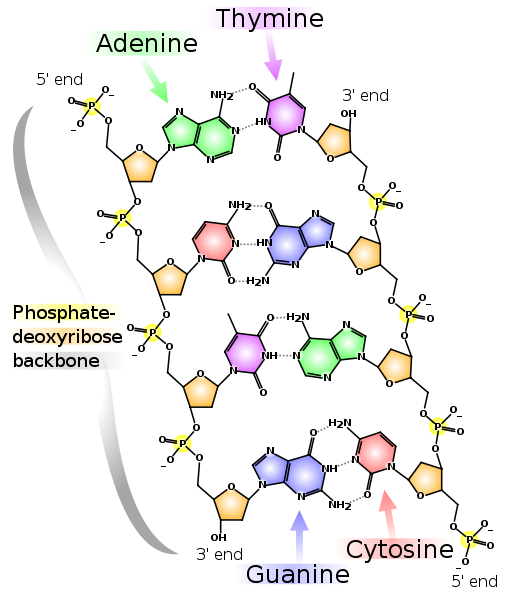 Zwei komplementäre DNA-Stränge. Die gestrichelten Linien zeigen die Wasserstoffbrücken zwischen den Basen. Wie zu sehen ist, bilden Adenin und Thymin zwei Wasserstoffbrückenbindungen untereinander und Guanin und Cytosin drei. Daher ist die GC-Bindung stärker und die GC-reichen Abschnitte doppelsträngiger DNA sind schwieriger in zwei Ketten zu trennen.
Zwei komplementäre DNA-Stränge. Die gestrichelten Linien zeigen die Wasserstoffbrücken zwischen den Basen. Wie zu sehen ist, bilden Adenin und Thymin zwei Wasserstoffbrückenbindungen untereinander und Guanin und Cytosin drei. Daher ist die GC-Bindung stärker und die GC-reichen Abschnitte doppelsträngiger DNA sind schwieriger in zwei Ketten zu trennen.Beachten Sie, dass jede der Ketten ein 5'-Ende und ein 3'-Ende hat. Es ist zu sehen, dass nahe dem 5'-Ende der linken Kette das 3'-Ende der rechten und umgekehrt ist, daher werden die Ketten "antiparallel" genannt. RNA hat auch ein 5'- und 3'-Ende. Die Positionen 5 'und 3' selbst wurden ausgewählt, um den Anfang und das Ende anzuzeigen, da durch sie kovalente Bindungen in den Ketten von DNA und RNA gebildet werden.
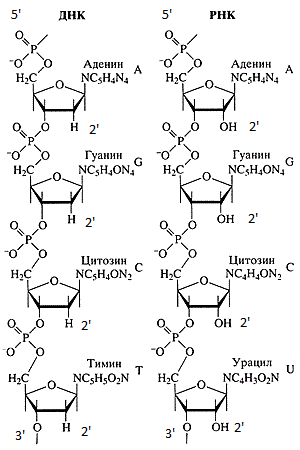 DNA- und RNA-Ketten.
DNA- und RNA-Ketten.DNA- und RNA-Sequenzen werden immer vom 5'-Ende bis zum 3'-Ende aufgezeichnet. Dafür gibt es eine Reihe von Gründen:
- Die Synthese neuer DNA- und RNA-Ketten beginnt am 5'-Ende ( DNA-Polymerase (Enzyme, die eine komplementäre DNA-Kette auf einer DNA- oder RNA-Matrix synthetisieren) und RNA-Polymerase (Enzyme, die eine komplementäre RNA-Kette auf einer DNA- oder RNA-Matrix synthetisieren)). Richtung 3 '-> 5', so dass eine neue Kette in Richtung 5 '-> 3' synthetisiert wird);
- Das Ribosom liest die Codons und bewegt sich entlang der mRNA in Richtung 5 '-> 3';
- Die Aminosäuresequenz ist in der DNA-Codierungskette in der Richtung 5 '-> 3' geschrieben (ein signifikanter Teil der mRNA ist eine exakte Kopie der DNA-Codierungsregion, wobei Thymin durch Uracil ersetzt ist und an Position 2 'natürlich eine Hydroxylgruppe (-OH) anstelle von Wasserstoff vorhanden ist);
- Schließlich ist es nur praktisch, eine allgemein akzeptierte Aufnahmeregel zu haben.
Ein Gen ist ein Teil der genomischen DNA, der die Sequenz der Nukleotide eines RNA-Moleküls definiert:
- Codierende RNA: Messenger-RNA (mRNA), in der die Aminosäuresequenz des entsprechenden Proteins als Codons codiert ist. Sie können auch den Namen "Informations-RNA" finden, dann sieht die Abkürzung wie "mRNA" aus;
- Nichtkodierende RNA: Transport-RNA, ribosomale RNA und andere.
Die Rolle von tRNA besteht darin, Aminosäuren an den mRNA-Ribosomenkomplex abzugeben. Darüber hinaus ist tRNA für die Erkennung von mRNA-Codons verantwortlich. Dazu enthält jede tRNA das sogenannte „Anticodon“ - ein zum mRNA-Codon komplementäres Triplett.
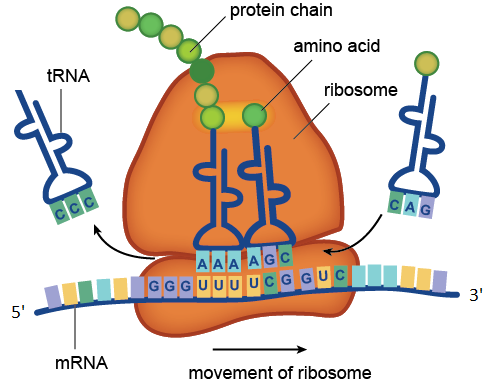 Der durch das Ribosom katalysierte Translationsprozess. In der Figur werden die in der mRNA enthaltenen UUU- und UCG-Codons von den in den tRNA-Molekülen enthaltenen AAA- und AGC-Anticodons erkannt. Transport-RNA mit Anticodon-CCC hat der wachsenden Proteinkette bereits ihre Aminosäure gegeben, und tRNA mit Anticodon-CAG wartet in der Schlange. Die in der Figur gezeigte Darstellung des mRNA-Moleküls besteht aus vier Codons: GGGUUUUCGGUC. Das Codon GGG entspricht der Aminosäure Glycin, UUU Phenylalanin, UCG Serin, GUC Valin. Diese mRNA-Region codiert also ein Proteinfragment mit der Aminosäuresequenz Glycin-Phenylalanin-Serin-Valin.
Der durch das Ribosom katalysierte Translationsprozess. In der Figur werden die in der mRNA enthaltenen UUU- und UCG-Codons von den in den tRNA-Molekülen enthaltenen AAA- und AGC-Anticodons erkannt. Transport-RNA mit Anticodon-CCC hat der wachsenden Proteinkette bereits ihre Aminosäure gegeben, und tRNA mit Anticodon-CAG wartet in der Schlange. Die in der Figur gezeigte Darstellung des mRNA-Moleküls besteht aus vier Codons: GGGUUUUCGGUC. Das Codon GGG entspricht der Aminosäure Glycin, UUU Phenylalanin, UCG Serin, GUC Valin. Diese mRNA-Region codiert also ein Proteinfragment mit der Aminosäuresequenz Glycin-Phenylalanin-Serin-Valin.Ribosomale RNAs sind unverzichtbare Bestandteile des Ribosoms. Die Hauptfunktion von rRNA besteht darin, den Translationsprozess sicherzustellen: Sie ist daran beteiligt, Informationen aus mRNA unter Verwendung von Adaptermolekülen von tRNA zu lesen und die Bildung von Peptidbindungen zwischen an tRNA gebundenen Aminosäuren und der wachsenden Proteinkette zu katalysieren.
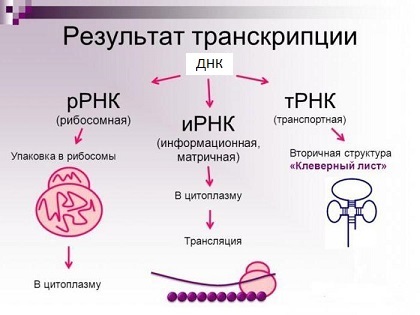 Die Haupttypen von RNA-Molekülen (tatsächlich gibt es viel mehr).
Die Haupttypen von RNA-Molekülen (tatsächlich gibt es viel mehr).Ein Protein hingegen ist eine Kette von Aminosäuren, die über eine Peptidbindung kovalent miteinander verbunden sind (Sie können sehen, wie es im Spoiler etwas weiter ist). Nach der Synthese sollte die Aminosäurekette eine bestimmte räumliche Struktur annehmen - "
Konformation " (
sie haben mir bereits über die räumliche Struktur von Proteinen
bei Geektimes berichtet ). Darüber hinaus bestehen viele große Proteine tatsächlich aus mehreren Proteinen, die durch hydrophobe Wechselwirkungen und Wasserstoffbrückenbindungen zu einer einzigen stabilen Struktur kombiniert werden. In diesem Fall wird jedes der "Bauproteine" als "Untereinheit" bezeichnet, und das resultierende große Protein wird als "Multisubunit" bezeichnet.
20 Aminosäuren, aus denen Proteine bestehen  Ribosomaler Komplex. Das Bild stammt aus der OlegKovalevskiy- Publikation „3D-Druck von Proteinmolekülmodellen“ .
Ribosomaler Komplex. Das Bild stammt aus der OlegKovalevskiy- Publikation „3D-Druck von Proteinmolekülmodellen“ .Im Fall von Genen, die ein Protein codieren, sieht der Prozess der Decodierung genetischer Informationen folgendermaßen aus:
- Die RNA-Polymerase erkennt den Promotor und bindet an ihn (wenn er "offen" ist, werden wir die Regulation der Aktivität des Promotors weiter diskutieren);
- Basierend auf dem Prinzip der Komplementarität synthetisiert das RNA-Polymeraseenzym auf einer DNA-Matrix den "Blindwert" der Matrizen-RNA (Prä-mRNA, in Eukaryoten) oder der fertigen funktionellen mRNA (in Prokaryoten). Dieser Vorgang wird als "Transkription" bezeichnet .
- (nur in Eukaryoten) Das Prä-mRNA-Molekül wird modifiziert ("reift") und wird zu funktioneller mRNA;
- mRNA wird vom Ribosom erkannt, einem Enzym, das den Triplettcode der mRNA dekodiert und darauf basierend ein Peptid / Protein synthetisiert. Die Aminosäuren, aus denen das Ribosom das Protein bildet, werden im Komplex mit Transport-RNA ( tRNA ) abgegeben. Dieser Vorgang wird als "Rundfunk" bezeichnet .
- Das Peptid / Protein kann posttranslationale Modifikationen erfahren ("Reifung" in Analogie zu mRNA) und wird funktionsfähig. Ein wichtiger Faktor ist, dass das System der posttranslationalen Modifikation von Eukaryoten viel komplexer und vielfältiger ist als das von Prokaryoten. Daher kann nicht jedes eukaryotische Protein von einem Bakterium korrekt synthetisiert werden.
Zusätzlich zu den kodierenden Regionen enthält das Genom zahlreiche Fragmente, die auf die eine oder andere Weise auch an der Transkription beteiligt sind. In der Nähe des Gens befindliche und als Promotoren bezeichnete Parzellen werden von RNA-Polymerasen erkannt (sie sagen, dass das Gen unter der Kontrolle dieses Promotors steht). Unterschiedliche Promotoren werden von unterschiedlichen RNA-Polymerasen erkannt. Beispielsweise wird ein Gen unter der Kontrolle eines
Bakteriophagen- Promotors nicht in Bakterien transkribiert, wenn die RNA-Polymerase des entsprechenden Bakteriophagen nicht darin synthetisiert wird.
Im Allgemeinen .
Jedes Gen kann auch mehrere regulatorische Sequenzen aufweisen, die sich entweder direkt in der Nähe des Promotors befinden (oder sogar mit diesem überlappen) oder in einem Abstand von Zehntausenden von Nukleotidpaaren von diesem. Transkriptionsverstärkende Elemente werden als
"Enhancer" bezeichnet, transkriptionsunterdrückende Elemente werden als Schalldämpfer bezeichnet, und die Proteine, die mit ihnen interagieren, werden als
Transkriptionsfaktoren bezeichnet . Obwohl es auch üblich ist, Transkriptionsfaktoren als die notwendigen Komponenten des Transkriptionsinitiationskomplexes zu bezeichnen, ohne die eine Transkription im Prinzip nicht möglich ist. Tatsache ist, dass nur um die Synthese des RNA-Moleküls auf der DNA-Matrix in Eukaryoten und Archaeen zu starten, der Aufbau des gesamten supramolekularen Komplexes erforderlich ist. Der einfachste derartige Komplex umfasst das RNA-Polymerase-Holoenzym und sechs sogenannte
"gemeinsame Transkriptionsfaktoren" (TFIIA, TFIIB, TFIID, TFIIE, TFIIF und TFIIH). Der Komplex selbst wird als
„Transkriptionsvorinitiationskomplex“ bezeichnet (
Video , jede Komponente des Komplexes wird in der einen oder anderen Farbe hervorgehoben).
Der prokaryotische Transkriptionskomplex ist völlig anders, so dass es keinen Sinn macht, das eukaryotische Gen mit dem eukaryotischen Promotor in das Bakterium einzubetten. Ein prokaryotisches Analogon der üblichen Transkriptionsfaktoren von Eukaryoten und Archaeen kann als Protein bezeichnet werden, das als
"Sigma-Faktor" bezeichnet wird .
 Prokaryotischer Transkriptionskomplex. Die in der Figur gezeigten Buchstaben sind allgemein anerkannte Bezeichnungen der entsprechenden Untereinheiten. σ70 - Sigma-Faktor von E. coli- Haushaltsgenen
Prokaryotischer Transkriptionskomplex. Die in der Figur gezeigten Buchstaben sind allgemein anerkannte Bezeichnungen der entsprechenden Untereinheiten. σ70 - Sigma-Faktor von E. coli- HaushaltsgenenDie Genome von Prokaryoten und Eukaryoten haben viele Gemeinsamkeiten, und das bereits erwähnte zentrale Dogma der Molekularbiologie gilt für beide Königreiche. Es gibt jedoch auch viele signifikante Unterschiede. Zum Beispiel ist ein Bakterium durch ein System von Operons gekennzeichnet - Gene, die zusammen gruppiert sind und am selben Prozess beteiligt sind und nicht separat, sondern als Teil einer langen mRNA transkribiert werden. Bei Eukaryoten ist alles völlig anders: Die an einem Prozess beteiligten Gene sind über verschiedene Chromosomen verteilt, und die Gene selbst sind
durch nichtkodierende Regionen
von Introns in kodierende Fragmente
von Exons unterteilt. In diesem Fall wird das Gen zunächst vollständig transkribiert, und dann werden bereits im RNA-Stadium die Introns herausgeschnitten und die Exons vernetzt, um die kodierende mRNA zu bilden. Dieser Vorgang wird als
Spleißen bezeichnet . Gleichzeitig können nicht alle verfügbaren Exons in die fertige mRNA eingenäht werden, sondern nur ein Teil davon spricht in diesem Fall von
„alternativem Spleißen“ . Somit kann eine eukaryotische Zelle mehrere Proteine synthetisieren, während sie dasselbe Gen transkribiert. Dies hat unter anderem eine sehr wichtige Konsequenz: Es macht oft keinen Sinn, das eukaryotische Gen „wie im Chromosom“ in das Bakterium einzufügen, da das Bakterium einfach nicht spleißen kann.
Es gibt noch einen weiteren wichtigen Unterschied. Prokaryoten zeichnen sich durch das Vorhandensein von DNA-basiertem genetischem Material außerhalb des Ring- "Chromosoms" aus, den sogenannten
"Plasmiden" - kleinen kreisförmigen doppelsträngigen DNA-Molekülen. Außerdem fehlen Prokaryoten Organellen, einschließlich des Kerns: Alle Komponenten einer Bakterienzelle können sich frei im intrazellulären Raum bewegen. Eukaryoten haben jedoch keine Plasmide, aber es gibt
Plastiden und
Mitochondrien, in deren Genom Plasmide enthalten sind (nach der fundiertesten Hypothese sind Plastiden und Mitochondrien „Nachkommen“ der prokaryotischen Architektur des Genoms von Cyanobakterien und Bakterien, die von alten einzelligen Proto-Eukaryoten eingeschlossen werden). Darüber hinaus ist das Vorhandensein eines Kerns und anderer intrazellulärer Kompartimente, die von einer eigenen Membran umgeben sind, bereits typisch für Eukaryoten. Daher erfordert die Gentechnik von eukaryotischen Zellen andere Ansätze als die Gentechnik von Bakterien.
Der genetische Code selbst ist wie folgt aufgebaut. Jedes Gen / Exon besteht aus einem Satz von Tripletts / Codons - Sequenzen von drei Nukleotiden, zwischen denen keine Lücken bestehen. Die Triplett-Organisation gilt sowohl für Gene in der DNA als auch für den kodierenden Teil der mRNA. Während des Translationsprozesses „erkennen“ Transport-RNAs (tRNAs), die eine bestimmte Aminosäure tragen, ihre entsprechenden Drei-Buchstaben-Tripletts. Das Ribosom trennt die Aminosäure von der tRNA und bindet sie an die wachsende Aminosäurekette, die am Ende der Translation entweder sofort zu einem reifen, voll funktionsfähigen Protein wird oder bevor sie zusätzlich eine Reihe von Modifikationen erfährt. In diesem Fall entspricht nur eine Aminosäure jedem Triplett, aber mehrere verschiedene Codons können einer Aminosäure entsprechen. Dies ist verständlich, da es im genetischen Standardcode 61 codierende Codons und
nur 20 proteinogene Aminosäuren gibt (Gesamtcodons natürlich 4 * 4 * 4 = 64, aber drei davon sind nicht codierend, stattdessen dienen sie als Signal zum Stoppen der Translation und werden " Codons stoppen ”).
 Codons im genetischen Standardcode. Danke an Wikipedia für das Bild.
Codons im genetischen Standardcode. Danke an Wikipedia für das Bild.Proteine sind also genau jene Elemente, die das letzte Glied in der Kette zwischen genomischer DNA und den Eigenschaften des Körpers sind, dem sogenannten
„Phänotyp“ . Um die für uns wichtigen Eigenschaften des Organismus irgendwie zu verändern, müssen wir daher seine DNA so verändern, dass infolgedessen bestimmte Proteine in seinen Zellen erscheinen, die uns das Zielergebnis liefern. Dies ist die Grundidee aller Gentechnik.
1) Zu welchen Zwecken werden Bakterien in der Gentechnik eingesetzt und warum?
Also haben wir herausgefunden, wie und warum die Sequenz der genomischen DNA die Eigenschaften und Merkmale des Körpers beeinflusst. Natürlich ist es sehr gut, wenn das Merkmal vollständig durch nur ein Gen bestimmt wird - das Einfügen eines kleinen Fragments ist kein ernstes Problem mehr. Beispielsweise wird die Resistenz einer Pflanze gegen ein Herbizid oder einen Schädling häufig durch ein einzelnes Gen bestimmt, so dass es in solchen Fällen nicht schwierig ist, Sorten mit der gewünschten Resistenz zu erzeugen (im Gegensatz dazu, eine solche Pflanze auf den Markt zu bringen). Gleiches gilt für viele Antibiotikaresistenzen von Bakterien (tatsächlich haben Bakterien viele Schutzmechanismen gegen Antibiotika, aber sie wirken auf unabhängige Weise). Das entgegengesetzte Beispiel ist beispielsweise ein Versuch von Wissenschaftlern, Pflanzen beizubringen, Stickstoff aus der Atmosphäre zu absorbieren. Tatsache ist, dass die einzige Stickstoffquelle für Pflanzen der Boden ist, in dem stickstoffhaltige Verbindungen, die zur Assimilation durch die Pflanze geeignet sind, von Mikroorganismen synthetisiert werden (oder von einem fürsorglichen Gärtner oder einem vorbeikommenden Hund in Form von Düngemitteln eingebracht werden). Offensichtlich wäre die Schaffung einer Pflanze mit einem alternativen Ernährungsmechanismus für die Landwirtschaft sehr vorteilhaft. Leider ist dieser Prozess so kompliziert, dass das Problem seiner "Übertragung" vom Mikroorganismus auf die Pflanze bisher nicht gelöst wurde.
Wenn unser Ziel schließlich darin besteht, Protein für einen bestimmten Zweck zu erhalten (Struktur und Funktionen des Proteins zu untersuchen, darauf basierende medizinische Präparate oder Laborreagenzien herzustellen usw.), dann sind wir natürlich auch sehr zufrieden mit der Integration eines einzelnen Gens in die Zelle, das In diesem Fall ist es üblich, den "Erzeugerorganismus" zu nennen.
Ein Bakterium in der Gentechnik ist ein potenzielles Ausgangsmaterial für die Schaffung von:
- ein Produzent des Proteins, das wir im Labor- oder Industriemaßstab benötigen;
- Wirkstoff bei einer bestimmten chemischen Umwandlung einer Verbindung in eine andere, sei es ein Fermentationsprozess in der Lebensmittelindustrie, die Schaffung günstigerer Bedingungen für das Pflanzenwachstum durch die Einführung eines „Bakteriendüngerherstellers“ im Boden oder die Entsorgung von Stahlschrott;
- Klonotek von Genen (ein Thema, dessen gute Beschreibung die Größe des Artikels auf unanständig erhöht);
- ein medizinisch bedeutendes Medikament zum Beispiel zur Wiederherstellung der Mikroflora des Magen-Darm-Trakts;
- Bakterienstämme von Agrobacterium tumefaciens zur anschließenden genetischen Veränderung von Pflanzen.
* Ich könnte etwas vergessen, daher sind Ergänzungen in den Kommentaren willkommen.
Eine interessante Tatsache ist, dass die ersten erfolgreichen Experimente auf dem Gebiet der Gentechnik von Bakterien lange vor der wegweisenden Arbeit von Watson und Crick stattfanden. Darüber hinaus wurde auf der Grundlage dieser Experimente die Tatsache bewiesen, dass die Informationen in der DNA enthalten sind, wonach die Wissenschaftler ihre Zeit nicht mehr mit Hypothesen über RNA und Protein verbringen konnten.
Diese Arbeit, die 1944 durchgeführt wurde, ist als
Avery-, MacLeod- und McCarthy-Experiment bekannt und basiert auf der
Arbeit von Frederick Griffith , bei der festgestellt wurde, dass eine Infektion mit abgetöteten pathogenen und lebenden nicht pathogenen Pneumokokkenstämmen die Entwicklung der Krankheit verursacht einzeln verursachen sie keine signifikanten Symptome. Aus diesem Experiment wurde geschlossen, dass ein abgetötetes Bakterium in der Lage ist, etwas an einen nicht pathogenen „Kollegen“ zu übertragen, wodurch es gefährlich wird. Aber was geben sie aneinander weiter? Bis 1944 gab es drei Hauptkandidaten: DNA, RNA und Protein. Um den Transporter zu etablieren, wurde ein elegantes Experiment durchgeführt: Zu diesem Zeitpunkt waren bereits Enzyme verfügbar, die in der Lage waren, DNA (DNase), RNA (RNase) und Proteine (Proteinase) getrennt zu zerstören. Es wurde gezeigt, dass die Übertragung pathogener Eigenschaften nicht nur in den Fällen auftrat, in denen die Herstellung eines toten pathogenen Stammes mit DNase behandelt wurde und nicht von der Behandlung des Arzneimittels mit RNase und Proteinase abhängig war.
Somit wurde nachgewiesen, dass DNA der Informationsträger über die Zeichen ist. Darüber hinaus wurde deutlich gezeigt, dass ein spontanes Eindringen eines fremden DNA-Moleküls in eine Bakterienzelle möglich ist.
Warum sind Bakterien mit offensichtlichen Mängeln so beliebt (zum Beispiel dem Fehlen eukaryotischer posttranslationaler Modifikationen)? Alles ist einfach.
Sie sind unprätentiös im Betrieb, einfach zu bedienen und erfordern keine teuren Nährmedien.2) Wie entsteht ein genetisches Konstrukt, das in das Bakterium eingeführt wird?
Die moderne Gentechnik von Bakterien besteht hauptsächlich in der Einführung eines Plasmidvektors (eines modifizierten Bakterienplasmids, das das Zielgen und eine Reihe anderer notwendiger Elemente enthält, auf die weiter unten eingegangen wird). Das Ändern des Bakterienchromosoms ist weniger typisch, aber dieses Verfahren ist auch nicht ungewöhnlich: Beispielsweise wurde das T7-Bakteriophagen-RNA-Polymerase-Gen unter Verwendung eines auf Prophagen λ basierenden Vektors während der Erzeugung eines der in Laborplaque beliebten Stämme in das E. coli-Chromosom eingeführt . Es gibt drei Gründe, warum sich ein Forscher häufig dafür entscheidet, ein Gen in einen Plasmidvektor einzuführen:Ein typischer Plasmidvektor für die Arbeit mit Bakterien ist ein kleines zirkuläres doppelsträngiges DNA-Molekül, das das Zielproteingen unter der Kontrolle eines spezifischen Promotors und einer Reihe notwendiger Gene und regulatorischer Elemente trägt, deren Vorhandensein eine konstante Menge an Plasmid in der Zelle sicherstellt ("Kopierkontrolle"). Selbst im Fall einer hocheffizienten Synthese von mRNA ist der Vektor offensichtlich von geringem Nutzen, wenn er in einem Bakterium in der Menge eines Paares von Stücken vorhanden ist: Während des Teilungsprozesses ist die Wahrscheinlichkeit der Bildung von Tochterzellen ohne das notwendige Plasmid banal.Neben dem Gen und dem Promotor sind die Hauptelemente des Plasmidvektors:- ori ist die Ursprungsregion der Plasmidreplikation. Erforderlich, um eine konstante Menge an Plasmid und dessen Vererbung durch Tochterzellen aufrechtzuerhalten;
- — , , , . , , (« »). , , . , .
, β- (GUS). , . , . — (GFP) ( GUS GFP );
- , ( — , — );
- — , ( ). , «» .
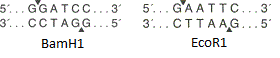 Die Figur zeigt die Seiten der Restriktionsendonuklease BamH1 und die EcoR1 . Beide Enzyme erkennen eine bestimmte Stelle aus sechs Basenpaaren und führen an verschiedenen Stellen Einzelstrangbrüche ein (angezeigt durch Dreieckspfeile). In diesem Fall fallen die Kettenbruchpunkte nicht zusammen, was bedeutet, dass "klebrige Enden" gebildet werden (wenn sie zusammenfallen, werden "stumpfe Enden" gebildet).
Die Figur zeigt die Seiten der Restriktionsendonuklease BamH1 und die EcoR1 . Beide Enzyme erkennen eine bestimmte Stelle aus sechs Basenpaaren und führen an verschiedenen Stellen Einzelstrangbrüche ein (angezeigt durch Dreieckspfeile). In diesem Fall fallen die Kettenbruchpunkte nicht zusammen, was bedeutet, dass "klebrige Enden" gebildet werden (wenn sie zusammenfallen, werden "stumpfe Enden" gebildet).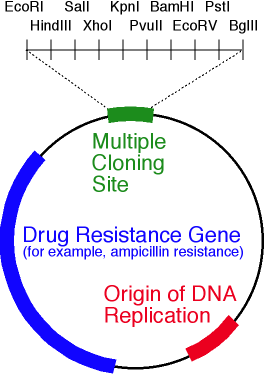 Ein vereinfachtes Schema eines Plasmidvektors. Die Figur zeigt ori, das Antibiotikaresistenzgen und den Polylinker, der 10 Restriktionsendonuklease-Stellen enthält.Nun, der Vektor liegt in unseren Händen. Wie kann man ein Gen darin einbetten? Und wo kann ich dieses Gen bekommen?Angenommen, wir kennen die Nukleotidsequenz des benötigten Gens. Gehen Sie dann wie folgt vor:
Ein vereinfachtes Schema eines Plasmidvektors. Die Figur zeigt ori, das Antibiotikaresistenzgen und den Polylinker, der 10 Restriktionsendonuklease-Stellen enthält.Nun, der Vektor liegt in unseren Händen. Wie kann man ein Gen darin einbetten? Und wo kann ich dieses Gen bekommen?Angenommen, wir kennen die Nukleotidsequenz des benötigten Gens. Gehen Sie dann wie folgt vor:- , ;
- .
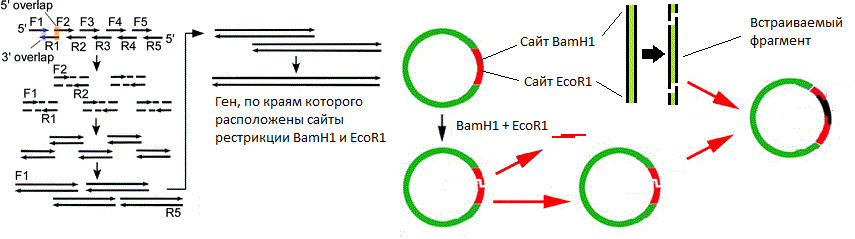 ( ). .Überlappende Primer werden so synthetisiert, dass nach dem Zusammenbau ein Gen voller Größe gebildet wird, an dessen Seiten sich Schnittstellen mit denselben Endonukleasen befinden, deren Stellen sich im Polylinker des Vektors befinden. Somit wird , wenn der Prozess notwendigen Endonukleasen (in meinem Beispiel sind Enzyme BamH1 und die EcoR1 ) Vektor und das Gen zusammengebaut wird, dann bilden sie klebrige Enden , die „einander kennen“ kann aufgrund von Wechselwirkungen von komplementären Nukleotiden sticky ends. Anerkennung allein reicht jedoch nicht aus, da sich die klebrigen Enden bisher nur aufgrund fragiler Wasserstoffbrückenbindungen aneinander festhalten. Dieses Problem wird gelöst, indem dem Reaktionsgemisch gleichzeitig geschnittener Vektor, geschnittenes Gen und DNA-Ligaseenzym zugesetzt werden., die Kettenbrüche in doppelsträngigen DNA-Molekülen eliminiert.Ein weiterer wichtiger Faktor bei der Genassemblierung ist die Tatsache, dass die Frequenzen des einen oder anderen Codons für verschiedene Organismen unterschiedlich sind, während in den Zellen normalerweise mehr dieser tRNAs vorhanden sind, die „populäreren“ Codons entsprechen. Da viele Aminosäuren von mehreren Codons codiert werden, ist es sehr wahrscheinlich, dass das sinnlose Kopieren eines Gens von einem Organismus zu einem anderen zu einer starken Verzögerung des Übersetzungsprozesses führen kann. Während viele Codons dieses Gens im neuen Organismus selten sind, wartet das Ribosom länger, wenn die gewünschte tRNA endlich eintrifft.
( ). .Überlappende Primer werden so synthetisiert, dass nach dem Zusammenbau ein Gen voller Größe gebildet wird, an dessen Seiten sich Schnittstellen mit denselben Endonukleasen befinden, deren Stellen sich im Polylinker des Vektors befinden. Somit wird , wenn der Prozess notwendigen Endonukleasen (in meinem Beispiel sind Enzyme BamH1 und die EcoR1 ) Vektor und das Gen zusammengebaut wird, dann bilden sie klebrige Enden , die „einander kennen“ kann aufgrund von Wechselwirkungen von komplementären Nukleotiden sticky ends. Anerkennung allein reicht jedoch nicht aus, da sich die klebrigen Enden bisher nur aufgrund fragiler Wasserstoffbrückenbindungen aneinander festhalten. Dieses Problem wird gelöst, indem dem Reaktionsgemisch gleichzeitig geschnittener Vektor, geschnittenes Gen und DNA-Ligaseenzym zugesetzt werden., die Kettenbrüche in doppelsträngigen DNA-Molekülen eliminiert.Ein weiterer wichtiger Faktor bei der Genassemblierung ist die Tatsache, dass die Frequenzen des einen oder anderen Codons für verschiedene Organismen unterschiedlich sind, während in den Zellen normalerweise mehr dieser tRNAs vorhanden sind, die „populäreren“ Codons entsprechen. Da viele Aminosäuren von mehreren Codons codiert werden, ist es sehr wahrscheinlich, dass das sinnlose Kopieren eines Gens von einem Organismus zu einem anderen zu einer starken Verzögerung des Übersetzungsprozesses führen kann. Während viele Codons dieses Gens im neuen Organismus selten sind, wartet das Ribosom länger, wenn die gewünschte tRNA endlich eintrifft.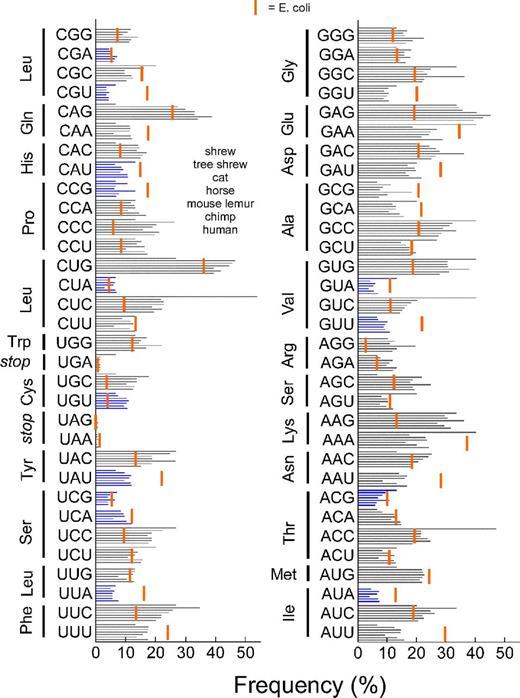 , , , , , . E. coli .Nun ein paar Worte zum Promoter. Die Auswahl eines geeigneten Promotors ist sehr wichtig, da der Transkriptionsprozess weitgehend davon abhängt. Promotoren werden bedingt in stark, mittel und schwach unterteilt. Die "Stärke" des Promotors wird dadurch bestimmt, wie aktiv die Gene unter seiner Kontrolle transkribiert werden, wobei alle anderen Dinge gleich sind: Je aktiver die Transkription, desto stärker der Promotor. Wenn wir einen Proteinproduzenten schaffen wollen, sollten wir natürlich mit starken Promotoren beginnen. In einigen Fällen schädigt eine zu schnelle Transkription (daher aktive Translation) die Zelle. In diesem Fall können Sie versuchen, einen schwächeren Promotor zu verwenden. Obwohl es in der Tat viel einfacher ist, die Transkriptionsaktivität eines bestehenden Produzenten zu beeinflussen, als einen neuen zu erstellen.Eine andere Sache ist wichtig. Oft wirken sich die vom Vektor codierten Proteine äußerst negativ auf die Lebensfähigkeit der Bakterien aus. Die Synthese dieser Proteine nimmt nicht nur eine große Menge an Ressourcen in Anspruch (und die Menge des Zielproteins sollte mindestens 10% des Gesamttrockengewichts der Zelle betragen), sondern sie schweben auch mit einer toten Last im Zytoplasma hin und her ! Daher ist es vorerst besser, die Expression eines der Zelle fremden Gens vollständig auszuschalten. Zu diesem Zweck wurden kontrollierte Expressionssysteme entwickelt, mit denen Sie die Expression des benötigten Gens "auf Befehl" "aktivieren" können. Die häufigsten sind:
, , , , , . E. coli .Nun ein paar Worte zum Promoter. Die Auswahl eines geeigneten Promotors ist sehr wichtig, da der Transkriptionsprozess weitgehend davon abhängt. Promotoren werden bedingt in stark, mittel und schwach unterteilt. Die "Stärke" des Promotors wird dadurch bestimmt, wie aktiv die Gene unter seiner Kontrolle transkribiert werden, wobei alle anderen Dinge gleich sind: Je aktiver die Transkription, desto stärker der Promotor. Wenn wir einen Proteinproduzenten schaffen wollen, sollten wir natürlich mit starken Promotoren beginnen. In einigen Fällen schädigt eine zu schnelle Transkription (daher aktive Translation) die Zelle. In diesem Fall können Sie versuchen, einen schwächeren Promotor zu verwenden. Obwohl es in der Tat viel einfacher ist, die Transkriptionsaktivität eines bestehenden Produzenten zu beeinflussen, als einen neuen zu erstellen.Eine andere Sache ist wichtig. Oft wirken sich die vom Vektor codierten Proteine äußerst negativ auf die Lebensfähigkeit der Bakterien aus. Die Synthese dieser Proteine nimmt nicht nur eine große Menge an Ressourcen in Anspruch (und die Menge des Zielproteins sollte mindestens 10% des Gesamttrockengewichts der Zelle betragen), sondern sie schweben auch mit einer toten Last im Zytoplasma hin und her ! Daher ist es vorerst besser, die Expression eines der Zelle fremden Gens vollständig auszuschalten. Zu diesem Zweck wurden kontrollierte Expressionssysteme entwickelt, mit denen Sie die Expression des benötigten Gens "auf Befehl" "aktivieren" können. Die häufigsten sind:- Ein System, das auf regulatorischen Elementen des Lactoseoperons E. coli ( lac- Peron) und einem starken Promotor basiert.
Tatsache ist, dass E. coli seine eigenen Ernährungsregeln hat. Erstens gibt es einen Mechanismus zur Unterdrückung der Aktivität des lac- Perons, der nur aktiviert wird, wenn keine Laktose in die Zelle gelangt. Das ist logisch: Warum Energie für die Synthese dessen verschwenden, was nicht nützlich ist? Sobald jedoch Laktose in ausreichenden Mengen in die Zelle gelangt, wird dieser Mechanismus ausgeschaltet.
Es gibt jedoch einen zweiten Mechanismus zur Unterdrückung der Aktivität des lac- Perons. Befindet sich Glucose im Medium, ernährt sich die Zelle ausschließlich von Glucose, da sie den zweiten Mechanismus der Hemmung der Transkription des lac- Perons aktiviert. Somit ist das lac- Peron nur dann aktiv, wenn sich nur Laktose in dem die Zelle umgebenden Raum befindet. Das Minus des Lactose-Operons ist ein extrem schwacher Promotor, daher wird es in Produzentenstämmen durch einen starken ersetzt. Starke Promotoren stammen häufig von Krankheitserregern. Die stärksten Promotoren, die in der Gentechnik von Prokaryoten am häufigsten verwendet werden, werden aus bakteriellen Viren - Bakteriophagen - isoliert. Beispielsweise ist der T7-Phagenpromotor weit verbreitet.
Übrigens werden auch einige starke Promotoren für die Gentechnik von Pflanzen aus Viren isoliert, beispielsweise ist dies der Promotor des Blumenkohlmosaikvirus.

Wie oben erwähnt, hat E. coli keine RNA-Polymerase, die Bakteriophagen-Promotoren erkennt, daher wird das RNA-Polymerase-Gen des entsprechenden Bakteriophagen zuvor in den Produzenten inseriert.
Das beliebte Proteinsynthesesystem auf E. coli- Basis trägt das T7-Phagen-RNA-Polymerase-Gen unter der Kontrolle des bakteriellen RNA-Polymerase-Promotors, der durch den Lacron- Mechanismus reguliert wird. Wenn dieser Stamm mit einem Vektor transformiert wird, der das Zielgen unter der Kontrolle des Komplexes "Phagen-T7-Promotor + Regulation des Promotor-Typ- Lacperon " trägt, entsteht ein zweistufiger Mechanismus zur Hemmung der Transkription des Zielgens .
Bei Verwendung dieses Designs werden dem Nährmedium gleichzeitig Glucose und Lactose zugesetzt. Für einige Zeit ernähren sich die Zellen von Glukose und teilen sich leise, da die Synthese eines Fremdproteins vollständig unterdrückt wird. Wenn die Glukose vorbei ist und die Zellen auf den Laktosestoffwechsel umstellen, ist bereits genügend Biomasse in der Kultur vorhanden. Es ist nur an der Zeit, mit der Synthese des benötigten Proteins zu beginnen. Dieses Verfahren wird als "automatische Induktion" bezeichnet.
Sie können es auch auf andere Weise tun: Fügen Sie dem Nährmedium keine Glukose und Laktose hinzu und fügen Sie dann, wenn die Kultur die gewünschte Dichte erreicht hat, das hinzu, was die Zelle für Laktose nimmt, aber nicht metabolisieren oder zerstören kann. Jetzt wird IPTG als solcher Induktor verwendet.
- Ein System, das auf dem Regulationsmechanismus des pL-Promotors des Bakteriophagen λ basiert.
Dieser Promotor wird durch das cI-Repressorprotein inaktiviert. Gleichzeitig wurde eine wärmeempfindliche Form dieses Proteins namens cI857 entdeckt: Dieser Transkriptionsfaktor behält seine Funktionalität bei einer Temperatur von etwa 30 ° C und verliert sie bei 42 ° C. Daher wird bei Verwendung eines solchen Systems die Bakterienkultur zuerst bei 30 ° C auf die gewünschte Dichte gezüchtet und dann die Temperatur auf 42 ° C erhöht, wodurch die Synthese des Zielproteins gestartet wird.
Nun, der Vektor ist entworfen. Dann ist das Kleine, eine geeignete Methode für die Einführung in die Bakterienzelle zu finden. Aber das ist eine ganz andere Geschichte.