
Als ich einen Artikel über die Entwicklung eines Anomaliedetektors schrieb, implementierte ich einen der Algorithmen namens Incremental Growing Neural Gas.
In Sowjetische Literatur Im russischen Segment des Internets wird dieses Thema eher schlecht behandelt, und es gab nur einen Artikel , und selbst dann mit der Anwendung dieses Algorithmus.
Was genau ist ein inkrementell wachsender neuronaler Gasalgorithmus?
Einführung
IGNG ist wie GNG ein adaptiver Clustering-Algorithmus.
Der Algorithmus selbst wird in einem Artikel von Prudent und Ennadji für 2005 beschrieben .
Wie bei GNG gibt es viele Datenvektoren X oder Erzeugungsfunktion f(t) , die Vektoren aus zufällig verteilten Daten liefert (Parameter t - Zeit oder Probennummer in der Probe).
Der Algorithmus legt diesen Daten keine zusätzlichen Einschränkungen auf.
Aber innen ist ganz anders als bei GNG.
Dieser Algorithmus ist auch insofern interessant, als er etwas genauer ist als die Neurogenese von GNG-Modellen.
Beschreibung des Algorithmus
Der Algorithmus zerlegt viele Daten in Cluster.
Im Vergleich zu GNG ist der Vorteil eine höhere Konvergenzrate.
Ideen, auf denen der Algorithmus basiert:
- Adaptive Resonanztheorie : Zuerst wird das nächste Neuron gesucht, und wenn die Differenz den Schwellenwert (den „Wachsamkeitsparameter“) nicht überschreitet, werden die Gewichte angepasst oder auf andere Weise die Neuronenkoordinaten im Datenraum geändert. Wenn der Schwellenwert nicht überschritten wurde, werden neue Neuronen erzeugt, die den Wert der Datenprobe besser approximieren.
- Sowohl Verbindungen als auch Neuronen haben einen Altersparameter (GNG hat nur Verbindungen), der zunächst Null ist, aber mit dem Lernen zunimmt.
- Ein Neuron erscheint nicht sofort: Zuerst erscheint ein Embryo (oder ein Keimneuron), dessen Alter mit jeder Iteration zunimmt, bis es reift. Nach dem Training nehmen nur reife Neuronen an der Klassifizierung teil .
Hauptzyklus
Die Arbeit beginnt mit einem leeren Diagramm. Parameter sigma initialisiert durch die Standardabweichung der Trainingsprobe:
sigma= sqrt frac1N sum limitNi=1 left(xi− barx right)2
Wo: barx - der Durchschnitt zwischen den Koordinaten der Stichprobe.
Die Hauptschleife bei jedem Schritt verringert den Wert sigma Dies ist die Näherungsschwelle und berechnet die Differenz zwischen der vorherigen Stufe der Clusterqualität und der Stufe, die nach der Clusterbildung durch das IGNG-Verfahren erhalten wurde .
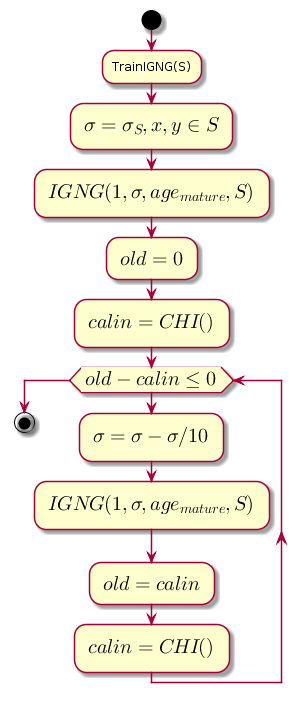
Diagrammcode.@startuml start :TrainIGNG(S); :<latex>\sigma = \sigma_S,x,y \in S</latex>; :<latex>IGNG(1, \sigma, age_{mature}, S)</latex>; :<latex>old = 0</latex>; :<latex>calin = CHI()</latex>; while (<latex>old - calin \leq 0</latex>) :<latex>\sigma=\sigma - \sigma / 10</latex>; :<latex>IGNG(1, \sigma, age_{mature}, S)</latex>; :<latex>old = calin</latex>; :<latex>calin = CHI()</latex>; endwhile stop @enduml
CHI ist der Kalinsky-Kharabaz-Index, der die Qualität der Clusterbildung zeigt:
CHI= fracB/(c−1)W/(n−c)
Wo:
- n - die Anzahl der Datenproben.
- c - die Anzahl der Cluster (in diesem Fall die Anzahl der Neuronen).
- B - Matrix der internen Dispersion (die Summe der quadratischen Abstände zwischen den Koordinaten der Neuronen und dem Durchschnitt aller Daten).
- W - Matrix der externen Dispersion (die Summe der quadratischen Abstände zwischen allen Daten und dem nächsten Neuron).
Je größer der Indexwert ist, desto besser, denn wenn die Differenz zwischen den Indizes nach dem Clustering und bevor sie negativ ist, kann angenommen werden, dass der Index positiv wurde und den vorherigen überschritt, d. H. Clustering erfolgreich abgeschlossen.
IGNG-Verfahren
Dies ist die grundlegende Prozedur des Algorithmus.
Es ist in drei sich gegenseitig ausschließende Phasen unterteilt:
- Keine Neuronen gefunden.
- Ein zufriedenstellendes Neuron wurde gefunden.
- Es wurden zwei gefunden, die die Bedingungen des Neurons erfüllen.
Wenn eine der Bedingungen erfüllt ist, werden die anderen Schritte nicht ausgeführt.
Zunächst wird ein Neuron nach der besten ungefähren Datenprobe durchsucht:
c1=min(dist( xi, omegac))
Hier dist(x omega,x xi) - Entfernungsberechnungsfunktion, die normalerweise eine euklidische Metrik ist .
Wenn das Neuron nicht gefunden wird oder zu weit von den Daten entfernt ist, d.h. erfüllt das Näherungskriterium nicht dist( xi, omegac) leq sigma wird ein neues embryonales Neuron mit Koordinaten erzeugt, die den Koordinaten der Probe im Datenraum entsprechen.

Wenn die Näherungsprüfung bestanden wurde, wird ein zweites Neuron auf die gleiche Weise durchsucht und auf Nähe zur Datenprobe geprüft.
Wenn das zweite Neuron nicht gefunden wird, wird es erstellt.

Wenn zwei Neuronen gefunden wurden, die die Bedingung der Nähe zur Datenprobe erfüllen, werden ihre Koordinaten gemäß der folgenden Formel korrigiert:
epsilon(t)hc,ci= beginFälle epsilonb, ifc=ci epsilonn, ifesgibtdieVerbindungzwischenc=ci0,inothercase endcase
Delta omegac= epsilon(t)hc,c1 parallel xi− omegac parallel omegac= omegac+ Delta omegac
Wo:
- epsilon(t) - Anpassungsschritt.
- ci Ist die Nummer des Neurons.
- hc,c1 - Neuronen-Nachbarschaftsfunktion c mit dem Gewinner-Neuron (in diesem Fall wird 1 für direkte Nachbarn zurückgegeben, andernfalls 0 für den Anpassungsschritt zur Berechnung omega wird nur für direkte Nachbarn ungleich Null sein).
Mit anderen Worten wird die Koordinate (Gewicht) des siegreichen Neurons in geändert epsilonb∗ Delta omegai und alle seine direkten Nachbarn (die durch eine Kante des Diagramms mit ihm verbunden sind) auf epsilonn∗ Delta omegai wo omegai - Koordinate des entsprechenden Neurons vor der Änderung.
Dann wird eine Verbindung zwischen den beiden siegreichen Neuronen hergestellt, und wenn sie bereits hergestellt wurde, wird ihr Alter zurückgesetzt.
Das Alter aller anderen Beziehungen nimmt zu.
Alle Mitteilungen, deren Alter die Konstante überschritten hat agemax werden gelöscht.
Danach werden alle isolierten (diejenigen, die keine Verbindung zu anderen haben) reifen Neuronen entfernt.
Das Alter der unmittelbaren Neuronen neben dem siegreichen Neuron nimmt zu.
Wenn das Alter eines der Keimbahnneuronen überschritten wird agereife Er wird ein reifes Neuron.
Das endgültige Diagramm enthält nur reife Neuronen.
Eine Bedingung zum Abschließen des folgenden IGNG-Verfahrens kann als feste Anzahl von Zyklen angesehen werden.
Der Algorithmus ist unten dargestellt (das Bild ist anklickbar):
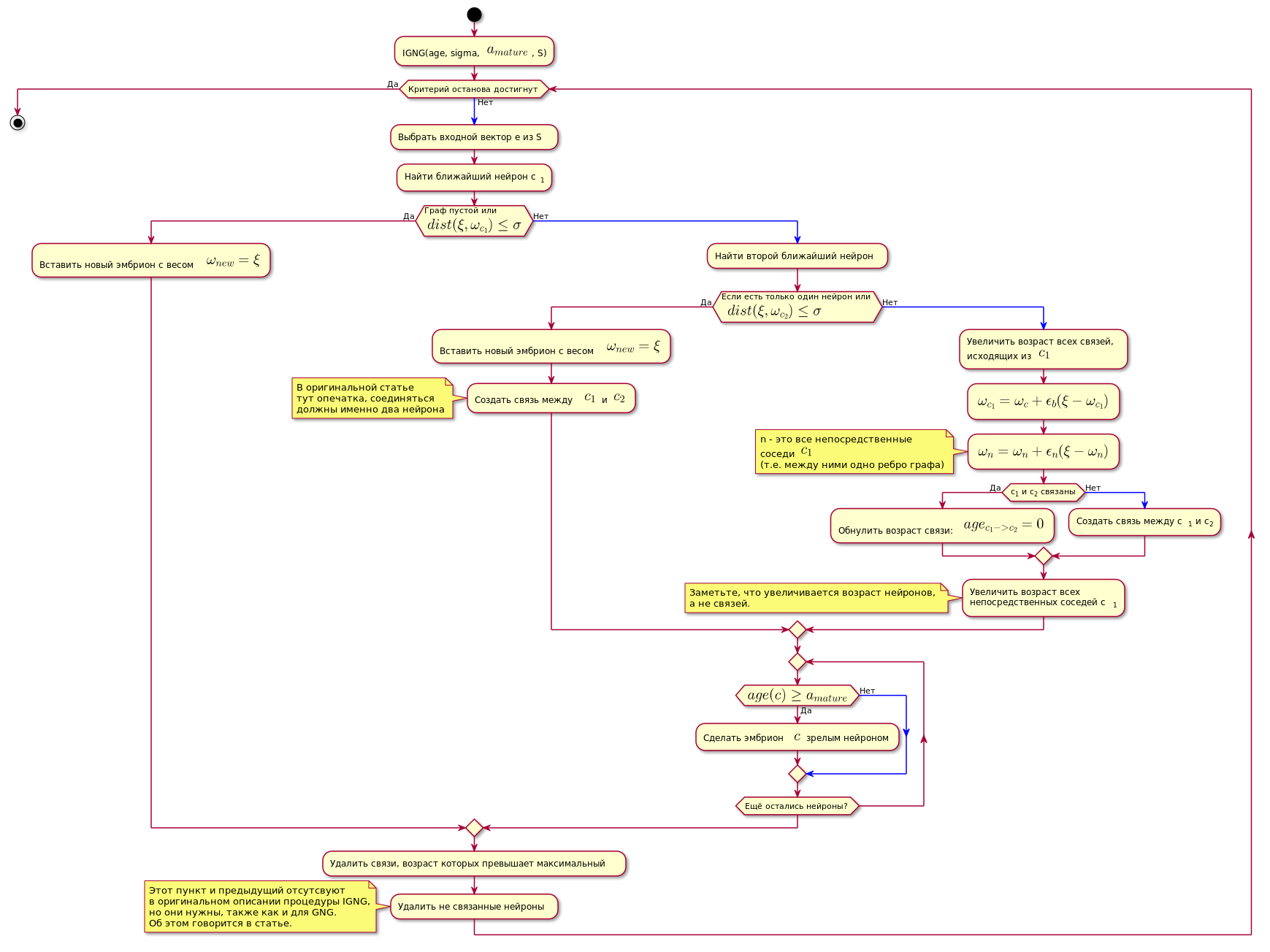
Diagrammcode. @startuml skinparam nodesep 10 skinparam ranksep 20 start :IGNG(age, sigma, <latex>a_{mature}</latex>, S); while ( ) is () -[#blue]-> : e S; : c<sub>1</sub>; if ( \n<latex>dist(\xi, \omega_{c_1}) \leq \sigma</latex>) then () : <latex>\omega_{new} = \xi</latex>; else () -[#blue]-> : ; if ( \n <latex>dist(\xi, \omega_{c_2}) \leq \sigma</latex>) then () : <latex>\omega_{new} = \xi</latex>; : <latex>c_1</latex> <latex>c_2</latex>; note , end note else () -[#blue]-> : ,\n <latex>c_1</latex>; :<latex>\omega_{c_1} = \omega_c + \epsilon_b(\xi - \omega_{c_1})</latex>; :<latex>\omega_n = \omega_n + \epsilon_n(\xi - \omega_n)</latex>; note n - <latex>c_1</latex> (.. ) end note if (c<sub>1</sub> c<sub>2</sub> ) then () : : <latex>age_{c_1 -> c_2} = 0</latex>; else () -[#blue]-> : c<sub>1</sub> c<sub>2</sub>; endif : \n c<sub>1</sub>; note , , . end note endif repeat if (<latex>age(c) \geq a_{mature}</latex>) then () : $$ ; else () -[#blue]-> endif repeat while ( ?) endif : , ; : ; note IGNG, , GNG. . endnote endwhile () stop @enduml
Implementierung
Das Netzwerk wurde in Python mithilfe der NetworkX- Diagrammbibliothek implementiert. Das Ausschneiden des Codes aus dem Prototyp im vorherigen Artikel ist unten angegeben. Es gibt auch kurze Erklärungen für den Code.
Wenn sich jemand für den vollständigen Code interessiert, finden Sie hier einen Link zum Repository .
Ein Beispiel für den Algorithmus:

Der Großteil des Codes class NeuralGas(): __metaclass__ = ABCMeta def __init__(self, data, surface_graph=None, output_images_dir='images'): self._graph = nx.Graph() self._data = data self._surface_graph = surface_graph