Hallo Habr! Heute setzen wir die letzte Geschichte über DNA fort. Darin haben wir darüber gesprochen, wie
viel es passiert, wie DNA gespeichert wird und warum es so wichtig ist . Heute beginnen wir mit einem historischen Hintergrund und enden mit den Grundlagen der Kodierung von Informationen in der DNA.
Die Geschichte
Die DNA selbst wurde bereits 1869 von Johann Friedrich Mischer aus den weißen Blutkörperchen isoliert, die er aus Eiter erhielt. Weiße Blutkörperchen sind weiße Blutkörperchen, die eine Schutzfunktion erfüllen. Es gibt ziemlich viele von ihnen im Eiter, weil sie dazu neigen, Gewebe zu beschädigen, wo Bakterienzellen sie "fressen". Er isolierte eine Substanz, die Stickstoff und Phosphor enthält. Zuerst wurde es das Nuklein genannt, aber als es saure Eigenschaften entdeckte, wurde der Name in Nukleinsäure geändert. Die biologische Funktion der neu entdeckten Substanz war unklar, und es wurde lange Zeit angenommen, dass Phosphor darin gespeichert war. Schon zu Beginn des 20. Jahrhunderts glaubten viele Biologen, dass DNA nichts mit der Übertragung von Informationen zu tun habe, da die Struktur des Moleküls, wie es damals schien, zu eintönig sei und nicht so viele Informationen codieren könne.
Bis 1901 isolierte und beschrieb Albrecht Kossel die fünf Stickstoffbasen, aus denen DNA und RNA bestehen. Wenig später stellte Peter Leuven fest, dass die Kohlenhydratkomponente von Nukleinsäuren Desoxyribose und Ribose sind. Nukleinsäuren, zu denen Ribose gehört, wurden Ribonukleinsäuren oder kurz RNA genannt und solche, die Desoxyribose, Desoxyribonukleinsäuren oder DNA enthielten.
Nun stellte sich die Frage, wie die einzelnen Verbindungen miteinander verbunden sind. Dazu musste der DNA-Strang zerstört werden und untersucht werden, was nach der Zerstörung passieren würde. Hierzu wurde das DNA-Polymer hydrolysiert. Leuven änderte jedoch die Hydrolysemethode. Anstatt viele Stunden in einer sauren Umgebung zu kochen, verwendete er jetzt Enzyme. Diesmal wurden nicht nur einzelne Adenine, Guanine, Thymine, Cytosine, Desoxyribosen und Phosphorsäuren aus Hydrolysaten isoliert, sondern auch größere Fragmente, beispielsweise Verbindungen stickstoffhaltiger Basen mit Kohlenhydraten oder Kohlenhydrate mit Phosphorsäure. Gleichzeitig
wurden in Nukleinsäurehydrolysaten keine Verbindungen gefunden, die aus zwei Stickstoffbasen oder Verbindungen vom Basenphosphorsäuretyp bestehen . Das heißt, es wurde klar, dass sich Phosphorsäure mit Zucker verbindet, und dies wiederum mit einer stickstoffhaltigen Base. Es wurde vorgeschlagen, dass Verbindungen von stickstoffhaltigen Basen mit einem Kohlenhydrat als Nukleoside bezeichnet werden und Phosphorsäureester von Nukleosiden als Nukleotide bezeichnet werden.
Als Ergebnis dieser Arbeiten gelangte Leuven zu dem Schluss, dass Nukleinsäuren Polymere sind. Nukleotide dienen als Monomere. Der Gehalt jedes der vier Nukleotide in DNA oder RNA schien nach der damaligen chemischen Analyse Leven gleich zu sein. Daher schlug Leuven die folgende Theorie der Struktur von Nukleinsäuren vor: Sie sind Polymere, deren Monomere Blöcke von vier in Reihe geschalteten Nukleotiden sind.
Die damalige Theorie der Tetranukleotidstruktur sah durchaus gerechtfertigt aus, da sie in alle Lehrbücher der Vorkriegszeit eingegangen war. Das Problem der DNA-Funktion blieb jedoch unklar. Es dauerte fast ein halbes Jahrhundert, um dieses Problem zu klären.
Es kam eine Zeit, in der Biologen Informationen über die Verteilung von Nukleinsäuren in verschiedenen Arten von tierischen und pflanzlichen Geweben, in Bakterien und Viren in einigen einzelligen Organismen sammelten.
Zu dieser Zeit glaubte die wissenschaftliche Gemeinschaft ernsthaft, dass es Proteine waren, die für die Speicherung genetischer Informationen verantwortlich waren. Die traditionelle Vorstellung von der primären Rolle von Proteinen im Lebensprozess ließ uns nicht glauben, dass eine so wichtige Substanz wie eine Substanz der Vererbung alles andere als Protein sein könnte. Die Struktur der Proteine war äußerst unterschiedlich, was über Nukleinsäuren nicht aussagen konnte. Der berühmte sowjetische Genetiker-Zytologe N.K. Koltsov berechnete, dass durch Variation der Sequenz von 20 Aminosäuren, aus denen das Proteinmolekül besteht, Billionen unterschiedlicher Proteine erzeugt werden können.
Wenn wir in der einfachsten Form drucken möchten, wie logarithmische Tabellen gedruckt werden, würden diese Billionen Moleküle alle vorhandenen Druckereien der Welt zur Erfüllung dieses Plans bereitstellen und 50.000 Bände mit 100 gedruckten Blättern pro Jahr produzieren. Dann wäre so viel Zeit bis zum Ende der durchgeführten Arbeiten vergangen. wie viel ist seit der archäischen Zeit unserer Tage vergangen.
Wirklich viel ... 20 im 20. ... Aber Sequenzen sind viel länger als 20 Aminosäuren.
Und hier ist, was A. R. Kizel darüber schreibt - einer der gelehrtesten Biochemiker der Zeit.
Aus den gerade gegebenen Ansichten über die Rolle der Nukleinsäure ... folgt, dass sie nicht an der Struktur von Genen beteiligt ist und dass die Gene aus einem anderen Material bestehen. Wir kennen dieses Material immer noch nicht zuverlässig, obwohl es in den meisten Fällen direkt als Protein bezeichnet wird.
Der erste Erfolg kam aus der Mikrobiologie. 1944 wurden die Ergebnisse der Experimente von Avery und Mitarbeitern (USA) zur Transformation von Bakterien veröffentlicht. Ein paar Worte zur Transformation.
Die Transformation selbst wurde 1928 vom Mikrobiologen Griffiths entdeckt.
Griffith arbeitete mit Kulturen von Pneumokokken (Streptococcus pneumoniae), dem Erreger einer der Formen der Lungenentzündung. Einige Stämme dieses Bakteriums sind virulent und verursachen eine Lungenentzündung. Ihre Zellen sind mit einer Polysaccharidkapsel beschichtet, die das Bakterium vor der Wirkung des Immunsystems schützt. In Kultur bilden solche Bakterien große glatte Kolonien mit regelmäßiger Kugelform. Aus diesem Grund werden sie S-Stämme genannt (vom Englischen glatt - glatt).
Es gibt verschiedene virulente Pneumokokkenstämme, die sich in Antikörpern unterscheiden, die im Körper produziert werden, wenn Bakterien in den Körper eindringen. Sie werden als IS, IIS, IIIS usw. bezeichnet. Von Zeit zu Zeit mutieren einige Zellen virulenter S-Stämme, verlieren die Fähigkeit, die Polysaccharidmembran zu synthetisieren, und werden avirulent. In der Kultur bilden sie kleine raue Kolonien mit unregelmäßiger Form, weshalb sie als R-Stämme bezeichnet werden (vom englischen Rough - Rough). Manchmal treten umgekehrte Mutationen auf, die die Fähigkeit zur Synthese der Polysaccharidmembran wiederherstellen, jedoch nur in Gruppen der entsprechenden Stämme:
IIS - IIR
IIIS - IIIR
Dies legt nahe, dass avirulente R-Stämme immer dem virulenten S-Stamm der Eltern entsprechen.
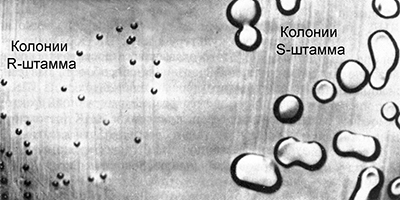
Griffith führte verschiedene Gruppen von Labormäusen in einen virulenten und avirulenten Pneumokokkenstamm ein. In der ersten Kontrollgruppe führte die Injektion eines virulenten IIIS-Stammes zum Tod von Tieren. Tiere der zweiten Kontrollgruppe blieben nach Injektion des avirulenten Stammes IIR am Leben. Danach erhitzte Griffith die Lösung mit der Kultur des virulenten Stammes IIIS auf eine Temperatur von 60 ° C, was zum Tod von Bakterien führte. Er führte Bakterien, die durch Erhitzen getötet wurden, in die dritte Gruppe experimenteller Mäuse ein. Die Tiere blieben am Leben, was im Prinzip zu erwarten war. Dies ist jedoch nicht alles. Er führte Teile überlebender Mäuse in die Bakterien des avirulenten Stammes IIR ein.
Es schien, dass dies keine schrecklichen Konsequenzen für Mäuse haben konnte. Entgegen den Erwartungen starben die Tiere jedoch. Als Bakterien aus ihren Körpern isoliert und in Kultur ausgesät wurden, stellte sich heraus, dass sie zum virulenten Stamm IIIS gehören.

Die Tatsache, dass die Zellen, die den Tod von Mäusen verursachten, eine Polysaccharidmembran vom Typ III anstelle von II synthetisierten, zeigte, dass sie nicht als Ergebnis der umgekehrten Mutation IIR - IIS entstanden sein konnten. Daraus machte Griffith eine sehr wichtige Schlussfolgerung. Avirulente Bakterien des Stammes IIR können sich in irgendeiner Weise in virulente Bakterien verwandeln, die mit hitzegetöteten Bakterien des Stammes IIIS interagieren, die noch im Körper von Mäusen verbleiben. Mit anderen Worten, die avirulenten Bakterien des Stammes IIR erhalten einen Faktor von toten Bakterien des Stammes IIIS, der sie in virulente Bakterien verwandelt. Was dieser Faktor war, wusste Griffith jedoch nicht.
Eigentlich wurde dieses Phänomen als bakterielle Transformation bezeichnet. Es ist eine unidirektionale Übertragung erblicher Merkmale von einer Bakterienzelle auf eine andere.
Nun zurück zu Averys Erfahrungen. Der Aufbau ihrer Experimente ähnelt etwas Griffiths 'Experimenten. Avery und die Mitarbeiter haben es sich zur Aufgabe gemacht, die chemische Natur des Transformators herauszufinden. Sie zerstörten die Suspension von Pneumokokken und entfernten Proteine, Kapselpolysaccharid und RNA aus dem Extrakt, jedoch blieb die transformierende Aktivität des Extrakts erhalten. Die transformierende Aktivität des Arzneimittels ging während seiner Behandlung mit kristallinem Trypsin oder Chymotrypsin (zerstörende Proteine), Ribonuklease (zerstört RNA) nicht verloren. Es war klar, dass das Medikament weder Protein noch RNA war. Die transformierende Aktivität des Arzneimittels ging jedoch vollständig verloren, wenn es mit Desoxyribonuklease (DNA-schädigend) behandelt wurde, und vernachlässigbare Mengen des Enzyms verursachten eine vollständige Inaktivierung des Arzneimittels. Somit wurde gefunden, dass der transformierende Faktor in Bakterien reine DNA ist. Diese Schlussfolgerung war eine bedeutende Entdeckung, und Avery war sich dessen sehr wohl bewusst. Er schrieb, dass dies genau das ist, wovon die Genetik lange geträumt hat, nämlich die Substanz des Gens. Hier scheint es ein Beweis zu sein. Aber zu stark war der Glaube an Protein als Substanz der Vererbung. Einige glaubten, dass die Transformation und jene unbedeutenden Proteinverunreinigungen verursachen kann, die im Arzneimittel verblieben sind.
Der neue Beweis für die direkte genetische Rolle der DNA war die Erfahrung der Virologen Hershey und Chase. Sie arbeiteten mit dem Bakteriophagen T2 (Bakteriophagen - Bakterienviren), der das Bakterium
Escherichia coli (E. coli) infiziert.
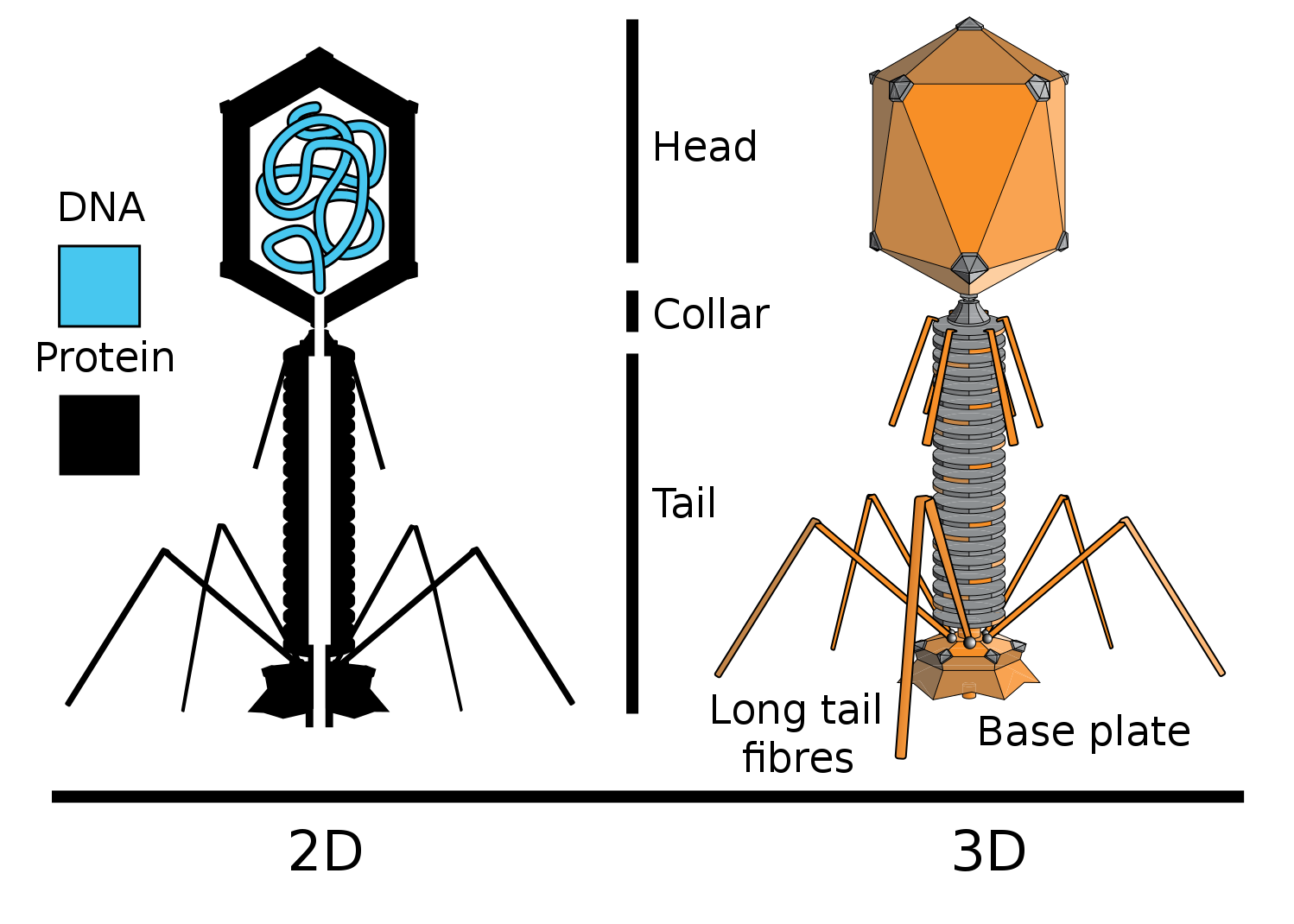
Eigentlich was sie getan haben. Sie enthielten radioaktiven Phosphor in der Zusammensetzung der DNA einiger Bakteriophagen (P32) und das Schwefelisotop in der Zusammensetzung der Proteine anderer (S35). Zu diesem Zweck wurden einige Bakterien auf einem Medium unter Zusatz von radioaktivem Phosphor im Phosphation gezüchtet, während andere auf einem Medium unter Zugabe von radioaktivem Schwefel im Sulfation gezüchtet wurden. Dann wurde den Bakteriophagen T2 zu diesen Bakterien hinzugefügt, die sich in Bakterienzellen vermehrten und eine radioaktive Markierung in ihrer DNA (P ist in DNA, aber nicht in Proteinen) oder Proteinen (S ist in Proteinen, aber nicht in DNA) enthielten.
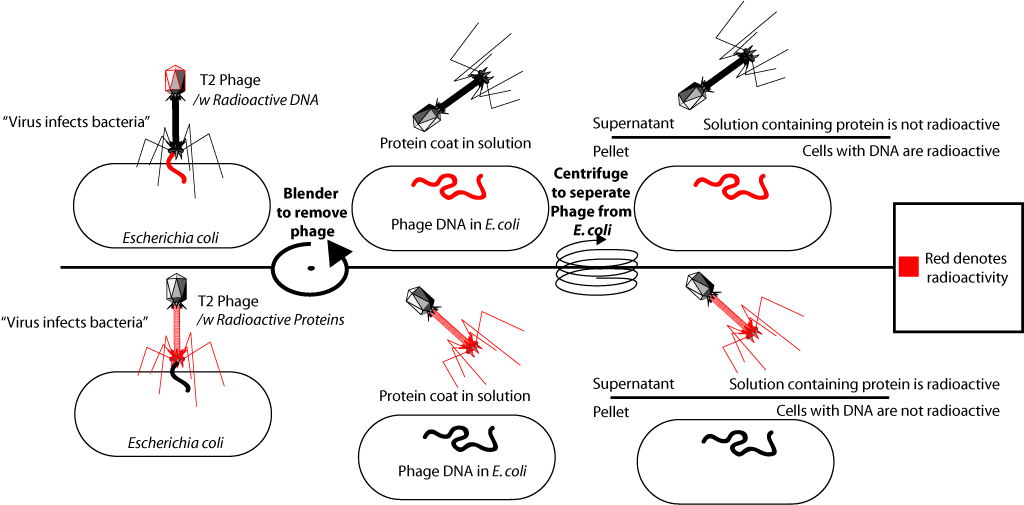
Nach der Isolierung der radioaktiv markierten Bakteriophagen wurden sie der Kultur frischer (isotopenfreier) Bakterien zugesetzt. Was zur Infektion dieser Bakterien führte. Der Bakteriophage bindet an die Bakterienzelle und „injiziert“ deren DNA. Danach wurde das Medium mit den Bakterien in einem speziellen Mischer heftig geschüttelt (es wurde gezeigt, dass die Phagenmembranen von der Oberfläche der Bakterienzellen getrennt wurden), und dann wurden die infizierten Bakterien vom Medium getrennt. Als im ersten Experiment Phosphor-32-markierte Bakteriophagen zu den Bakterien gegeben wurden, stellte sich heraus, dass sich die radioaktive Markierung in den Bakterienzellen befand. Wenn im zweiten Experiment mit Schwefel-35 markierte Bakteriophagen zu Bakterien gegeben wurden, wurde die Markierung in einer Fraktion des Mediums mit Proteinbeschichtungen gefunden, jedoch nicht in den Bakterienzellen. Dies bestätigte, dass das Material, das die Bakterien infizierte, DNA war. Da vollständige Viruspartikel, die Virusproteine enthalten, in infizierten Bakterien gebildet werden, ist dieses Experiment zu einem der entscheidenden Beweise dafür geworden, dass genetische Informationen (Informationen über die Struktur von Proteinen) in der DNA enthalten sind.
Diese Entdeckungen haben viele Biologen dieser Zeit stark beeinflusst. Insbesondere auf den für seine Regeln berühmten Chargaff. Er glaubte, dass Avery im Wesentlichen eine „neue Sprache“ entdeckte oder zumindest zeigte, wo er danach suchen sollte.
Charguff begann nach einem Unterschied in der Nukleotidzusammensetzung und Anordnung der Nukleotide in DNA-Präparaten zu suchen, die aus verschiedenen Quellen erhalten wurden. Und da es zu dieser Zeit keine Methoden gab, um die chemische Charakterisierung von DNA genau zu bestimmen, musste er sich diese einfallen lassen. Ihm wurde gezeigt, dass die alte Tetranukleotid-Theorie der Struktur von Nukleinsäuren falsch ist. DNA in verschiedenen Organismen ist in Zusammensetzung und Struktur sehr unterschiedlich. Gleichzeitig wurden neue Tatsachen entdeckt, die für andere natürliche Polymere bisher nicht bekannt waren, nämlich die Regelmäßigkeit des Verhältnisses einzelner Basen in der Zusammensetzung aller untersuchten DNA. Jetzt kennen sogar Schulkinder sie als die Regeln von Chargaff.
- Die Menge an Adenin ist gleich der Menge an Thymin und Guanin an Cytosin: A = T, G = Ts.
- Die Anzahl der Purine entspricht der Anzahl der Pyrimidine: A + G = T + C.
- Es fließt vom ersten und zweiten. Die Anzahl der Basen mit Aminogruppen in Position 6 entspricht der Anzahl der Basen mit Ketogruppen in Position 6: A + C = T + G.
Wir haben den Mechanismus
im letzten Artikel angesprochen, daher werde ich hier nicht weiter darauf eingehen.
Langsam näherten wir uns zwei legendären Menschen, die die Struktur der DNA entdeckten. Francis Crick und James Watson trafen sich 1951 zum ersten Mal. Watson beschloss dann, die Struktur der DNA in Angriff zu nehmen. Als Biologe verstand er, dass man bei der Auswahl einer bestimmten DNA-Struktur die Existenz eines einfachen Prinzips der Verdoppelung des in seine Struktur eingebetteten DNA-Moleküls berücksichtigen muss. In der Tat ist eine der wichtigsten Eigenschaften von Genen die Übertragung von Erbinformationen.
Crick entwickelte die Theorie der Röntgenbeugung durch Spiralen, mit der festgestellt werden kann, ob sich die untersuchte Struktur in einer Spiralkonformation befindet oder nicht. Zu diesem Zeitpunkt gab es bereits Röntgenaufnahmen von DNA. Sie wurden in London von Maurice Wilkins und Rosalind Franklin empfangen.
Durch die Art der Röntgenaufnahme der DNA erkannten Watson und Crick, dass sich die untersuchte Struktur in einer spiralförmigen Konformation befand. Sie wussten auch, dass ein DNA-Molekül eine lange lineare Polymerkette ist, die aus Nukleotidmonomeren besteht. Das Phosphodesoxyribose-Grundgerüst dieses Polymers ist kontinuierlich und stickstoffhaltige Basen sind an der Seite der Desoxyribose-Reste angebracht. Um die Modelle zu konstruieren, blieb die Frage zu lösen, wie viele Ketten linearen Polymers in einer kompakten Struktur gestapelt waren.
Basierend auf der Röntgenaufnahme der DNA der B-Form schlugen Watson und Crick vor, dass das DNA-Molekül aus zwei linearen Polynukleotidketten mit einem Phosphodesoxyribose-Rückgrat außerhalb des Moleküls und stickstoffhaltigen Basen darin besteht. Was später bestätigt wurde. Es blieb nur die Frage der Anordnung der stickstoffhaltigen Basen der beiden Ketten innerhalb des Bispirals zu lösen.
In Anbetracht möglicher Kombinationen stickstoffhaltiger Basenpaare stellte Watson fest, dass Adenin-Thymin- und Guanin-Cytosin-Paare gleich groß sind und durch Wasserstoffbrücken stabilisiert werden. Die Regeln von Chargaff wurden sofort erklärt: Wenn in einer Doppelhelix-DNA Adenin einer Kette immer mit Thymin einer anderen Kette verbunden ist und Guanin immer mit Cytosin gepaart ist, sollte das Adenin in der DNA immer so viel wie Thymin und Guanin sein - so viel wie viel Cytosin. Es war auch klar, wie die Verdoppelung des DNA-Moleküls erfolgen sollte. Jeder Strang ist komplementär zum anderen, und bei der DNA-Replikation müssen sich die Doppelhelixstränge trennen, und jeder dazu komplementäre Strang sollte auf jedem Polynukleotidstrang vervollständigt werden. Es gab auch mehrere Theorien, aber darüber in einer Woche im nächsten Artikel.
Codierungsinformationen
Wir wissen also, dass DNA ein Informationsträger ist, wir wissen, woraus sie besteht. Wie die Informationen verschlüsselt sind, ist jedoch noch nicht klar.
Lassen Sie uns von der Aufgabe gehen. DNA kodiert für 20 Aminosäuren (wir können sagen, dass 21, aber bisher berühren wir Selenocystenin nicht). Es gibt 4 Optionen für Nukleotide. Das heißt, ein Nukleotid kann 4 Varianten codieren, 2 - 16, 3 - 64. Es ist logisch anzunehmen, dass der Code ein Triplett ist (dh drei Basen codieren eine Aminosäure). Über experimentelle Bestätigung können Sie
hier lesen. Ich fürchte, es gibt schon viel Geschichte ...
Tatsächlich haben wir 64 Varianten und 20 Aminosäuren. Aminosäuren können von verschiedenen Codons codiert werden. Es gibt auch Start- und Stoppcodons, von denen aus das Lesen beginnt.
Vergessen Sie nicht, dass zuerst die DNA in RNA eingelesen wird, mit der sie bereits in das Protein einliest.
Die folgende Tabelle zeigt die Entsprechung von RNA-Codons zu Aminosäuren. Denken Sie daran, dass die RNA kein Thymin enthält, sondern stattdessen Uracil verwendet wird.

Wenn Sie das Startcodon in der Tabelle nicht gefunden haben, suchen Sie nach AUG. Es codiert Methionin und ist auch das Ausgangsprodukt. Bei der Translation der Gene von Prokaryoten-, Plastiden- und Mitochondriengenen ist die Ausgangsaminosäure N-Formylmethionin (dies dient nur als Referenz).
Wenn Sie den ganzen Weg von der DNA bis zum Protein malen, bekommen wir so etwas.
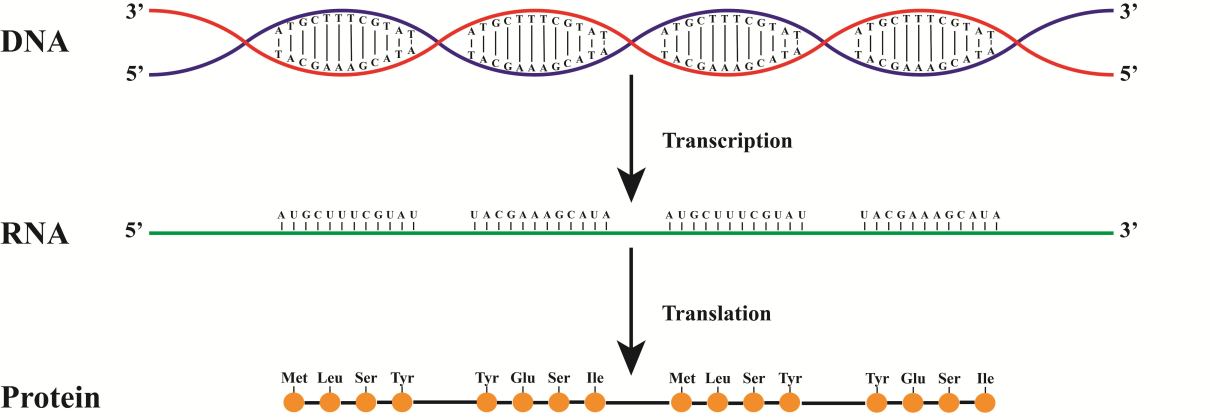
In dieser Figur stammt die Synthese aus der roten Kette. Infolgedessen fällt die RNA mit der blauen Kette zusammen (vergessen Sie nicht, T durch Y zu ersetzen).
Wie gesagt, mehrere Codons können jede Aminosäure codieren. Auf den ersten Blick scheint dies kein besonders notwendiger Nebeneffekt der Codon-Redundanz zu sein. Aber er hat tatsächlich eine ziemlich wichtige Rolle.
Hier werden wir ein wenig auf Mutationen eingehen. Sie kommen in verschiedenen Arten. Von Chromosomen, wenn ganze Chromosomenstücke aus dem Genom entfernt werden, werden sie ausgetauscht, dupliziert, um sie zu zeigen, wenn eine stickstoffhaltige Base durch eine andere ersetzt wird. Konzentrieren Sie sich auf Punktmutationen.
Was können Punktmutationen bewirken?
Das Codon kann beginnen, eine andere Aminosäure zu codieren, was nicht immer beängstigend ist. Solche Mutationen werden Missense mutatsimi genannt (dh mit einer Änderung der Bedeutung). Dies kann die Struktur des Proteins beeinflussen.
Wenn beispielsweise eine positiv geladene Aminosäure durch eine negativ geladene ersetzt wird, kann dies das Protein instabil machen oder dazu führen, dass es sich in eine andere Konformation faltet (ja, die lineare Sequenz von Aminosäuren faltet sich normalerweise in eine bestimmte Form) und kann seine Funktionen nicht erfüllen (oder beginnt dies zu tun) besser, es riecht schon nach Evolution).Insbesondere hat Hämoglobin S eine einzelne Nucleotidänderung (A zu T) im codierenden Gen. Infolgedessen wird das für Glutamat kodierende GAG-Triplett durch das für Valin kodierende THG ersetzt. Hämoglobin S kann auch Sauerstoff transportieren, macht es aber schlimmer als normales Hämoglobin.Im Hikari-Hämoglobinmolekül wird Lysin durch Asparagin ersetzt, es ist jedoch immer noch gut, Sauerstoff zu übertragen.Betrachten Sie als Beispiel für Funktionsverlust Hämoglobin M. Eine weitere Punktmutation im Hämoglobin-Gen führt zu einem vollständigen Funktionsverlust (Histidin ändert sich im aktiven Zentrum zu Tyrosin).Übrigens sieht die Proteinfaltung so aus, wenn Sie alle Nuancen weglassen. Was könnte sonst noch passieren?Das Ersetzen einer stickstoffhaltigen Base kann auch zum Auftreten eines Stoppcodons in der Mitte der Sequenz führen, oder umgekehrt, das Stoppcodon am Ende verschwindet. Der Ausgang ist entweder eine unvollständige Schaltung oder eine extrem lange Schaltung, die in jedem Fall nicht normal funktionieren kann. Solche Mutationen nennt man Unsinn.
Was könnte sonst noch passieren?Das Ersetzen einer stickstoffhaltigen Base kann auch zum Auftreten eines Stoppcodons in der Mitte der Sequenz führen, oder umgekehrt, das Stoppcodon am Ende verschwindet. Der Ausgang ist entweder eine unvollständige Schaltung oder eine extrem lange Schaltung, die in jedem Fall nicht normal funktionieren kann. Solche Mutationen nennt man Unsinn.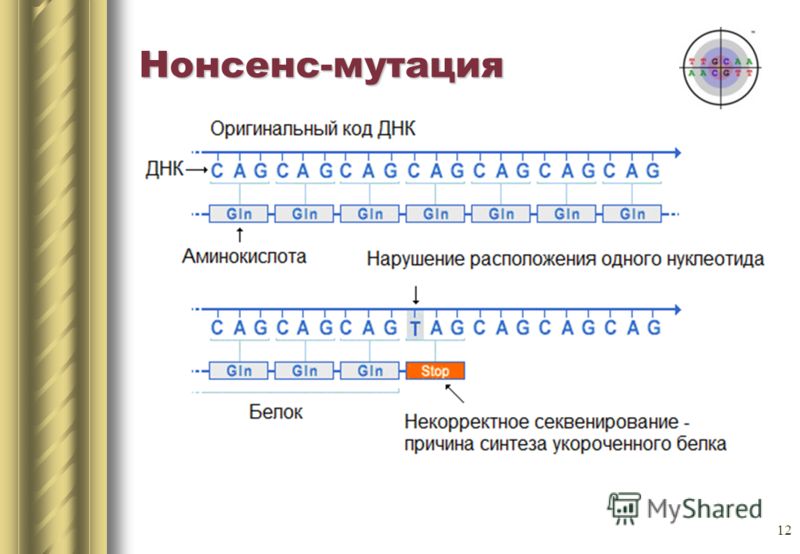 Es gibt eine dritte Art von Mutation - die Mutation zum Schweigen zu bringen. Tatsächlich wird das Codon durch ein anderes Coding für dieselbe Aminosäure ersetzt. Die Eigenschaften des Proteins ändern sich nicht.Um das allgemeine Schema zusammenzufassen.
Es gibt eine dritte Art von Mutation - die Mutation zum Schweigen zu bringen. Tatsächlich wird das Codon durch ein anderes Coding für dieselbe Aminosäure ersetzt. Die Eigenschaften des Proteins ändern sich nicht.Um das allgemeine Schema zusammenzufassen.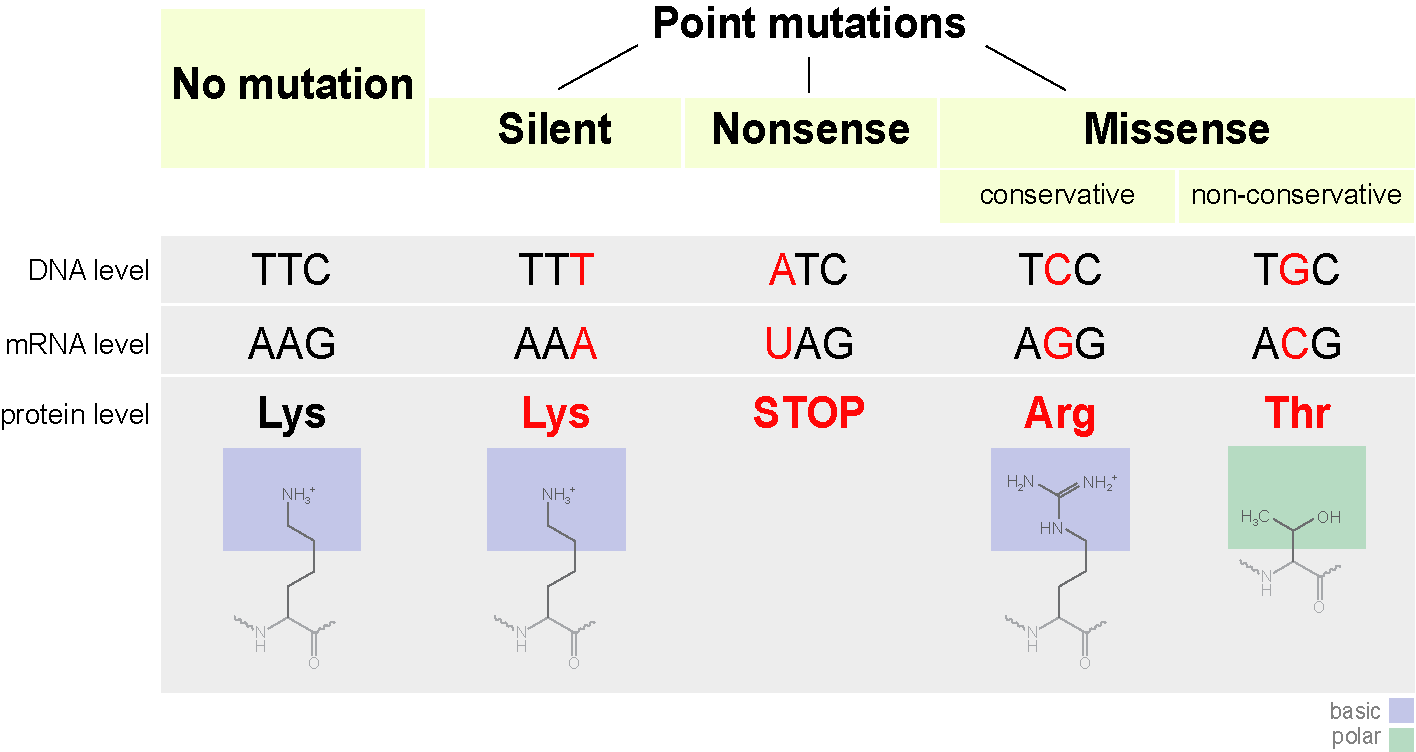 Abschließend möchte ich noch auf ein interessantes Feature eingehen. Eine einzelne Aminosäure kann von mehreren Codons codiert werden. Das wissen wir. Aber was heißt das? Der Körper verwendet alle Codons gleichzeitig zum Codieren. Aber manche öfter, manche weniger.Vergleichen Sie Menschen und ... E. coli ( Escherichia coli ) in der Häufigkeit der Verwendung von Cystein-kodierenden Codons.Es wird von zwei Codons UGU und UGC codiert.HumanesUGU 10.6UGC 12.6Escherichia coli (Stamm O127: H6)UGU 19.1UGC 0.0Stellen ist das Auftreten eines Tripletts pro Tausend. Es ist ersichtlich, dass wir beide Codons mit ungefähr der gleichen Häufigkeit verwenden, während E. coli fast kein UGC-Codon verwendet.Diese Funktion muss beachtet werden, insbesondere wenn Sie in der Gentechnik tätig sind und das Genprodukt eines Organismus in einem anderen produzieren möchten. Wenn Sie versuchen, ein menschliches Gen mit dem häufigen Auftreten des UGC-Codons in die E. coli dieses Stammes einzufügen, werden Sie enttäuscht sein. In einer Zelle sind Aminosäuren mit Transport-RNAs assoziiert, von denen jede ihrem eigenen Codon entspricht. Die dem UGC-Codon entsprechende tRNA ist also extrem klein, was die Synthese stark verlangsamt.Bei Interesse können Sie hier die Unterschiede in der Codonzusammensetzung verschiedener Organismen sehen.Die Codonzusammensetzung kann zwischen Organismen verschiedener Arten und verschiedenen Stämmen stark variieren. So Escherichia coliO157: H7 EDL933 ist auch in Bezug auf UGC und UGU zunehmend weniger. Oder hier ist ein anderes Beispiel. Die in verschiedenen Ländern isolierten Tuberkelbazillusstämme weisen ebenfalls eine unterschiedliche Codezusammensetzung auf .
Abschließend möchte ich noch auf ein interessantes Feature eingehen. Eine einzelne Aminosäure kann von mehreren Codons codiert werden. Das wissen wir. Aber was heißt das? Der Körper verwendet alle Codons gleichzeitig zum Codieren. Aber manche öfter, manche weniger.Vergleichen Sie Menschen und ... E. coli ( Escherichia coli ) in der Häufigkeit der Verwendung von Cystein-kodierenden Codons.Es wird von zwei Codons UGU und UGC codiert.HumanesUGU 10.6UGC 12.6Escherichia coli (Stamm O127: H6)UGU 19.1UGC 0.0Stellen ist das Auftreten eines Tripletts pro Tausend. Es ist ersichtlich, dass wir beide Codons mit ungefähr der gleichen Häufigkeit verwenden, während E. coli fast kein UGC-Codon verwendet.Diese Funktion muss beachtet werden, insbesondere wenn Sie in der Gentechnik tätig sind und das Genprodukt eines Organismus in einem anderen produzieren möchten. Wenn Sie versuchen, ein menschliches Gen mit dem häufigen Auftreten des UGC-Codons in die E. coli dieses Stammes einzufügen, werden Sie enttäuscht sein. In einer Zelle sind Aminosäuren mit Transport-RNAs assoziiert, von denen jede ihrem eigenen Codon entspricht. Die dem UGC-Codon entsprechende tRNA ist also extrem klein, was die Synthese stark verlangsamt.Bei Interesse können Sie hier die Unterschiede in der Codonzusammensetzung verschiedener Organismen sehen.Die Codonzusammensetzung kann zwischen Organismen verschiedener Arten und verschiedenen Stämmen stark variieren. So Escherichia coliO157: H7 EDL933 ist auch in Bezug auf UGC und UGU zunehmend weniger. Oder hier ist ein anderes Beispiel. Die in verschiedenen Ländern isolierten Tuberkelbazillusstämme weisen ebenfalls eine unterschiedliche Codezusammensetzung auf .Zusammenfassend
Diesmal gab es viel Geschichte und relativ wenig Biologie. Dies wird nicht wieder vorkommen. Wir haben darüber gesprochen, wie klar wurde, dass DNA ein Informationsträger ist, wie sie in der DNA selbst gespeichert ist. Wir haben über die Redundanz des Gencodes gesprochen und darüber, wozu er führt. Mutationen und der Unterschied in der Häufigkeit der Verwendung bestimmter Codons waren leicht betroffen.Nächstes Mal werden wir über die DNA-Replikation sprechen.PS: Es wird auch Geschichte geben, aber viel weniger. Ich werde versuchen, solche Pausen nicht mehr schriftlich zu machen.