
Wissenschaftler führten eine Party in Tic-Tac-Toe mit DNA durch. Klingt lustig, aber dies ist nur die Spitze des Eisbergs, erschüttert von einem Hauch von Humor der Wissenschaftler selbst. Tatsächlich enthüllt die heutige Studie neue Methoden zur Überwachung der Kinetik der Wechselwirkung von DNA-Nanostrukturen, die zusammen komplexe Aufgaben erfüllen können. Die Verwendung von DNA als Hauptbestandteil des Systems ist keine leichte Aufgabe, aber dies hält Wissenschaftler nicht davon ab, deren Geist voller Ideen ist, sondern Herzen voller Begeisterung. Wie haben sie es also geschafft, die DNA-Blöcke nach einem bestimmten Muster zu bewegen, wie die Studie gezeigt hat, und wie sehen die Aussichten für diese Technik in der Zukunft aus? Für Antworten wenden wir uns dem Bericht der Forschungsgruppe zu. Lass uns gehen.
StudienbasisIn dieser Studie demonstrierten Wissenschaftler eine neue Technik zum Bewegen von DNA-Kacheln, die auf den Prinzipien von Stützgelenken und Verzweigungsversätzen basiert. Diese Prinzipien ähneln der Standardverschiebung von DNA-Strängen, zielen jedoch auf größere Objekte ab, genauer gesagt auf DNA-Strukturen.
Diese Technik ermöglichte es, die Kinetik von DNA-Kacheln zu kontrollieren und die gewünschte Reaktionskette in Mehrstruktursystemen zu realisieren. Das Tic-Tac-Toe-Spiel wurde nicht zum Spaß durchgeführt, sondern um die Möglichkeit zu demonstrieren, die DNA der Kachel in beliebiger Reihenfolge und Position zu verschieben.
Um die Mechanismen biologischer Systeme zu verstehen, müssen ihre Hauptkomponenten betrachtet werden, unter denen molekulare Strukturen eine der wichtigsten sind. DNA als Trägermolekül gilt als eines der besten Materialien für die Untersuchung und Schaffung selbstorganisierter Nanostrukturen.
Unter den auf DNA basierenden Techniken hat die DNA-Origami-Technik in den letzten Jahren immer mehr an Beliebtheit gewonnen, sodass Sie Strukturen aus DNA jeder gewünschten Form und Konfiguration erstellen können.
Die Komponenten eines einzelnen DNA-Origamis können nach Belieben gedreht und verdreht werden. Bisher war es jedoch nicht möglich, eine kontrollierte Wechselwirkung zwischen mehreren DNA-Origamis als Komponenten einer größeren und daher komplexeren Struktur zu erreichen. Im Moment war es nur möglich, mehrere DNA-Origami zu verbinden / zu trennen.
In der heutigen Studie haben Wissenschaftler eine neue DNA-Origami-Interaktionstechnik demonstriert. Es gibt eine Struktur, in die eine DNA-Origami-Kachel eingebettet ist, die eine andere Kachel aus dem allgemeinen Array verdrängt und aufgrund der
Bindungsdomäne * am Rand der Kachel (Drehpunkt / Verbindungspunkt) mit der Struktur verbunden ist.
Bindungs- / Bindungsdomäne * - eine Proteindomäne, die Teil der Proteinkette ist, aber getrennt davon funktionieren kann.
Als Beispiel haben Wissenschaftler drei Rekonfigurationsoptionen erstellt: wettbewerbsfähig, sequentiell und kooperativ. Die Gesamtheit dieser „Bewegungen“ (schrittweise Rekonfiguration) wurde als Tic-Tac-Toe-Spiel demonstriert, bei dem jeder Spieler 9 einzigartige DNA-Origami hatte, die in beliebiger Reihenfolge auf einem Feld von 264 x 264 nm bewegt werden können.
DNA-Fliesenbildung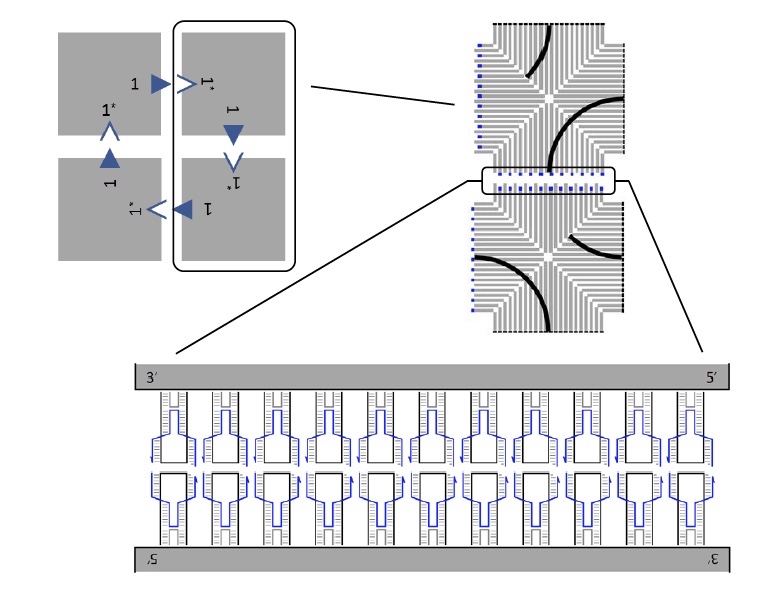 DNA-Kachelstrukturen.
DNA-Kachelstrukturen.Zuvor entwickelten die Wissenschaftler vor Durchführung dieser Studie eine Technik zur Erstellung einzelner DNA-Kacheln. Jede Kachel an zwei Kanten hat 11 Heftklammern, von denen jede am Stapeln beteiligt ist.
Das Bild oben zeigt eine Anordnung von 4 Kacheln (oben links), von denen jede über Klammern mit der benachbarten verbunden ist.
Die Forscher erwogen mehrere mögliche Optionen zum Erstellen größerer Arrays mit einer großen Anzahl von Kacheln. Dafür war es theoretisch notwendig, dass die Verbindung zwischen den Fliesen schwach genug war. Dies würde es ihr ermöglichen, das Array zu brechen und neu aufzubauen. Beobachtungen zeigten jedoch, dass es möglich ist, rekonfigurierbare Arrays mit mehreren Kacheln zu erstellen, selbst bei Temperaturen, bei denen die Verbindung zwischen den Kacheln dauerhaft sein sollte.
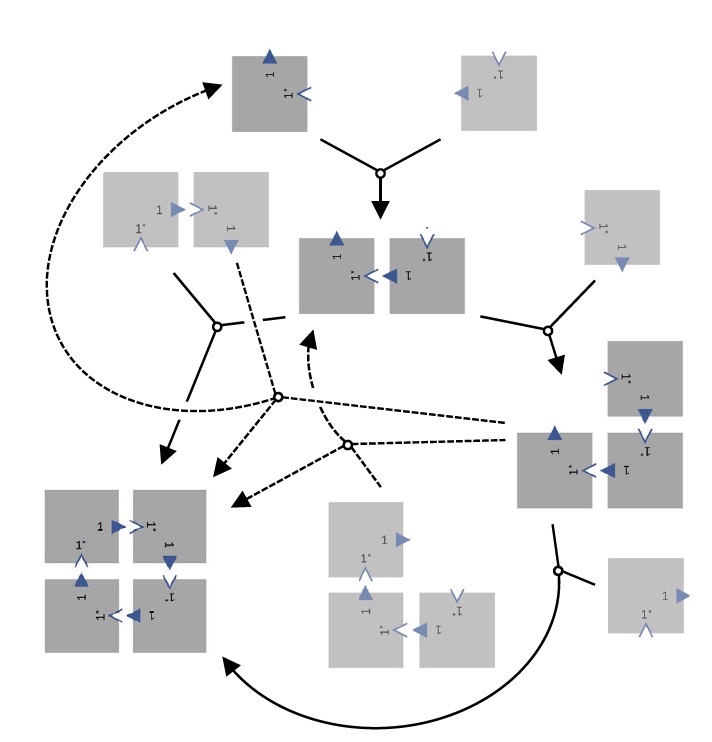 Neukonfiguration von Kacheln.
Neukonfiguration von Kacheln.Dieses Phänomen hat zwei mögliche Erklärungen. Erstens tritt die Bindungsbildung bei einer ausreichend hohen Temperatur auf, während sie in den Temperaturbereich der Prozessreversibilität fällt. Das zweite - das Dimer und das Trimer gehen eine Verdrängungsreaktion ein, daher wird ein 2 × 2-Array gebildet, wenn das Monomer freigesetzt wird. Es können auch 2 Kopien von Trimeren hergestellt werden, die ein 2 × 2-Array bilden, das ein Dimer freisetzt.
Wenn während der Verschiebungsreaktionen keine spontane Trennung der Fliesen auftritt, gibt es keine kinetischen Fallen, aufgrund derer die Fliesen in einer 2x2-Konfiguration gesammelt werden.
Es musste überprüft werden, ob ein DNA-Origami einen anderen in der allgemeinen Struktur verdrängen würde, ohne sich spontan innerhalb der Struktur selbst zu binden. Mit anderen Worten, ob der DNA-Origami Initiative zeigt oder nicht. Hierzu wurden zwei Versuche durchgeführt.
 Praktische Erfahrung: 2 DNA-Kacheln (Quadrate) + 1 DNA-Dreieck, das eine gemeinsame Struktur bilden sollte.
Praktische Erfahrung: 2 DNA-Kacheln (Quadrate) + 1 DNA-Dreieck, das eine gemeinsame Struktur bilden sollte.Im ersten Experiment waren 2 DNA-Kacheln und 1 DNA-Dreieck beteiligt, das Experiment selbst wurde bei konstanter Temperatur durchgeführt. Strukturell könnte ein Dreieck entweder dieselbe Verbindungsdomäne wie die Quadrate haben, oder es könnte eine zusätzliche Verbindungsdomäne haben, die das benachbarte Quadrat ergänzt.
In der ersten Version des Experiments bleiben 2 Quadrate miteinander verbunden, während das Dreieck mit einem der Quadrate verbunden ist. In der zweiten Version ersetzte ein Dreieck eines der Quadrate und verschob es sozusagen. Dies hat in der Praxis gezeigt, dass es möglich ist, komplexe DNA-Origami zu modifizieren. Jetzt müssen wir jedoch sicherstellen, dass dieser Prozess von Wissenschaftlern gesteuert wird und nicht aufgrund natürlicher Kräfte von selbst abläuft.
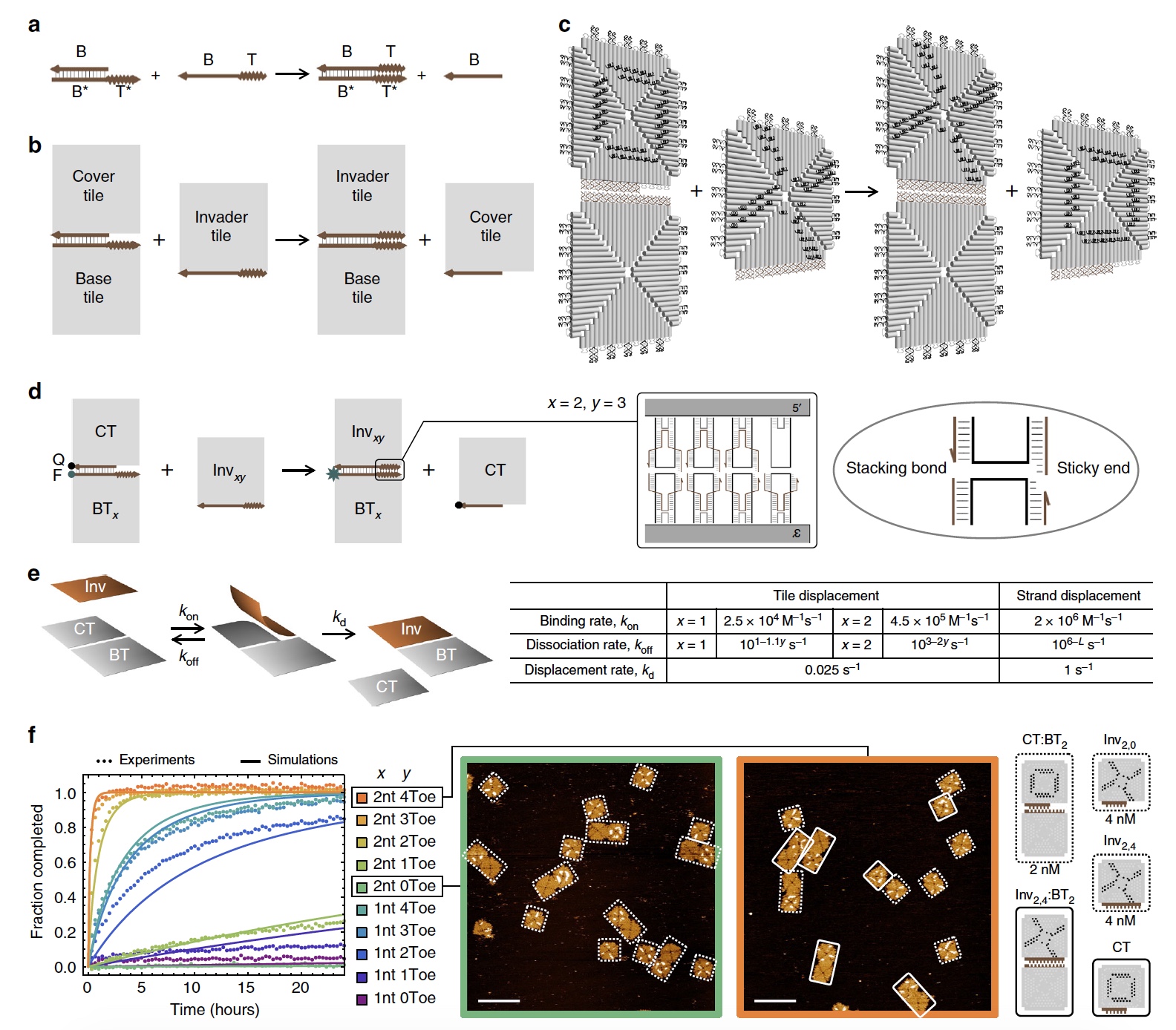 Bild Nr. 1
Bild Nr. 1Wenn wir über Verdrängungsreaktionen in DNA-Ketten sprechen, dann haben wir hier eine Kette mit einer Bindungsdomäne, die mit einer „freien“ komplementären Domäne verbunden ist. So erhalten wir eine doppelsträngige Struktur, in der der Prozess der Verzweigungsmigration stattfindet, wenn sich die Ketten durch den Verbindungspunkt (
1a ) bewegen. Ein ähnlicher Prozess tritt bei den DNA-Verdrängungsreaktionen von Fliesen (
1b und
1c ) auf.
Aus Bild
1c können wir auch erkennen, dass jede DNA-Kachel aus 4 gleichschenkligen Dreiecken besteht, die durch geschweifte Klammern verbunden sind, über die wir bereits Bescheid wissen.
In Bezug auf das Tic-Tac-Toe-Spiel: Zur bequemeren Visualisierung wurde der Prozess der Verschiebung und Rekonfiguration der Teilnehmer mit einem Kreuz (X) und einem Zeh (O) mittels Klammern in zwei verschiedenen Farben markiert. Schauen Sie sich Bild Nr. 1 genauer an. X (schwarz) und O (weiß) sind auf den Kacheln sichtbar.
Kinetik von Fliesen-DNA-VerdrängungsreaktionenMit einfachen Worten, um die Kontrolle über die Kinetik eines Prozesses zu erlangen, müssen einige Reaktionen schneller und andere langsamer werden. Auf diese Weise kann das Verbinden, Trennen und Neukonfigurieren der Kacheln erreicht werden. Sie müssen jedoch den Bereich der Kinetik bestimmen, der dies ermöglicht. Zu diesem Zweck haben Wissenschaftler Verbindungspunkte (zwischen Kacheln) mit sozusagen unterschiedlichen Kopplungsstärken erstellt.
Die Anzahl der Heftklammern auf dem Connector in der Zweigmigrationsdomäne war für alle Kacheln gleich. Aber hier variierte die Domänendomäne der Referenzpunkte (Bindungspunkte) von 0 bis 4 (
1d ).
Das Experiment umfasste die Hauptkachel (alle 4 Kommunikationspunkte sind „aktiv“) und die Zubehörkacheln (die Anzahl der Verbindungspunkte variierte von 0 bis 4). Am Ende der Zweigmigrationsdomäne wurden 2 Heftklammerpaare mit einem Fluorophor und einem Quencher modifiziert. Wenn die Zubehörkachel in Kontakt mit der Hauptkachel bleibt, wird das Fluorophor gelöscht, was zu einem niedrigen Fluoreszenzsignal führt. Wenn jedoch die Zubehörkachel (mit der Klappe) getrennt wird, wird das Fluoreszenzsignal verstärkt. Somit kann bestimmt werden, wie der Prozess abläuft.
Einen Tag später konnten aus den Beobachtungen erste Schlussfolgerungen gezogen werden. Wissenschaftler stellen fest, dass die fluoreszierenden Trajektorien der Zubehörkacheln mit 0 Verbindungsklammern praktisch unverändert blieben. Es wurde auch festgestellt, dass Fliesen mit der gleichen Länge der Verbindungsrippe, jedoch mit einer großen Anzahl von Verbindungspunkten, eine viel schnellere Kinetik aufweisen. Es ist merkwürdig, dass die Verbindungspunkte mit den Heftklammern mit 1-Nucleotid und 2-Nucleotid sehr unterschiedliche Sättigungsraten (Sättigungsraten) aufweisen.
Bild
1e zeigt das mathematische Modell, das zur numerischen Abschätzung der Kinetik verwendet wird. Wissenschaftler selbst sagen, dass das Modell sehr einfach ist und häufig zur Beurteilung der Kinetik bei DNA-Kettenverschiebungsreaktionen verwendet wird und daher auch für DNA-Kacheln geeignet ist.
Eine vergleichende Analyse von (
1f ) Simulationen (simulierten Reaktionen) und Experimenten ermöglichte es, diejenigen Parameter zu bestimmen, die den Rekombinationsprozess von DNA-Kacheln beeinflussen.
Zunächst stellen Wissenschaftler fest, dass die Geschwindigkeit des Verbindens von Kacheln 10-100-mal (abhängig von der Länge der Kante der Verbindungskachel) geringer ist als die Geschwindigkeit des Verbindens von DNA-Strängen. Eine gemeinsame Sache zwischen den Prozessen der Verschiebung von Fliesen und Fäden ist, dass mit einer Zunahme der Anzahl von Nukleotiden an den Verbindungspunkten die Zerfallsrate exponentiell abnimmt. Gleichzeitig ist diese Geschwindigkeit bei Kacheln 40-mal niedriger als bei DNA-Strängen. Die maximale Gesamtverschiebung für DNA-Kacheln (Reaktionsgeschwindigkeit) betrug 4,5 × 10
5 M
–1 * s
–1 . Wenn die Konzentration niedrig ist (<50 nM), begrenzt die Geschwindigkeit der bimolekularen Bindung die Geschwindigkeit der Verschiebung der Fliesen. Wenn die Konzentration mehr als 50 Nanomol beträgt, begrenzt die Geschwindigkeit der monomolekularen Verschiebung die Verschiebungsrate der Fliesen.
Darüber hinaus führten die Wissenschaftler mehrere Experimente durch, in denen drei Haupttypen der Rekonfiguration beschrieben wurden: kompetitiv, sequentiell und kooperativ. Und jetzt schauen wir uns die Ergebnisse der Experimente für jedes von ihnen genauer an.
Wettbewerbsfähige RekonfigurationUm eine wettbewerbsfähige Rekonfiguration zu erreichen, schlugen die Wissenschaftler vor, eine Sigmoidfunktion als Reaktion auf die Signalkonzentration zu verwenden, um so ein auf Informationen basierendes Rekonfigurationssystem zu erhalten. Diese Funktion ist ein wichtiges Element der digitalen Logikberechnung von DNA-Strangverdrängungsprozessen und kann daher auch auf DNA-Kacheln angewendet werden.
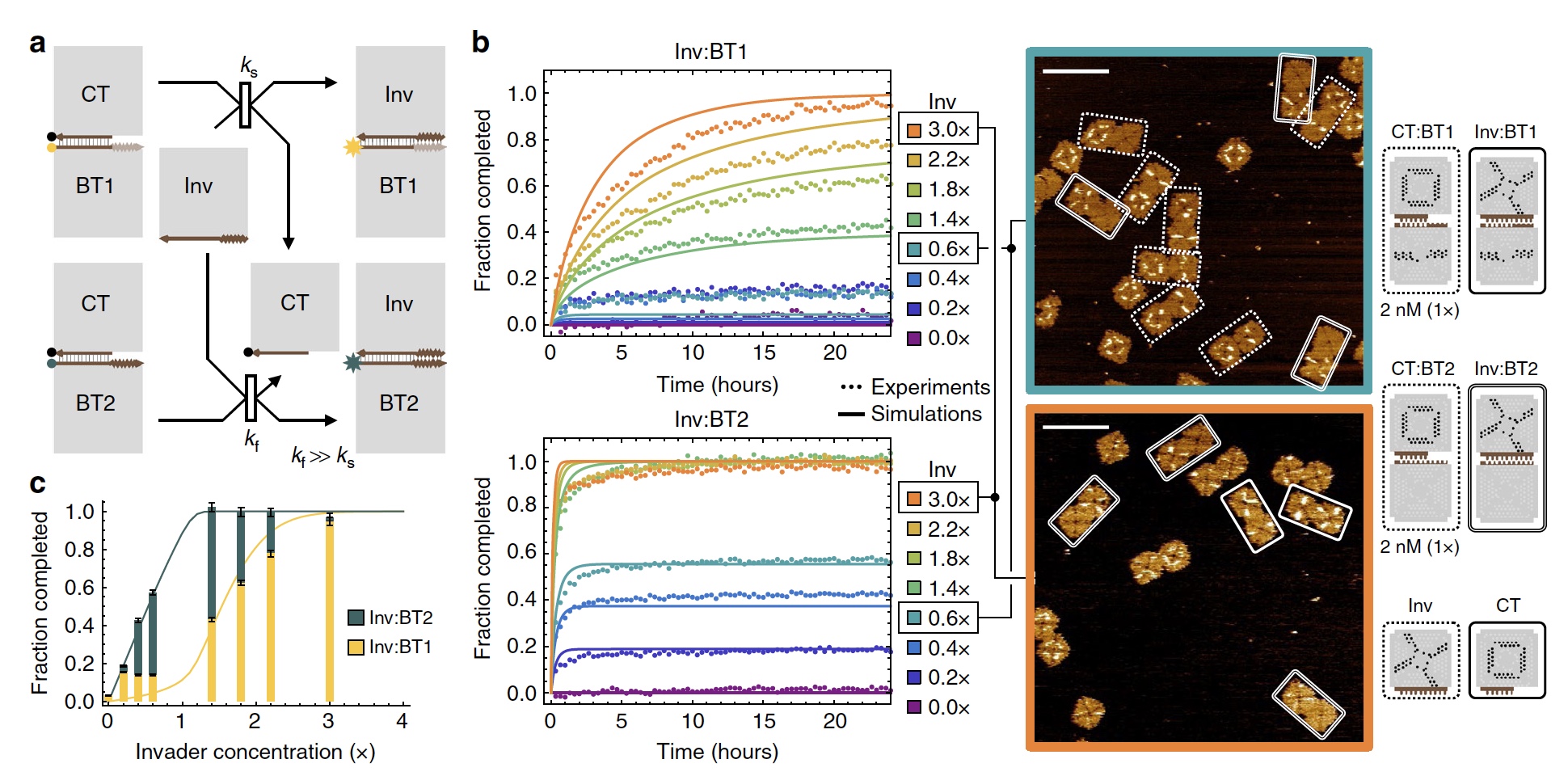 Bild Nr. 2
Bild Nr. 2Um die „Arbeit“ der Sigmoidfunktion zu demonstrieren, haben Wissenschaftler zwei konkurrierende Kachelverschiebungsreaktionen erstellt, die von derselben untergeordneten Kachel aktiviert werden. Der Unterschied in ihrer Geschwindigkeit (
2a ).
Bei Zubehörkacheln besteht der Verbindungspunkt aus vier 2-Nucleotid-Heftklammern. Es wird erwartet, dass es sich mit einem geeigneten Verbindungspunkt und mit vier 1-Nucleotid-Klammern verbindet. Die Reaktionsgeschwindigkeit der Zubehörkacheln unterscheidet sich um das 18-fache. Wenn die Konzentration der Zubehörkacheln weniger als 2 nM betrug, wurde eine schnellere Reaktion (
2b ) ausgelöst. Wenn die Konzentration größer als 2 nM war, wurde eine langsamere Reaktion gestartet.
Nach 24 Stunden wurde festgestellt, dass das Ergebnis einer schnelleren Reaktion linear anstieg und das Ergebnis einer langsameren Reaktion eine Sigmoidfunktion zeigte (
2c ).
Sequentielle RekonfigurationNach dem Wettbewerb war die Neukonfiguration konsistent. Dieser Typ ist viel komplizierter, bietet aber mehr Kontrolle über den Prozess, es sei denn, natürlich funktioniert alles.
Daher haben die Wissenschaftler ein 2x2-Array erstellt, in dem die erste Unterkachel eine andere Kachel verschiebt und den zuvor verwendeten Verbindungspunkt freigibt. Als nächstes verschiebt die zweite untergeordnete Struktur von 2 Kacheln 2 Kacheln aus dem Array. Eine Art Kaskadenverschiebung (
3a ).
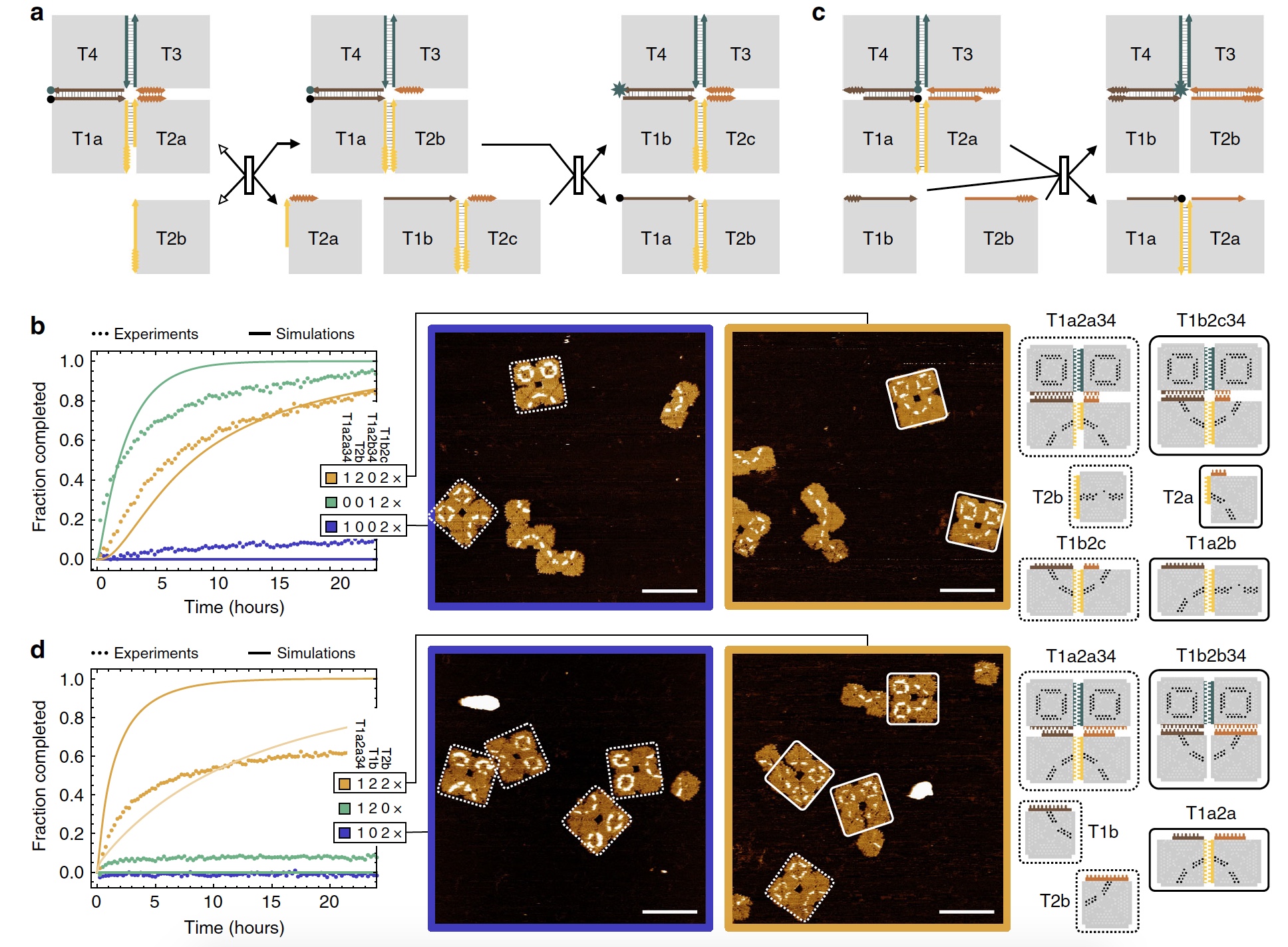 Bild Nr. 3
Bild Nr. 3Wissenschaftler haben festgestellt, dass die Verschiebung an den Ecken der Fliesen langsam ist, etwa 100-mal langsamer als am Rest der Fliesenkante. Daher müssen solche Zwischenverschiebungszustände für komplexere Rekonfigurationsverfahren berücksichtigt werden.
In Bild
3b sehen wir recht erfolgreiche Ergebnisse des Experiments mit sequentieller Rekonfiguration (orange Kurve).
Und jetzt gibt es etwas mehr Kreativität von Forschern. Achten Sie auf die obigen Bilder (
3b ): Arrays, die auf die erste untergeordnete Kachel, aber nicht auf die zweite reagierten, sehen am Ende wie ein „trauriges Gesicht“ aus. Aber die Arrays, in denen die Verbindung mit beiden untergeordneten Kacheln erfolgreich war, sehen aus wie ein „lächelndes Gesicht“. Und Sie sagen, Wissenschaftler haben keinen Sinn für Humor.
Laut Wissenschaftlern ist der Rekonfigurationsprozess selbst umso komplizierter, je komplexer die Struktur der Reaktionsteilnehmer ist, aber desto interessanter ist das Ergebnis. Die Verwendung von zwei Arten von Teilnehmern (2 × 2-Array und Zwei-Kacheln-Struktur) zeigte die folgenden Ergebnisse zur Verschiebungseffizienz nach 48 Stunden Beobachtung: 83,3 ± 9,8% und 90,5 ± 6,1%. Mit anderen Worten, Verdrängungsreaktionen sind ausgezeichnet. Es gibt einige Fehler, aber sie sind unbedeutend.
Die Experimente mit sequentieller Rekonfiguration ermöglichten nicht nur das Verständnis der Grundprinzipien der Sequenz von Kommunikationspunkten und der "Kaskade" der Reaktion, sondern zeigten auch neue Merkmale. Das System kann auf mehr als ein Signal reagieren, das angibt, welche Anweisungen ausgeführt werden müssen und was dafür verfügbar ist. Dieses Signal wird im Experiment in Form von zwei Arten von untergeordneten Strukturen dargestellt. Selbst wenn die erste untergeordnete Struktur früher als die zweite mit der Basis in Kontakt kommt, zeigt die Struktur immer noch die erwartete Rekonfiguration.
Diese Prozesse laufen größtenteils von selbst ab. Wissenschaftler kontrollieren sie nur geringfügig, greifen jedoch nicht vollständig ein. Der nächste Schritt in den Experimenten besteht darin, die Kontrolle über den Rekonfigurationsprozess zu verbessern und die Möglichkeit der Programmierung dieses Prozesses zu untersuchen.
Kooperative RekonfigurationIn diesem Experiment gab es 2 Zubehörkacheln, die mit den beiden Kanten des 2x2-Arrays (
3s ) verbunden waren. Wenn nur eine Kachel vorhanden ist, sollte sie in die Mitte des Arrays verschoben werden. Darüber hinaus können Sie durch das Fehlen einer zweiten Kachel den Vorgang umkehren und die Zubehörkachel wiederholt vom Array trennen. Bei Vorhandensein beider untergeordneter Kacheln treten sofort zwei Prozesse der Verzweigungsmigration auf, die in der Mitte des Arrays konvergieren, und als Ergebnis erhalten wir eine kooperative Verschiebung.
Die Erfahrung mit der Fluoreszenz hat gezeigt, dass bei nur einer untergeordneten Kachel das Fluoreszenzsignal sehr schwach ist, jedoch verstärkt wird, wenn beide Kacheln vorhanden sind (
3d : blaue und grüne Kurve - schwaches Signal, gelb - starkes Signal).
Wie im vorherigen Experiment wurden Arrays, die nur mit einer untergeordneten Kachel interagieren, als "trauriges Gesicht" markiert, und Arrays und beide Kacheln waren "lächelndes Gesicht". Darüber hinaus bildete sich letzteres in 68,0 ± 7,7% der Fälle.
Tic Tac ToeUnd schließlich das „Tic-Tac-Toe“ -Experiment. In diesem Experiment wurde ein 3x3-Array verwendet, das 9 einzigartige Fliesenverschiebungsreaktionen in beliebiger Reihenfolge ermöglicht (
4a ).
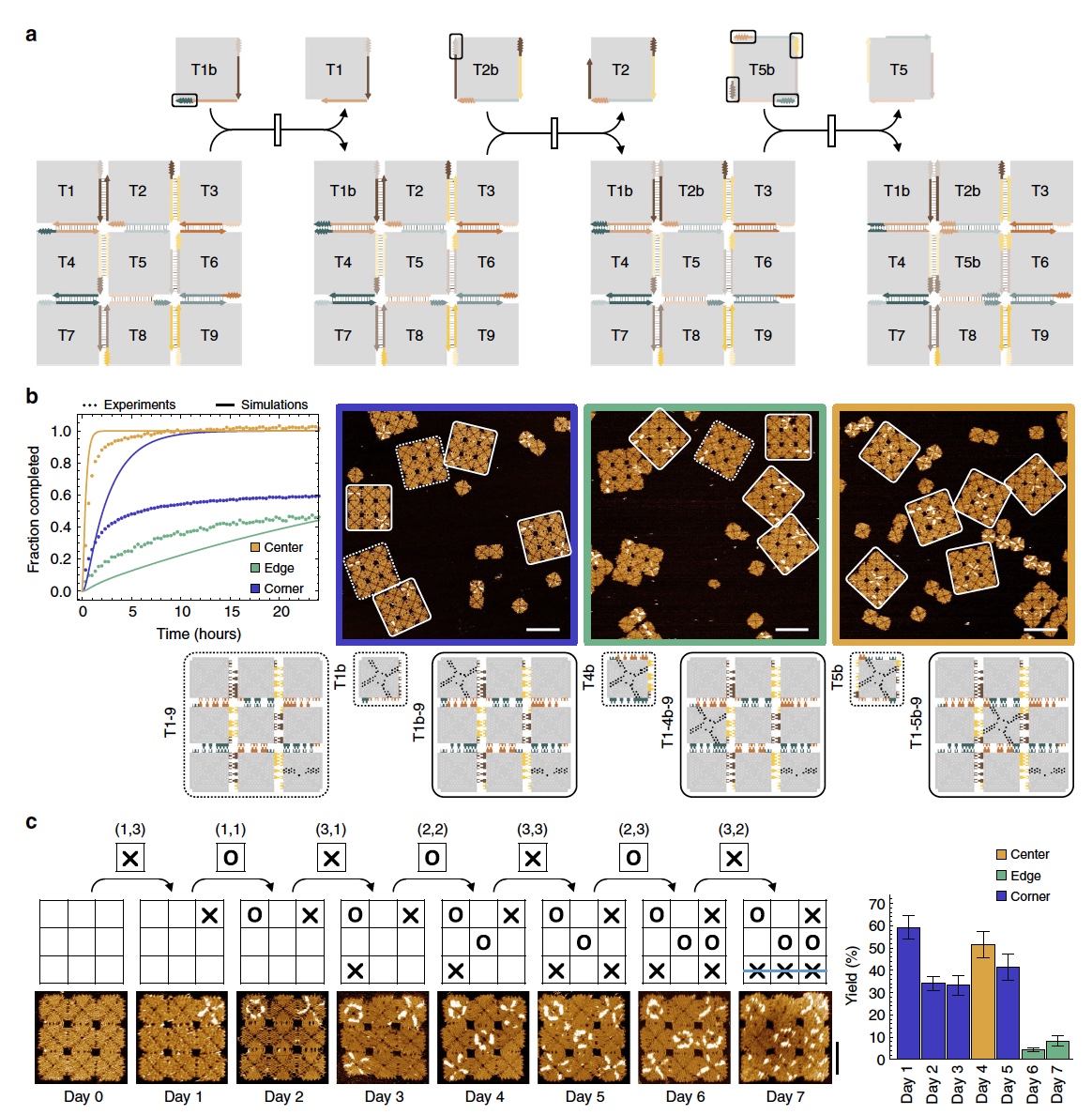 Bild Nr. 4
Bild Nr. 4Dieses Experiment ist eine Kombination mehrerer Arten von Reaktionen gleichzeitig: die Verschiebung der Eckkachel, die Verschiebung der Randkachel und die Verschiebung der zentralen Kachel. Entlang des Umfangs der Anordnung zwischen den benachbarten Kacheln befand sich ein Verbindungspunkt, insgesamt 8 solcher Punkte. 4 von ihnen waren an der Verschiebung der Eckkachel beteiligt, wenn die Zubehörkachel mit dem entsprechenden Verbindungspunkt in Kontakt mit der Randkachel in der Nähe der Ecke steht. In diesem Moment wandert der Zweig innerhalb des Randes der Kachel und um einen Winkel von 90 °, und die zuvor mit dem Array verbundene Eckkachel wird freigegeben und in das Array selbst integriert (innerhalb des Arrays, als es sich zuvor am Rand befand). Die anderen 4 Verbindungspunkte wurden verwendet, um die Verschiebung der Randkacheln zu aktivieren.
Das Schwierigste in diesem Experiment war es, aufgrund der begrenzten Anzahl verfügbarer Kommunikationspunkte eine Verschiebung der zentralen Kachel einzuleiten. Daher wurden 4 zusätzliche Punkte zwischen der zentralen Kachel und allen benachbarten Kacheln hinzugefügt (
4a ).
Die Beobachtungen zeigten hervorragende Ergebnisse. Alle Unterkacheln wurden in das Array eingebettet, das wie von Wissenschaftlern geplant wieder aufgebaut wurde. Der Umwandlungsprozentsatz innerhalb des Arrays war wie folgt: 78,4 ± 6,0% für die Verschiebung von Eckkacheln, 52,8 ± 6,0% für die Verschiebung von Randkacheln und 100% der zentralen Kacheln.
Und jetzt wenden wir uns direkt dem Spiel selbst zu, das alle Arten von Reaktionen in Form einer Kaskade kombiniert. Das Spielbrett war ein 3x3-Array, alle Kacheln waren „sauber“, das heißt, sie waren in keiner Weise markiert.
Jeder der Spieler, von denen es natürlich 2 gab, erhielt 9 untergeordnete Plättchen mit den Markierungen „X“ und „O“. Wenn Sie in diesem Spiel einen Zug machen, wird dieses Plättchen im Abstand von 24 Stunden zur Testkammer hinzugefügt (eine lange Charge Tic-Tac-Toe, nicht wahr?).
Videodemonstration des Spielens von Tic-Tac-Toe mit DNA-Kacheln.Beobachtungen (
4b und
4c ) zeigten, dass das Spielfeld auf alle Züge reagierte, dh die Neukonfiguration erfolgte gemäß den Anweisungen der Spieler (Hinzufügen markierter Kacheln).
Das ganze Problem war, dass am Ende des Spiels auf dem Spielfeld viele zusätzliche Kacheln vorhanden waren und die Wahrscheinlichkeit ihrer spontanen Verschiebung zunahm. Somit wurde auch die Genauigkeit der Rekonfiguration verringert. Am Ende des Spiels betrug dieser Indikator nur 8,3 ± 2,3%. Das Ergebnis wurde jedoch immer noch erzielt, wie wir aus den obigen Bildern sehen können.
Für eine detailliertere Kenntnis der Nuancen von Forschung und Experimenten empfehle ich dringend, dass Sie sich den
Bericht von Wissenschaftlern und
zusätzliche Materialien dazu ansehen.
NachwortDas Spiel der Tic-Tac-Toe-DNA-Kacheln wurde nicht zum Spaß durchgeführt, sondern als Methode zur Demonstration einer sehr ernsthaften Studie, die die Fähigkeit aufzeigt, die Prozesse der Verschiebung von Elementen in aus DNA gebauten Nanostrukturen zu manipulieren.
Natürlich bleibt noch viel zu polieren: Die Anzahl der Zubehörkacheln, die die endgültige Rekonfigurationsrate beeinflussen, die Reaktionsgeschwindigkeit, die Genauigkeit der Verschiebung, die Erweiterung möglicher Verschiebungsoptionen und natürlich die Vergrößerung der Dimensionen der rekonfigurierbaren DNA-Struktur.
Jetzt widmen viele Wissenschaftler den nicht standardmäßigen und nicht trivialen Mechanismen zur Durchführung von Prozessen, die bereits zum Standard geworden sind, immer mehr Aufmerksamkeit. Die Speicherung und Verarbeitung von Informationen, die Diagnose und Behandlung von Krankheiten, die Untersuchung komplexer Systeme, einschließlich biologischer Systeme - all diese Prozesse können mithilfe von DNA durchgeführt werden, da dieses unglaublich kleine Objekt unglaubliche Eigenschaften und Merkmale verbirgt, die wir noch nicht enthüllt haben.Übrigens stellt sich heraus, dass Sie dieses Spiel spielen können, wenn Sie die Abfrage "Tic-Tac-Toe" bei Google eingeben. :) (Ich weiß nicht wie jemand, aber ich habe das noch nie bemerkt)Und natürlich Freitag offtopic (ich werde das Neujahrsthema fortsetzen, wenn Sie nichts dagegen haben):
Ein bisschen süß unter dem Baum :)
Vielen Dank für Ihre Aufmerksamkeit, bleiben Sie neugierig und haben Sie ein schönes Wochenende, Jungs.
Vielen Dank für Ihren Aufenthalt bei uns. Gefällt dir unser Artikel? Möchten Sie weitere interessante Materialien sehen? Unterstützen Sie uns, indem Sie eine Bestellung
aufgeben oder Ihren Freunden empfehlen, einen
Rabatt von 30% für Habr-Benutzer auf ein einzigartiges Analogon von Einstiegsservern, das wir für Sie erfunden haben: Die ganze Wahrheit über VPS (KVM) E5-2650 v4 (6 Kerne) 10 GB DDR4 240 GB SSD 1 Gbit / s von $ 20 oder wie teilt man den Server? (Optionen sind mit RAID1 und RAID10, bis zu 24 Kernen und bis zu 40 GB DDR4 verfügbar).
VPS (KVM) E5-2650 v4 (6 Cores) 10GB DDR4 240GB SSD 1Gbps ,
.
Dell R730xd 2 mal günstiger? 2 Intel Dodeca-Core Xeon E5-2650v4 128GB DDR4 6x480GB SSD 1Gbps 100 $249 ! Lesen Sie mehr über
den Aufbau eines Infrastrukturgebäudes. Klasse mit Dell R730xd E5-2650 v4 Servern für 9.000 Euro für einen Cent?