Wer sammelte in der Kindheit nicht gern Konstrukteure? Ich erinnere mich noch an diese rote Schachtel mit einer Reihe von Metallteilen, Werkzeugen und einem Meer möglicher Ergebnisse, wenn es nur Fantasie, Zeit und Begierde gäbe. LEGO sollte auch nicht vergessen werden, obwohl hier alles etwas einfacher und viel bunter war. Was jedoch komplizierter ist, sind die Entwickler von Nanostrukturen, die auf DNA-Origami basieren. Bisher wurden alle "Details" solcher Strukturen manuell modelliert, was viel Zeit und Mühe in Anspruch nahm. Stellen Sie sich vor, Sie müssen alle LEGO-Teile selbst erstellen, bevor Sie sie zu einem riesigen Roboter mit Lasern, Düsentriebwerken und einem Maschinengewehr auf Ihrer Schulter zusammenbauen. Aber etwas Kindheitserinnerungen führten uns in die falsche Steppe.
Witze beiseite. Heute lernen wir den Algorithmus kennen, mit dem Sie automatisch DNA-Origami in einer ziemlich unterschiedlichen Form erstellen können. Zuvor wurde die Form der DNA-Stränge manuell auf das Notwendige geändert, was die Möglichkeiten eines solchen Verfahrens stark einschränkte. Mit demselben Algorithmus können Sie automatisch Designer-DNA-Teile erstellen, mit denen Sie zweidimensionale und dreidimensionale Nanostrukturen bilden können. Außerdem steht dieser Algorithmus allen zur Verfügung. Ein Bericht des Forschungsteams hilft uns herauszufinden, was wie funktioniert. Lass uns gehen.
StudienbasisDie Verwendung von DNA als Baumaterial in Nanostrukturen mit einer Vielzahl von Anwendungen in den letzten Jahren ist zum Ziel vieler Studien und Experimente geworden. Viele von ihnen haben hervorragende Ergebnisse erzielt, aber wie wir wissen, sind der Perfektion keine Grenzen gesetzt. Als Beispiel zitieren die Forscher monodisperse DNA-Strukturen im Megadalt-Maßstab mit einem hohen Gerinnungskoeffizienten, die unter Verwendung langer ssDNA-Frames (einzelsträngige DNA) - einzelsträngige / einzelsträngige DNA - realisiert wurden. In diesem Fall ist jedoch die Länge der DNA-Stränge die Hauptbeschränkung für die Größe einer einzelnen Origami-DNA. Diese Einschränkung kann durch die Bildung einer Struktur umgangen werden, die aus mehreren Origami gleichzeitig besteht.
Wir wissen auch bereits (
Tic-Tac-Toe: eine Demonstration des kontrollierten Prozesses der Rekonfiguration von DNA-Strukturen ), dass einige DNA-Strukturen mithilfe von DNA-Kacheln erstellt werden können, wodurch Sie eine komplexe Geometrie dieser Struktur bilden können.
Aber auch hier kommt es darauf an, dass der Prozess manuell ausgeführt wird, und dies ist lang und trostlos. Was mögen Wissenschaftler? Das ist richtig, Automatisierung oder zumindest teilweise Automatisierung. Ähnliche Tools sind bereits verfügbar (ein Beispiel für Wissenschaftler - caDNAno oder Tiamat), diese Programme haben jedoch ihre Nachteile. Erstens die manuelle Erstellung des Rahmens. Zweitens die Einschränkungen der obigen Algorithmen in Bezug auf die Systematisierung des Entwurfs der Geometrie der Struktur.
Kurz gesagt, in diesem Artikel haben sich die Forscher das Ziel gesetzt, einen Algorithmus zu entwickeln, der fast alles alleine macht. Somit kann jede Person nur die Parameter der Außenhülle der gewünschten Struktur einstellen, und das Programm füllt den inneren Teil unabhängig mit allen erforderlichen DNA-Threads, Heftklammern, Sequenzen und mehr. Sie können den Prozess auch in umgekehrter Reihenfolge ausführen - stellen Sie sozusagen alle "Innenseiten" ein, das Strukturraster und der Algorithmus bilden das richtige und optimalste Framework dafür.
Dieses Open-Source-Programm (PERDIX) steht absolut jedem zur Verfügung, wofür dank der Forscher. Somit kann jeder nicht nur mit dem Programm selbst „herumspielen“, sondern möglicherweise auch einige Verbesserungen an seinem Code vornehmen. Wo Sie es herunterladen und wie Sie es starten können, erfahren Sie am Ende des Artikels.
Schauen wir uns nun Beispiele für die Anwendung dieses Programms an, die uns das Forschungsteam zur Verfügung gestellt hat.
PERDIX-Validierungsergebnisse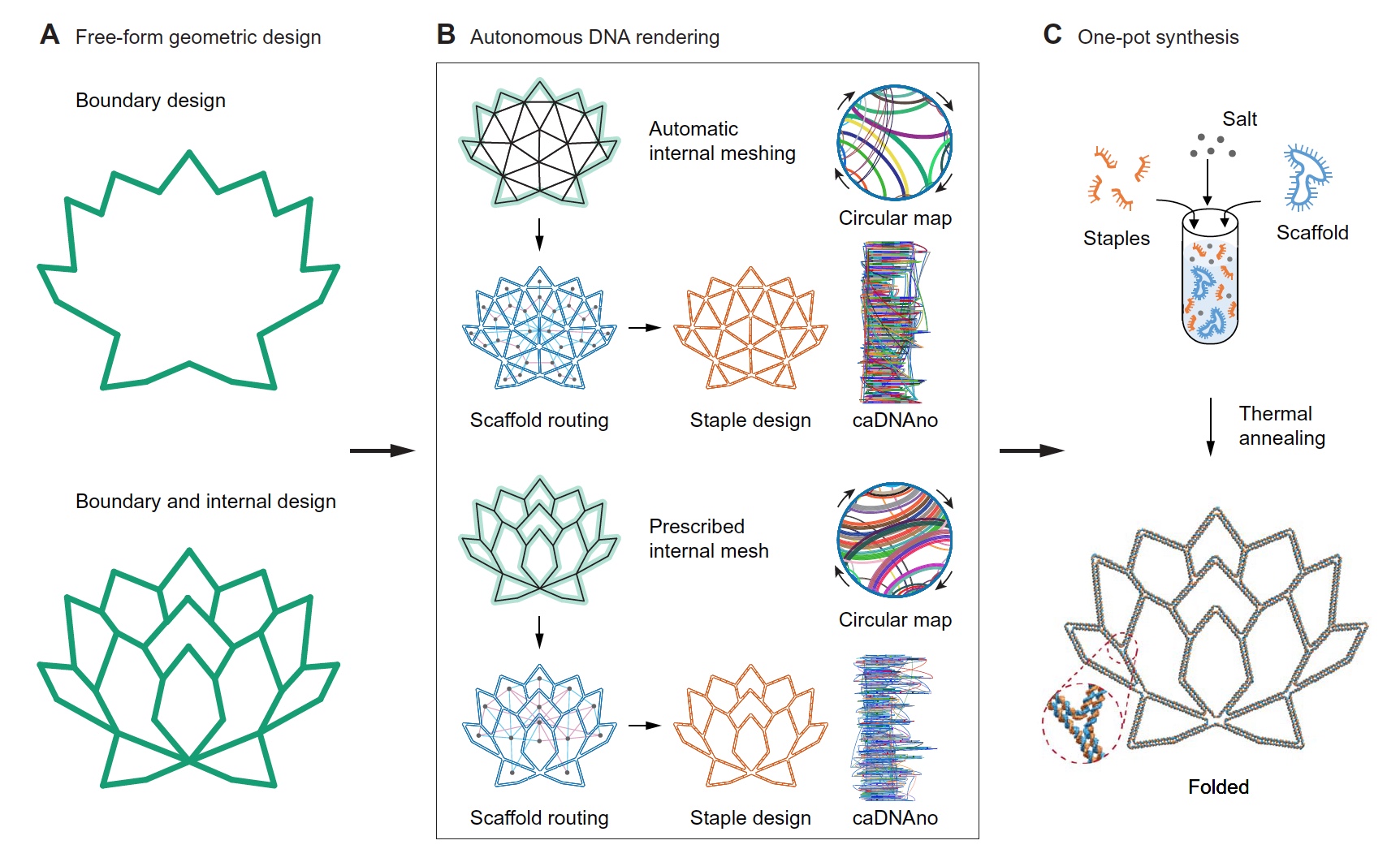 Bild Nr. 1
Bild Nr. 1Als Eingabedaten wird entweder das Bild des Randes der gewünschten Struktur (
1A , oben) oder ein Bild mit genaueren geometrischen Details (
1A , unten) verwendet. Ferner füllt das Programm selbst den Innenraum.
Wenn nur der „Umriss“ angegeben wird, wird die interne Geometrie des Gitters der Struktur durch Dreiecke unter Verwendung des Open-Source-Programms DistMesh gebildet, bei dem nur die Gitterdichte als notwendiger Parameter eingegeben werden muss.
Wenn die Form und die geometrischen Linien angegeben sind (
1A , unten), wird Shapely verwendet - ein Python-Paket zum Bearbeiten und Analysieren flacher geometrischer Objekte. Erzeugt formschön ein polygonales Netz, in dem seine Linien und ihre Schnittpunkte die erforderliche Geometrie des Rahmens bilden.
Ferner war es nach Bestimmung der gewünschten Form erforderlich, die Nennlänge der Rippe einzustellen, die ≥ 38 bp (gepaarte Basen) oder 12,58 nm sein sollte. Somit sind definitiv mindestens zwei Paarungskreuzungen pro Kante in der erforderlichen Struktur vorhanden.
Dann werden alle Ziellinien der zukünftigen Struktur in DX-Kanten transformiert, in denen alle Eckpunkte Verbindungspunkte mit mehreren Gittern sind (
1B ). Nach den Prinzipien der Graphentheorie wird automatisch die optimale Skelettstruktur gebildet, wonach der Prozess der Zuweisung einer Sequenz zusätzlicher DNA-Klammern abgeschlossen ist. Die manuelle Bearbeitung der Ausgabedaten (Ergebnisse) erfolgt über die generierte caDNAno-Datei.
Wissenschaftler nennen ein sehr wichtiges Merkmal ihres Systems die Tatsache, dass die Rippen nicht einer ganzzahligen Anzahl von Doppel-B-DNA-Helices (10,5 bp) entsprechen müssen. Auf diese Weise erhalten Sie mehr Freiheit bei der Gestaltung der Geometrie der Struktur.
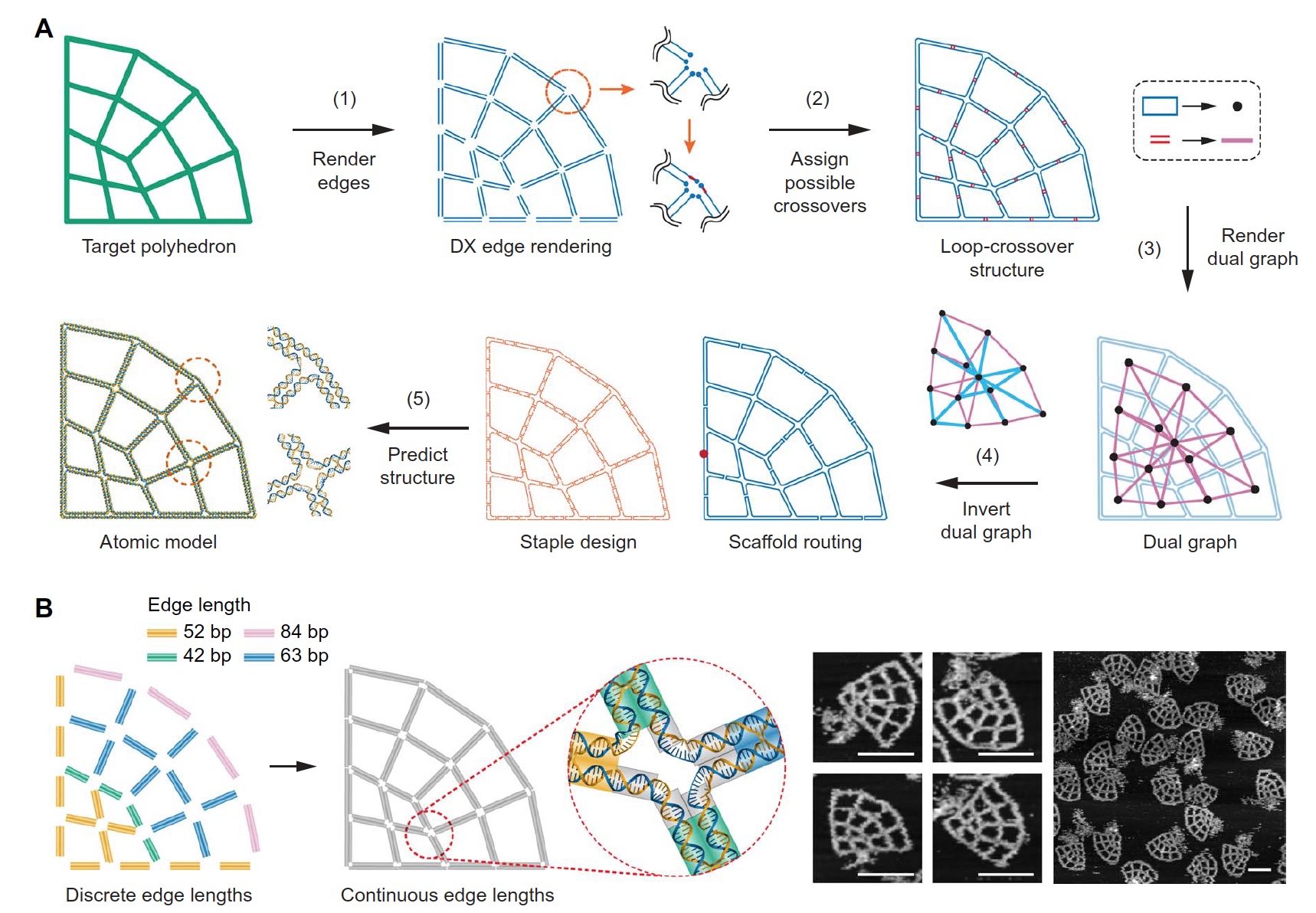 Bild Nr. 2
Bild Nr. 2Um den automatischen Teil des Prozesses zum Formen der Geometrie der Struktur schnell und genau ausführen zu können, müssen zunächst alle Kanten in DX-Motive konvertiert werden, indem jede Kante der Zielgeometrie mit zwei antiparallelen Rahmenlinien (
2A ) gerendert wird. Dann werden sie an einem einzelnen Scheitelpunkt kombiniert, wodurch jede Kante Teil einer Schleife wird. Und dies führt wiederum zur Bildung einer einzelnen großen externen Schleife, die aus kleinen Schleifen besteht. Der nächste Schritt besteht darin, alle möglichen Überkreuzungsoptionen zwischen benachbarten Schleifen zu finden, dh die Bildung einer Schleifenschnittstruktur.
Der dritte Schritt in diesem Prozess besteht darin, alle resultierenden Strukturen als eine Menge von Knoten und alle Überkreuzungen als Kanten der zukünftigen Struktur darzustellen.
Als nächstes wird der Spanning Tree des Doppelgraphen berechnet und anschließend in ein ssDNA-Framework konvertiert. Dieser Vorgang ist vollautomatisch. Und schließlich wird auf atomarer Ebene ein dreidimensionales Strukturmodell erzeugt.
Fig.
2B zeigt zwei Optionen zur Bildung der erforderlichen Geometrie der Struktur: mit diskreter Kantenlänge oder mit kontinuierlicher Kantenlänge. Mit der zweiten Option können Sie eine Struktur mit einer beliebigen Kantenlänge erstellen, wodurch wiederum die größte Koagulation erzielt werden kann. Diese Variationen der Geometriestrukturierung wurden unter Verwendung eines Raster-Rasterkraftmikroskops verifiziert (Bilder rechts in Bild
2B ).
 Bild Nr. 3
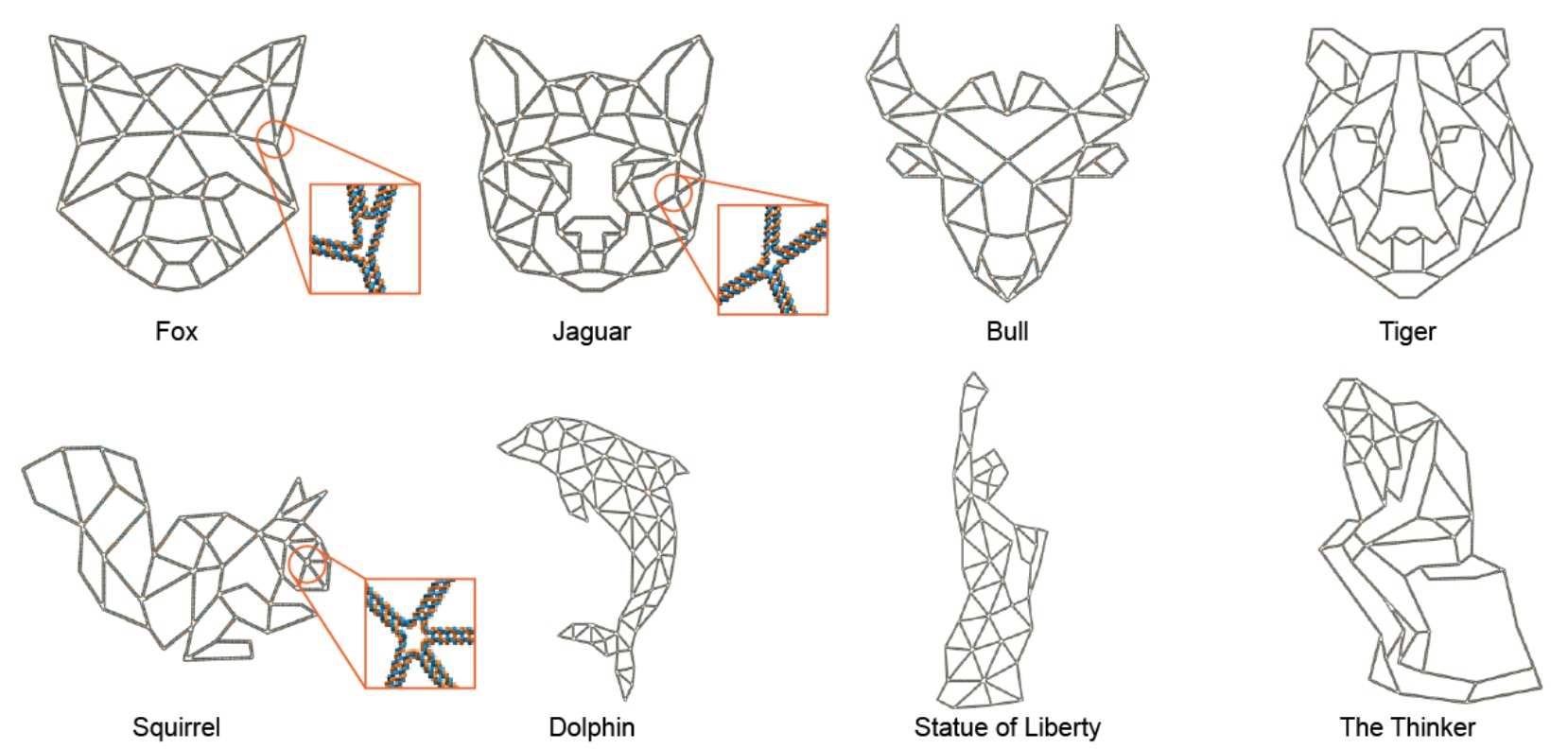
Bild Nr. 3Die Forscher haben sich auch zum Ziel gesetzt, Aspekte der Zielgeometrie wie mechanische Steifheit, Formgenauigkeit, Größe der Struktur, Größe des inneren Netzes und Arten seiner Elemente genauer zu untersuchen. Es war auch notwendig zu verstehen, wie wichtig die N-Zweige spielen, dh die Anzahl der Verbindungen zwischen den Kanten und infolgedessen die Gesamtkomplexität der Struktur. Die Analyse der AFM-Daten zeigte, dass zweidimensionale Strukturen mit einer großen Anzahl von Verzweigungen und einer längeren Kantenlänge (aufgrund ungepaarter Nukleotide) ziemlich gut gebildet werden und die Heterogenität der Partikel in ihrer Struktur minimal ist.
Das interne Gitter der Struktur hatte drei Optionen: Dreiecke mit derselben Richtung, Quadrate und Dreiecke mit unterschiedlichen Richtungen (
3C ). Es war das Modell mit Dreiecken, das im Gegensatz zum Modell mit Quadraten die größte Genauigkeit bei der Bildung der erforderlichen Geometrie zeigte. Das Vorhandensein gemischter Richtungen im Dreiecksmodell ermöglichte es sozusagen, symmetrischere N-Zweige und eine genauere Form (weniger verzerrt) zu erhalten.
Wissenschaftler stellen fest, dass die Analyse des inneren Netzes der simulierten Struktur äußerst wichtig ist, um die mechanische Steifigkeit dieser Struktur zu verstehen. Und genau das oben beschriebene Modell ist ideal, um ausreichend starke, genaue (im Hinblick auf die Einhaltung bestimmter Parameter) und flexible Strukturen zu schaffen.
 Bild Nr. 4
Bild Nr. 4Natürlich sind Wissenschaftler sozusagen nicht ohne Sinn für Schönheit. Daher beschlossen sie, die Möglichkeiten ihrer Entwicklung zu demonstrieren, indem sie Strukturen mit 15 verschiedenen Formen entwarfen, genauer gesagt mit verschiedenen inneren Gittern und Rahmen. Wir können die Optionen im Bild oben sehen: Hier sind die „üblichen“ Quadrate und ein Viertel eines Kreises und sogar ein Lotus.
Open Source SoftwareWie bereits erwähnt, steht die Arbeit der Forscher allen unter link () offen, wo Sie die erforderlichen Programmelemente finden (MATLAB, Python 2.7 und Shapely 1.6.4).
Was den Algorithmus zur Bildung der Geometrie von DNA-Origami-Strukturen betrifft, so werden die Anweisungen zum Herunterladen und Ausführen im Folgenden im Videoformat dargestellt. Ich werde alle Videos unter dem Spoiler verstecken, um den Artikel nicht zu dehnen.
Video Nr. 1: Start von PERDIXDer Link zum Herunterladen der erforderlichen Dateien im Video funktioniert jetzt nicht. Hier ist eine Alternative -
Download-Link .
Video Nr. 2: Entwerfen des Rahmens (Umfangs) einer Struktur in PERDIX
Video 3: Entwerfen des Frameworks und der internen Struktur in PERDIX
Video Nr. 4: Atommodelle (verschiedene Gitter)
Video Nr. 5: Atommodelle (N-Zweige)
Video Nr. 6: Atommodelle (L-Modell)
Video Nr. 7: Atommodelle (gebogener "Zweig")
Für eine detailliertere Kenntnis dieser Studie und ihrer Ergebnisse empfehle ich dringend, dass Sie sich den
Bericht der Forschungsgruppe und
zusätzliche Materialien dazu ansehen.
NachwortDiese Studie zielte darauf ab, einen ziemlich zeitaufwändigen und zeitaufwändigen Prozess zu vereinfachen. Und es hat geklappt. Mit dem PERDIX-Algorithmus können Sie Strukturen mit sehr unterschiedlichen Geometrien erstellen und gleichzeitig die minimal erforderlichen Parameter festlegen. DNA ist seit langem ein Untersuchungsgegenstand für Wissenschaftler, nicht nur als Teil aller Lebens- und Informationsträger, sondern auch als mögliche Option für die Grundlage zukünftiger Technologien. Solche Arbeiten ermöglichen es, detaillierter (und klarer) zu verstehen, wie breit die Möglichkeiten von Nanostrukturen sind, die auf der Basis von DNA-Origami erzeugt werden.
Der zweite angenehme Moment in dieser Studie ist, zumindest für mich, dass der Algorithmus für alle verfügbar ist. Jeder kann es benutzen, jeder kann es verbessern. Durch den uneingeschränkten Zugriff sowohl auf ihren Bericht als auch auf die Software selbst tragen Wissenschaftler nicht nur zur Popularisierung ihres wissenschaftlichen Fachgebiets bei, sondern auch zur Wissenschaft als solcher.
Ich traf Artikel, in denen Wissenschaftler das bezahlte System für den Zugang zu Berichten sehr kritisierten und argumentierten, dass dies eine zusätzliche Barriere zwischen Wissen und gewöhnlichen Menschen darstellt. Die Situation in dieser Angelegenheit ist sehr zweideutig und kontrovers, da jeder Geld verdienen sollte (sowohl wissenschaftliche Veröffentlichungen, Forschungsgruppen als auch wissenschaftliche Websites), aber die hohen Kosten für den Zugang zu einigen Studien machen sie für gewöhnliche Wissenschaftsliebhaber, Studenten und sogar einige Professoren und Studenten unzugänglich wissenschaftliche Figuren. Wie gesagt, die Frage ist nicht eindeutig, daher werden wir nicht viel darauf eingehen. Auf jeden Fall bin ich froh, dass unsere Helden uns heute vollen Zugang zu ihrer Arbeit gewährt haben, was nicht nur Aufmerksamkeit, sondern auch offene Bewunderung verdient.
Vielen Dank für Ihre Aufmerksamkeit, bleiben Sie neugierig und haben Sie eine gute Arbeitswoche, Jungs.
Vielen Dank für Ihren Aufenthalt bei uns. Gefällt dir unser Artikel? Möchten Sie weitere interessante Materialien sehen? Unterstützen Sie uns, indem Sie eine Bestellung
aufgeben oder Ihren Freunden empfehlen, einen
Rabatt von 30% für Habr-Benutzer auf ein einzigartiges Analogon von Einstiegsservern, das wir für Sie erfunden haben: Die ganze Wahrheit über VPS (KVM) E5-2650 v4 (6 Kerne) 10 GB DDR4 240 GB SSD 1 Gbit / s von $ 20 oder wie teilt man den Server? (Optionen sind mit RAID1 und RAID10, bis zu 24 Kernen und bis zu 40 GB DDR4 verfügbar).
VPS (KVM) E5-2650 v4 (6 Kerne) 10 GB DDR4 240 GB SSD 1 Gbit / s bis zum Frühjahr kostenlos, wenn Sie für einen Zeitraum von sechs Monaten bezahlen, können Sie
hier bestellen.
Dell R730xd 2 mal günstiger? Nur wir haben
2 x Intel Dodeca-Core Xeon E5-2650v4 128 GB DDR4 6 x 480 GB SSD 1 Gbit / s 100 TV von 249 US-Dollar in den Niederlanden und den USA! Lesen Sie mehr über
den Aufbau eines Infrastrukturgebäudes. Klasse mit Dell R730xd E5-2650 v4 Servern für 9.000 Euro für einen Cent?