
Hallo Habr!
Im vergangenen Herbst fand in Kaggle ein Wettbewerb zur Klassifizierung von handgezeichneten Quick Draw Doodle Recognition-Bildern statt, an dem unter anderem ein Team von R-Schiks, bestehend aus
Artem Klevtsov ,
Philip Upravitelev und
Andrey Ogurtsov, teilnahm . Wir werden den Wettbewerb nicht im Detail beschreiben, dies wurde bereits in einer
kürzlich erschienenen Veröffentlichung durchgeführt .
Dieses Mal gab es keine Medaillen mit landwirtschaftlichen Arzneimitteln, aber es wurden viele wertvolle Erfahrungen gesammelt. Deshalb möchte ich der Community einige der interessantesten und nützlichsten Dinge auf Kagl und in der täglichen Arbeit erzählen. Zu den behandelten Themen gehören: Hartes Leben ohne
OpenCV , Parsen von JSONs (diese Beispiele
zeigen die Integration von C ++ - Code in Skripte oder Pakete in R mithilfe von
Rcpp ), Parametrisierung von Skripten und Dockerisierung der endgültigen Lösung. Der gesamte Code aus der Nachricht in einer für den Start geeigneten Form ist im
Repository verfügbar.
Inhalt:
- Effektives Laden von Daten von CSV in die MonetDB-Datenbank
- Chargenvorbereitung
- Iteratoren zum Entladen von Stapeln aus der Datenbank
- Auswahl der Modellarchitektur
- Skriptparametrierung
- Docking-Skripte
- Verwenden mehrerer GPUs in der Google Cloud
- Anstelle einer Schlussfolgerung
1. Effektives Laden von Daten von CSV in die MonetDB-Datenbank
Die Daten in diesem Wettbewerb werden nicht in Form von vorgefertigten Bildern bereitgestellt, sondern in Form von 340 CSV-Dateien (eine Datei für jede Klasse), die JSONs mit Punktkoordinaten enthalten. Wenn Sie diese Punkte mit Linien verbinden, erhalten Sie das endgültige Bild mit einer Größe von 256 x 256 Pixel. Außerdem wird für jeden Datensatz eine Beschriftung angegeben, ob das Bild von dem zum Zeitpunkt der Datenerfassung verwendeten Klassifizierer korrekt erkannt wurde, der aus zwei Buchstaben bestehende Code des Wohnsitzlandes des Autors, eine eindeutige Kennung, ein Zeitstempel und ein Klassenname, die mit dem Dateinamen übereinstimmen. Eine vereinfachte Version der Quelldaten wiegt im Archiv 7,4 GB und nach dem Entpacken etwa 20 GB. Die vollständigen Daten nach dem Entpacken benötigen 240 GB. Die Organisatoren garantierten, dass beide Versionen dieselben Zeichnungen reproduzieren, d. H. Die Vollversion ist redundant. In jedem Fall wurde das Speichern von 50 Millionen Bildern in Grafikdateien oder in Arrays sofort als unrentabel angesehen, und wir beschlossen, alle CSV-Dateien aus dem Archiv train_simplified.zip in einer Datenbank zusammenzuführen und anschließend für jeden Stapel sofort Bilder mit der richtigen Größe zu generieren .
Als DBMS wurde die etablierte MonetDB gewählt, nämlich die Implementierung für R in Form des MonetDBLite- Pakets. Das Paket enthält eine eingebettete Version des Datenbankservers und ermöglicht es Ihnen, den Server direkt aus der R-Sitzung zu entfernen und dort damit zu arbeiten. Das Erstellen und Herstellen einer Datenbank erfolgt über einen Befehl:
con <- DBI::dbConnect(drv = MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR"))
Wir müssen zwei Tabellen erstellen: eine für alle Daten, die andere für Overhead-Informationen zu den heruntergeladenen Dateien (nützlich, wenn etwas schief geht und der Vorgang nach dem Laden mehrerer Dateien fortgesetzt werden muss):
Erstellen Sie Tabellen if (!DBI::dbExistsTable(con, "doodles")) { DBI::dbCreateTable( con = con, name = "doodles", fields = c( "countrycode" = "char(2)", "drawing" = "text", "key_id" = "bigint", "recognized" = "bool", "timestamp" = "timestamp", "word" = "text" ) ) } if (!DBI::dbExistsTable(con, "upload_log")) { DBI::dbCreateTable( con = con, name = "upload_log", fields = c( "id" = "serial", "file_name" = "text UNIQUE", "uploaded" = "bool DEFAULT false" ) ) }
Der schnellste Weg, Daten in die Datenbank zu laden, bestand darin, CSV-Dateien direkt mit SQL zu kopieren - dem Befehl COPY OFFSET 2 INTO tablename FROM path USING DELIMITERS ',','\\n','\"' NULL AS '' BEST EFFORT , wobei tablename ist Der Tabellenname und der path sind der Pfad zur Datei. Später wurde ein anderer Weg gefunden , um die Geschwindigkeit zu erhöhen: Ersetzen BEST EFFORT einfach BEST EFFORT durch LOCKED BEST EFFORT . Bei der Arbeit mit dem Archiv stellte sich heraus, dass die integrierte unzip in R mit einer Reihe von Dateien aus dem Archiv nicht korrekt funktioniert System unzip (mit dem Parameter getOption("unzip") ).
Funktion zum Schreiben in die Datenbank #' @title #' #' @description #' CSV- ZIP- #' #' @param con ( `MonetDBEmbeddedConnection`). #' @param tablename . #' @oaram zipfile ZIP-. #' @oaram filename ZIP-. #' @param preprocess , . #' `data` ( `data.table`). #' #' @return `TRUE`. #' upload_file <- function(con, tablename, zipfile, filename, preprocess = NULL) { # checkmate::assert_class(con, "MonetDBEmbeddedConnection") checkmate::assert_string(tablename) checkmate::assert_string(filename) checkmate::assert_true(DBI::dbExistsTable(con, tablename)) checkmate::assert_file_exists(zipfile, access = "r", extension = "zip") checkmate::assert_function(preprocess, args = c("data"), null.ok = TRUE) # path <- file.path(tempdir(), filename) unzip(zipfile, files = filename, exdir = tempdir(), junkpaths = TRUE, unzip = getOption("unzip")) on.exit(unlink(file.path(path))) # if (!is.null(preprocess)) { .data <- data.table::fread(file = path) .data <- preprocess(data = .data) data.table::fwrite(x = .data, file = path, append = FALSE) rm(.data) } # CSV sql <- sprintf( "COPY OFFSET 2 INTO %s FROM '%s' USING DELIMITERS ',','\\n','\"' NULL AS '' BEST EFFORT", tablename, path ) # DBI::dbExecute(con, sql) # DBI::dbExecute(con, sprintf("INSERT INTO upload_log(file_name, uploaded) VALUES('%s', true)", filename)) return(invisible(TRUE)) }
Wenn Sie die Tabelle vor dem Schreiben in die Datenbank konvertieren müssen, reicht es aus, die Funktion zu übergeben, mit der die Daten in das preprocess konvertiert werden.
Code zum sequentiellen Laden von Daten in die Datenbank:
Daten in die Datenbank schreiben # files <- unzip(zipfile, list = TRUE)$Name # , to_skip <- DBI::dbGetQuery(con, "SELECT file_name FROM upload_log")[[1L]] files <- setdiff(files, to_skip) if (length(files) > 0L) { # tictoc::tic() # pb <- txtProgressBar(min = 0L, max = length(files), style = 3) for (i in seq_along(files)) { upload_file(con = con, tablename = "doodles", zipfile = zipfile, filename = files[i]) setTxtProgressBar(pb, i) } close(pb) # tictoc::toc() } # 526.141 sec elapsed - SSD->SSD # 558.879 sec elapsed - USB->SSD
Die Datenladezeit kann abhängig von den Geschwindigkeitseigenschaften des verwendeten Laufwerks variieren. In unserem Fall dauert das Lesen und Schreiben innerhalb derselben SSD oder von einem USB-Flash-Laufwerk (Quelldatei) auf eine SSD (Datenbank) weniger als 10 Minuten.
Es dauert noch einige Sekunden, um eine Spalte mit einer Ganzzahlklassenbezeichnung und einer ORDERED INDEX ( ORDERED INDEX ) mit Zeilennummern zu erstellen, mit denen Fälle beim Erstellen von ORDERED INDEX ausgewählt werden:
Erstellen Sie zusätzliche Spalten und einen Index message("Generate lables") invisible(DBI::dbExecute(con, "ALTER TABLE doodles ADD label_int int")) invisible(DBI::dbExecute(con, "UPDATE doodles SET label_int = dense_rank() OVER (ORDER BY word) - 1")) message("Generate row numbers") invisible(DBI::dbExecute(con, "ALTER TABLE doodles ADD id serial")) invisible(DBI::dbExecute(con, "CREATE ORDERED INDEX doodles_id_ord_idx ON doodles(id)"))
Um das Problem der Erstellung eines Stapels „on the fly“ zu lösen, mussten wir die maximale Geschwindigkeit erreichen, mit der zufällige Zeichenfolgen aus der doodles Tabelle extrahiert werden. Dafür haben wir 3 Tricks benutzt. Die erste bestand darin, die Dimension des Typs zu reduzieren, in dem die Beobachtungs-ID gespeichert ist. Im Originaldatensatz wird der bigint Typ zum Speichern der ID benötigt, aber die Anzahl der Beobachtungen ermöglicht das Anpassen ihrer Bezeichner, die der Seriennummer entsprechen, in den int Typ. Die Suche ist viel schneller. Der zweite Trick bestand darin, ORDERED INDEX - diese Entscheidung wurde empirisch getroffen und alle verfügbaren Optionen sortiert. Der dritte bestand darin, parametrisierte Abfragen zu verwenden. Die Essenz der Methode besteht darin, den Befehl PREPARE einmal auszuführen und dann den vorbereiteten Ausdruck zu verwenden, wenn ein Heap des gleichen PREPARE erstellt wird. In Wirklichkeit liegt der Gewinn gegenüber dem einfachen SELECT im Bereich des statistischen Fehlers.
Das Füllen von Daten verbraucht nicht mehr als 450 MB RAM. Das heißt, der beschriebene Ansatz ermöglicht es Ihnen, Datensätze mit einem Gewicht von mehreren zehn Gigabyte auf fast jeder Budget-Hardware zu drehen, einschließlich einiger Single-Board-Computer, was ziemlich cool ist.
Es bleibt noch die Messung der Extraktionsrate von (zufälligen) Daten vorzunehmen und die Skalierung bei der Probenahme von Chargen unterschiedlicher Größe zu bewerten:
Benchmark-Datenbank library(ggplot2) set.seed(0) # con <- DBI::dbConnect(MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR")) # prep_sql <- function(batch_size) { sql <- sprintf("PREPARE SELECT id FROM doodles WHERE id IN (%s)", paste(rep("?", batch_size), collapse = ",")) res <- DBI::dbSendQuery(con, sql) return(res) } # fetch_data <- function(rs, batch_size) { ids <- sample(seq_len(n), batch_size) res <- DBI::dbFetch(DBI::dbBind(rs, as.list(ids))) return(res) } # res_bench <- bench::press( batch_size = 2^(4:10), { rs <- prep_sql(batch_size) bench::mark( fetch_data(rs, batch_size), min_iterations = 50L ) } ) # cols <- c("batch_size", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # batch_size min median max `itr/sec` total_time n_itr # <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 16 23.6ms 54.02ms 93.43ms 18.8 2.6s 49 # 2 32 38ms 84.83ms 151.55ms 11.4 4.29s 49 # 3 64 63.3ms 175.54ms 248.94ms 5.85 8.54s 50 # 4 128 83.2ms 341.52ms 496.24ms 3.00 16.69s 50 # 5 256 232.8ms 653.21ms 847.44ms 1.58 31.66s 50 # 6 512 784.6ms 1.41s 1.98s 0.740 1.1m 49 # 7 1024 681.7ms 2.72s 4.06s 0.377 2.16m 49 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = 1)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() DBI::dbDisconnect(con, shutdown = TRUE)
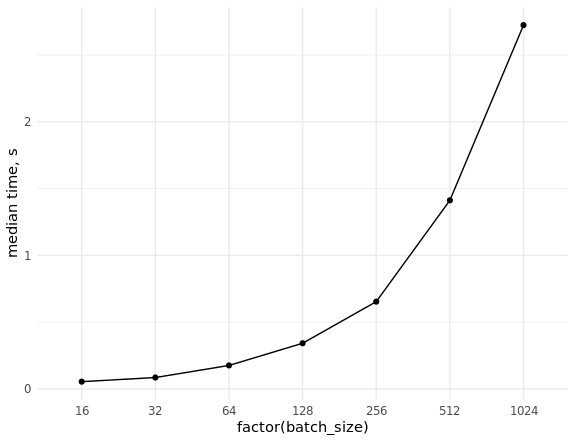
2. Vorbereitung der Chargen
Der gesamte Prozess der Chargenvorbereitung besteht aus folgenden Schritten:
- Analysieren mehrerer JSONs, die Linienvektoren mit Punktkoordinaten enthalten.
- Zeichnen farbiger Linien durch die Koordinaten der Punkte im Bild der gewünschten Größe (z. B. 256 x 256 oder 128 x 128).
- Konvertieren Sie die resultierenden Bilder in einen Tensor.
Im Rahmen des Wettbewerbs zwischen dem Kernel in Python wurde das Problem hauptsächlich mit OpenCV gelöst. Eines der einfachsten und offensichtlichsten Analoga auf R sieht folgendermaßen aus:
Implementieren Sie die Konvertierung von JSON in Tensor auf R. r_process_json_str <- function(json, line.width = 3, color = TRUE, scale = 1) { # JSON coords <- jsonlite::fromJSON(json, simplifyMatrix = FALSE) tmp <- tempfile() # on.exit(unlink(tmp)) png(filename = tmp, width = 256 * scale, height = 256 * scale, pointsize = 1) # plot.new() # plot.window(xlim = c(256 * scale, 0), ylim = c(256 * scale, 0)) # cols <- if (color) rainbow(length(coords)) else "#000000" for (i in seq_along(coords)) { lines(x = coords[[i]][[1]] * scale, y = coords[[i]][[2]] * scale, col = cols[i], lwd = line.width) } dev.off() # 3- res <- png::readPNG(tmp) return(res) } r_process_json_vector <- function(x, ...) { res <- lapply(x, r_process_json_str, ...) # 3- 4- res <- do.call(abind::abind, c(res, along = 0)) return(res) }
Das Zeichnen wird mit Standard-R-Tools durchgeführt und in einem im RAM gespeicherten temporären PNG gespeichert (unter Linux befinden sich die temporären R-Verzeichnisse im im RAM /tmp ). Anschließend wird diese Datei in Form eines dreidimensionalen Arrays mit Zahlen im Bereich von 0 bis 1 gelesen. Dies ist wichtig, da das häufigere BMP in ein Roharray mit hexadezimalen Farbcodes eingelesen wird.
Testen Sie das Ergebnis:
zip_file <- file.path("data", "train_simplified.zip") csv_file <- "cat.csv" unzip(zip_file, files = csv_file, exdir = tempdir(), junkpaths = TRUE, unzip = getOption("unzip")) tmp_data <- data.table::fread(file.path(tempdir(), csv_file), sep = ",", select = "drawing", nrows = 10000) arr <- r_process_json_str(tmp_data[4, drawing]) dim(arr) # [1] 256 256 3 plot(magick::image_read(arr))

Die Charge selbst wird wie folgt gebildet:
res <- r_process_json_vector(tmp_data[1:4, drawing], scale = 0.5) str(res) # num [1:4, 1:128, 1:128, 1:3] 1 1 1 1 1 1 1 1 1 1 ... # - attr(*, "dimnames")=List of 4 # ..$ : NULL # ..$ : NULL # ..$ : NULL # ..$ : NULL
Diese Implementierung schien uns nicht optimal zu sein, da die Bildung großer Chargen unangemessen viel Zeit in Anspruch nimmt und wir beschlossen, die Erfahrung unserer Kollegen mit der leistungsstarken OpenCV-Bibliothek zu nutzen . Zu diesem Zeitpunkt gab es kein fertiges Paket für R (es gibt noch kein Paket), daher wurde eine minimale Implementierung der erforderlichen Funktionalität in C ++ mit Integration in R-Code unter Verwendung von Rcpp geschrieben .
Um das Problem zu lösen, wurden die folgenden Pakete und Bibliotheken verwendet:
- OpenCV für Imaging und Strichzeichnung. Wir haben vorinstallierte Systembibliotheken und Header-Dateien sowie dynamische Verknüpfungen verwendet.
- xtensor für die Arbeit mit mehrdimensionalen Arrays und Tensoren. Wir haben Header-Dateien verwendet, die im gleichnamigen R-Paket enthalten sind. Mit der Bibliothek können Sie mit mehrdimensionalen Arrays arbeiten, sowohl in Zeilen- als auch in Spalten-Hauptreihenfolge.
- ndjson zum Parsen von JSON. Diese Bibliothek wird in xtensor automatisch verwendet, wenn sie im Projekt verfügbar ist.
- RcppThread zum Organisieren der Multithread-Verarbeitung eines Vektors aus JSON. Verwendete die von diesem Paket bereitgestellten Header-Dateien. Das Paket unterscheidet sich von dem populäreren RcppParallel unter anderem durch seinen eingebauten Interrupt-Mechanismus.
Es ist erwähnenswert, dass sich xtensor nur als Fund herausstellte: Neben umfangreichen Funktionen und hoher Leistung erwiesen sich die Entwickler als sehr reaktionsschnell und beantworteten die gestellten Fragen schnell und ausführlich. Mit ihrer Hilfe war es möglich, die Transformation von OpenCV-Matrizen in Xtensortensoren sowie eine Methode zur Kombination von dreidimensionalen Bildtensoren zu einem vierdimensionalen Tensor mit der richtigen Dimension (tatsächlich der Charge) zu implementieren.
Studienmaterialien für Rcpp, xtensor und RcppThread Um Dateien mithilfe von Systemdateien und dynamischer Verknüpfung mit im System installierten Bibliotheken zu kompilieren, haben wir den im Rcpp- Paket implementierten Plugin-Mechanismus verwendet. Um Pfade und Flags automatisch zu finden, haben wir das beliebte Linux-Dienstprogramm pkg-config verwendet .
Implementierung eines Rcpp-Plugins zur Verwendung der OpenCV-Bibliothek Rcpp::registerPlugin("opencv", function() { # pkg_config_name <- c("opencv", "opencv4") # pkg-config pkg_config_bin <- Sys.which("pkg-config") # checkmate::assert_file_exists(pkg_config_bin, access = "x") # OpenCV pkg-config check <- sapply(pkg_config_name, function(pkg) system(paste(pkg_config_bin, pkg))) if (all(check != 0)) { stop("OpenCV config for the pkg-config not found", call. = FALSE) } pkg_config_name <- pkg_config_name[check == 0] list(env = list( PKG_CXXFLAGS = system(paste(pkg_config_bin, "--cflags", pkg_config_name), intern = TRUE), PKG_LIBS = system(paste(pkg_config_bin, "--libs", pkg_config_name), intern = TRUE) )) })
Aufgrund des Plugins werden beim Kompilieren folgende Werte ersetzt:
Rcpp:::.plugins$opencv()$env # $PKG_CXXFLAGS # [1] "-I/usr/include/opencv" # # $PKG_LIBS # [1] "-lopencv_shape -lopencv_stitching -lopencv_superres -lopencv_videostab -lopencv_aruco -lopencv_bgsegm -lopencv_bioinspired -lopencv_ccalib -lopencv_datasets -lopencv_dpm -lopencv_face -lopencv_freetype -lopencv_fuzzy -lopencv_hdf -lopencv_line_descriptor -lopencv_optflow -lopencv_video -lopencv_plot -lopencv_reg -lopencv_saliency -lopencv_stereo -lopencv_structured_light -lopencv_phase_unwrapping -lopencv_rgbd -lopencv_viz -lopencv_surface_matching -lopencv_text -lopencv_ximgproc -lopencv_calib3d -lopencv_features2d -lopencv_flann -lopencv_xobjdetect -lopencv_objdetect -lopencv_ml -lopencv_xphoto -lopencv_highgui -lopencv_videoio -lopencv_imgcodecs -lopencv_photo -lopencv_imgproc -lopencv_core"
Der Code zum Implementieren der JSON-Analyse und zum Erstellen eines Stapels zum Übertragen auf das Modell wird unter dem Spoiler angegeben. Fügen Sie zunächst das lokale Projektverzeichnis hinzu, um nach Header-Dateien zu suchen (für ndjson erforderlich):
Sys.setenv("PKG_CXXFLAGS" = paste0("-I", normalizePath(file.path("src"))))
Implementierung der Konvertierung von JSON in Tensor in C ++ // [[Rcpp::plugins(cpp14)]] // [[Rcpp::plugins(opencv)]] // [[Rcpp::depends(xtensor)]] // [[Rcpp::depends(RcppThread)]] #include <xtensor/xjson.hpp> #include <xtensor/xadapt.hpp> #include <xtensor/xview.hpp> #include <xtensor-r/rtensor.hpp> #include <opencv2/core/core.hpp> #include <opencv2/highgui/highgui.hpp> #include <opencv2/imgproc/imgproc.hpp> #include <Rcpp.h> #include <RcppThread.h> // using RcppThread::parallelFor; using json = nlohmann::json; using points = xt::xtensor<double,2>; // JSON using strokes = std::vector<points>; // JSON using xtensor3d = xt::xtensor<double, 3>; // using xtensor4d = xt::xtensor<double, 4>; // using rtensor3d = xt::rtensor<double, 3>; // R using rtensor4d = xt::rtensor<double, 4>; // R // // const static int SIZE = 256; // // . https://en.wikipedia.org/wiki/Pixel_connectivity#2-dimensional const static int LINE_TYPE = cv::LINE_4; // const static int LINE_WIDTH = 3; // // https://docs.opencv.org/3.1.0/da/d54/group__imgproc__transform.html#ga5bb5a1fea74ea38e1a5445ca803ff121 const static int RESIZE_TYPE = cv::INTER_LINEAR; // OpenCV- template <typename T, int NCH, typename XT=xt::xtensor<T,3,xt::layout_type::column_major>> XT to_xt(const cv::Mat_<cv::Vec<T, NCH>>& src) { // std::vector<int> shape = {src.rows, src.cols, NCH}; // size_t size = src.total() * NCH; // cv::Mat xt::xtensor XT res = xt::adapt((T*) src.data, size, xt::no_ownership(), shape); return res; } // JSON strokes parse_json(const std::string& x) { auto j = json::parse(x); // if (!j.is_array()) { throw std::runtime_error("'x' must be JSON array."); } strokes res; res.reserve(j.size()); for (const auto& a: j) { // 2- if (!a.is_array() || a.size() != 2) { throw std::runtime_error("'x' must include only 2d arrays."); } // auto p = a.get<points>(); res.push_back(p); } return res; } // // HSV cv::Mat ocv_draw_lines(const strokes& x, bool color = true) { // auto stype = color ? CV_8UC3 : CV_8UC1; // auto dtype = color ? CV_32FC3 : CV_32FC1; auto bg = color ? cv::Scalar(0, 0, 255) : cv::Scalar(255); auto col = color ? cv::Scalar(0, 255, 220) : cv::Scalar(0); cv::Mat img = cv::Mat(SIZE, SIZE, stype, bg); // size_t n = x.size(); for (const auto& s: x) { // size_t n_points = s.shape()[1]; for (size_t i = 0; i < n_points - 1; ++i) { // cv::Point from(s(0, i), s(1, i)); // cv::Point to(s(0, i + 1), s(1, i + 1)); // cv::line(img, from, to, col, LINE_WIDTH, LINE_TYPE); } if (color) { // col[0] += 180 / n; } } if (color) { // RGB cv::cvtColor(img, img, cv::COLOR_HSV2RGB); } // float32 [0, 1] img.convertTo(img, dtype, 1 / 255.0); return img; } // JSON xtensor3d process(const std::string& x, double scale = 1.0, bool color = true) { auto p = parse_json(x); auto img = ocv_draw_lines(p, color); if (scale != 1) { cv::Mat out; cv::resize(img, out, cv::Size(), scale, scale, RESIZE_TYPE); cv::swap(img, out); out.release(); } xtensor3d arr = color ? to_xt<double,3>(img) : to_xt<double,1>(img); return arr; } // [[Rcpp::export]] rtensor3d cpp_process_json_str(const std::string& x, double scale = 1.0, bool color = true) { xtensor3d res = process(x, scale, color); return res; } // [[Rcpp::export]] rtensor4d cpp_process_json_vector(const std::vector<std::string>& x, double scale = 1.0, bool color = false) { size_t n = x.size(); size_t dim = floor(SIZE * scale); size_t channels = color ? 3 : 1; xtensor4d res({n, dim, dim, channels}); parallelFor(0, n, [&x, &res, scale, color](int i) { xtensor3d tmp = process(x[i], scale, color); auto view = xt::view(res, i, xt::all(), xt::all(), xt::all()); view = tmp; }); return res; }
Dieser Code sollte in die src/cv_xt.cpp und mit dem Befehl Rcpp::sourceCpp(file = "src/cv_xt.cpp", env = .GlobalEnv) . Sie benötigen außerdem nlohmann/json.hpp aus dem Repository, um zu funktionieren . Der Code ist in mehrere Funktionen unterteilt:
to_xt - eine Vorlagenfunktion zum Konvertieren der to_xt ( cv::Mat ) in den Tensor xt::xtensor ;parse_json - Die Funktion analysiert eine JSON-Zeichenfolge, extrahiert die Koordinaten von Punkten und packt sie in einen Vektor.ocv_draw_lines - ocv_draw_lines mehrfarbige Linien aus dem empfangenen ocv_draw_lines .process - kombiniert die oben genannten Funktionen und bietet außerdem die Möglichkeit, das resultierende Bild zu skalieren.cpp_process_json_str - ein Wrapper über die process , der das Ergebnis in ein R-Objekt (mehrdimensionales Array) exportiert;cpp_process_json_vector - Ein Wrapper über die Funktion cpp_process_json_str , mit dem Sie einen Zeichenfolgenvektor im Multithread-Modus verarbeiten können.
Zum Zeichnen mehrfarbiger Linien wurde das HSV-Farbmodell verwendet, gefolgt von der Konvertierung in RGB. Testen Sie das Ergebnis:
arr <- cpp_process_json_str(tmp_data[4, drawing]) dim(arr) # [1] 256 256 3 plot(magick::image_read(arr))

Vergleich der Implementierungsgeschwindigkeit in R und C ++ res_bench <- bench::mark( r_process_json_str(tmp_data[4, drawing], scale = 0.5), cpp_process_json_str(tmp_data[4, drawing], scale = 0.5), check = FALSE, min_iterations = 100 ) # cols <- c("expression", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # expression min median max `itr/sec` total_time n_itr # <chr> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 r_process_json_str 3.49ms 3.55ms 4.47ms 273. 490ms 134 # 2 cpp_process_json_str 1.94ms 2.02ms 5.32ms 489. 497ms 243 library(ggplot2) # res_bench <- bench::press( batch_size = 2^(4:10), { .data <- tmp_data[sample(seq_len(.N), batch_size), drawing] bench::mark( r_process_json_vector(.data, scale = 0.5), cpp_process_json_vector(.data, scale = 0.5), min_iterations = 50, check = FALSE ) } ) res_bench[, cols] # expression batch_size min median max `itr/sec` total_time n_itr # <chr> <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 r 16 50.61ms 53.34ms 54.82ms 19.1 471.13ms 9 # 2 cpp 16 4.46ms 5.39ms 7.78ms 192. 474.09ms 91 # 3 r 32 105.7ms 109.74ms 212.26ms 7.69 6.5s 50 # 4 cpp 32 7.76ms 10.97ms 15.23ms 95.6 522.78ms 50 # 5 r 64 211.41ms 226.18ms 332.65ms 3.85 12.99s 50 # 6 cpp 64 25.09ms 27.34ms 32.04ms 36.0 1.39s 50 # 7 r 128 534.5ms 627.92ms 659.08ms 1.61 31.03s 50 # 8 cpp 128 56.37ms 58.46ms 66.03ms 16.9 2.95s 50 # 9 r 256 1.15s 1.18s 1.29s 0.851 58.78s 50 # 10 cpp 256 114.97ms 117.39ms 130.09ms 8.45 5.92s 50 # 11 r 512 2.09s 2.15s 2.32s 0.463 1.8m 50 # 12 cpp 512 230.81ms 235.6ms 261.99ms 4.18 11.97s 50 # 13 r 1024 4s 4.22s 4.4s 0.238 3.5m 50 # 14 cpp 1024 410.48ms 431.43ms 462.44ms 2.33 21.45s 50 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = expression, color = expression)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() + scale_color_discrete(name = "", labels = c("cpp", "r")) + theme(legend.position = "bottom")
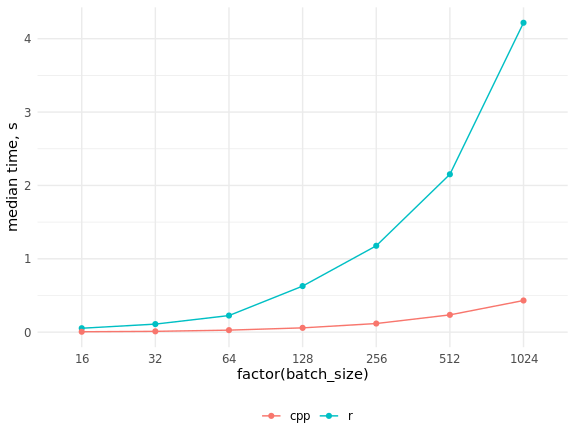
Wie Sie sehen können, stellte sich heraus, dass die Geschwindigkeitssteigerung sehr bedeutend war und es nicht möglich ist, C ++ - Code durch Parallelisieren von R-Code einzuholen.
3. Iteratoren zum Entladen von Stapeln aus der Datenbank
R hat einen wohlverdienten Ruf als Sprache für die Verarbeitung von Daten im RAM, während Python eher durch iterative Datenverarbeitung gekennzeichnet ist, die es einfach und unkompliziert macht, Berechnungen außerhalb des Kerns (Berechnungen mit externem Speicher) durchzuführen. Ein Beispiel für solche Berechnungen sind klassische und für uns im Zusammenhang mit dem beschriebenen Problem relevante tiefe neuronale Netze, die nach der Methode des Gradientenabfalls mit Annäherung des Gradienten bei jedem Schritt durch einen kleinen Teil der Beobachtungen oder einer Mini-Charge trainiert werden.
In Python geschriebene Deep-Learning-Frameworks verfügen über spezielle Klassen, die Iteratoren basierend auf Daten implementieren: Tabellen, Bilder in Ordnern, Binärformaten usw. Sie können vorgefertigte Optionen verwenden oder eigene für bestimmte Aufgaben schreiben. In R können wir die Keras Python-Bibliothek mit ihren verschiedenen Backends mit dem gleichnamigen Paket voll ausnutzen, das wiederum über dem Reticulate- Paket funktioniert. Letzteres verdient einen separaten großen Artikel; Sie können damit nicht nur Python-Code von R aus ausführen, sondern auch Objekte zwischen R- und Python-Sitzungen übertragen und automatisch alle erforderlichen Typkonvertierungen durchführen.
Wir haben die Notwendigkeit beseitigt, alle Daten im RAM zu speichern, da MonetDBLite verwendet wird. Die gesamte Arbeit im „neuronalen Netzwerk“ wird vom ursprünglichen Python-Code ausgeführt. Wir müssen lediglich einen Iterator basierend auf den Daten schreiben, da weder für R noch für Python eine solche Situation bereit ist. : ( R ). R numpy-, keras .
:
train_generator <- function(db_connection = con, samples_index, num_classes = 340, batch_size = 32, scale = 1, color = FALSE, imagenet_preproc = FALSE) { # checkmate::assert_class(con, "DBIConnection") checkmate::assert_integerish(samples_index) checkmate::assert_count(num_classes) checkmate::assert_count(batch_size) checkmate::assert_number(scale, lower = 0.001, upper = 5) checkmate::assert_flag(color) checkmate::assert_flag(imagenet_preproc) # , dt <- data.table::data.table(id = sample(samples_index)) # dt[, batch := (.I - 1L) %/% batch_size + 1L] # dt <- dt[, if (.N == batch_size) .SD, keyby = batch] # i <- 1 # max_i <- dt[, max(batch)] # sql <- sprintf( "PREPARE SELECT drawing, label_int FROM doodles WHERE id IN (%s)", paste(rep("?", batch_size), collapse = ",") ) res <- DBI::dbSendQuery(con, sql) # keras::to_categorical to_categorical <- function(x, num) { n <- length(x) m <- numeric(n * num) m[x * n + seq_len(n)] <- 1 dim(m) <- c(n, num) return(m) } # function() { # if (i > max_i) { dt[, id := sample(id)] data.table::setkey(dt, batch) # i <<- 1 max_i <<- dt[, max(batch)] } # ID batch_ind <- dt[batch == i, id] # batch <- DBI::dbFetch(DBI::dbBind(res, as.list(batch_ind)), n = -1) # i <<- i + 1 # JSON batch_x <- cpp_process_json_vector(batch$drawing, scale = scale, color = color) if (imagenet_preproc) { # c [0, 1] [-1, 1] batch_x <- (batch_x - 0.5) * 2 } batch_y <- to_categorical(batch$label_int, num_classes) result <- list(batch_x, batch_y) return(result) } }
, , , , ( scale = 1 256256 , scale = 0.5 — 128128 ), ( color = FALSE , color = TRUE ) , imagenet-. , [0, 1] [-1, 1], keras .
, data.table samples_index , , SQL- . keras::to_categorical() . , , steps_per_epoch keras::fit_generator() , if (i > max_i) .
, , JSON- ( cpp_process_json_vector() , C++) , . one-hot , , . data.table — "" data.table - R.
Core i5 :
library(Rcpp) library(keras) library(ggplot2) source("utils/rcpp.R") source("utils/keras_iterator.R") con <- DBI::dbConnect(drv = MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR")) ind <- seq_len(DBI::dbGetQuery(con, "SELECT count(*) FROM doodles")[[1L]]) num_classes <- DBI::dbGetQuery(con, "SELECT max(label_int) + 1 FROM doodles")[[1L]] # train_ind <- sample(ind, floor(length(ind) * 0.995)) # val_ind <- ind[-train_ind] rm(ind) # scale <- 0.5 # res_bench <- bench::press( batch_size = 2^(4:10), { it1 <- train_generator( db_connection = con, samples_index = train_ind, num_classes = num_classes, batch_size = batch_size, scale = scale ) bench::mark( it1(), min_iterations = 50L ) } ) # cols <- c("batch_size", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # batch_size min median max `itr/sec` total_time n_itr # <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 16 25ms 64.36ms 92.2ms 15.9 3.09s 49 # 2 32 48.4ms 118.13ms 197.24ms 8.17 5.88s 48 # 3 64 69.3ms 117.93ms 181.14ms 8.57 5.83s 50 # 4 128 157.2ms 240.74ms 503.87ms 3.85 12.71s 49 # 5 256 359.3ms 613.52ms 988.73ms 1.54 30.5s 47 # 6 512 884.7ms 1.53s 2.07s 0.674 1.11m 45 # 7 1024 2.7s 3.83s 5.47s 0.261 2.81m 44 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = 1)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() DBI::dbDisconnect(con, shutdown = TRUE)

, ( 32 ). /dev/shm , . , /etc/fstab , tmpfs /dev/shm tmpfs defaults,size=25g 0 0 . , df -h .
, :
test_generator <- function(dt, batch_size = 32, scale = 1, color = FALSE, imagenet_preproc = FALSE) { # checkmate::assert_data_table(dt) checkmate::assert_count(batch_size) checkmate::assert_number(scale, lower = 0.001, upper = 5) checkmate::assert_flag(color) checkmate::assert_flag(imagenet_preproc) # dt[, batch := (.I - 1L) %/% batch_size + 1L] data.table::setkey(dt, batch) i <- 1 max_i <- dt[, max(batch)] # function() { batch_x <- cpp_process_json_vector(dt[batch == i, drawing], scale = scale, color = color) if (imagenet_preproc) { # c [0, 1] [-1, 1] batch_x <- (batch_x - 0.5) * 2 } result <- list(batch_x) i <<- i + 1 return(result) } }
4.
mobilenet v1 , . keras , , R. : (batch, height, width, 3) , . Python , , ( , keras- ):
mobilenet v1 library(keras) top_3_categorical_accuracy <- custom_metric( name = "top_3_categorical_accuracy", metric_fn = function(y_true, y_pred) { metric_top_k_categorical_accuracy(y_true, y_pred, k = 3) } ) layer_sep_conv_bn <- function(object, filters, alpha = 1, depth_multiplier = 1, strides = c(2, 2)) { # NB! depth_multiplier != resolution multiplier # https://github.com/keras-team/keras/issues/10349 layer_depthwise_conv_2d( object = object, kernel_size = c(3, 3), strides = strides, padding = "same", depth_multiplier = depth_multiplier ) %>% layer_batch_normalization() %>% layer_activation_relu() %>% layer_conv_2d( filters = filters * alpha, kernel_size = c(1, 1), strides = c(1, 1) ) %>% layer_batch_normalization() %>% layer_activation_relu() } get_mobilenet_v1 <- function(input_shape = c(224, 224, 1), num_classes = 340, alpha = 1, depth_multiplier = 1, optimizer = optimizer_adam(lr = 0.002), loss = "categorical_crossentropy", metrics = c("categorical_crossentropy", top_3_categorical_accuracy)) { inputs <- layer_input(shape = input_shape) outputs <- inputs %>% layer_conv_2d(filters = 32, kernel_size = c(3, 3), strides = c(2, 2), padding = "same") %>% layer_batch_normalization() %>% layer_activation_relu() %>% layer_sep_conv_bn(filters = 64, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 128, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 128, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 256, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 256, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 1024, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 1024, strides = c(1, 1)) %>% layer_global_average_pooling_2d() %>% layer_dense(units = num_classes) %>% layer_activation_softmax() model <- keras_model( inputs = inputs, outputs = outputs ) model %>% compile( optimizer = optimizer, loss = loss, metrics = metrics ) return(model) }
. , , , . , imagenet-. , . get_config() ( base_model_conf$layers — R- ), from_config() :
base_model_conf <- get_config(base_model) base_model_conf$layers[[1]]$config$batch_input_shape[[4]] <- 1L base_model <- from_config(base_model_conf)
keras imagenet- :
get_model <- function(name = "mobilenet_v2", input_shape = NULL, weights = "imagenet", pooling = "avg", num_classes = NULL, optimizer = keras::optimizer_adam(lr = 0.002), loss = "categorical_crossentropy", metrics = NULL, color = TRUE, compile = FALSE) { # checkmate::assert_string(name) checkmate::assert_integerish(input_shape, lower = 1, upper = 256, len = 3) checkmate::assert_count(num_classes) checkmate::assert_flag(color) checkmate::assert_flag(compile) # keras model_fun <- get0(paste0("application_", name), envir = asNamespace("keras")) # if (is.null(model_fun)) { stop("Model ", shQuote(name), " not found.", call. = FALSE) } base_model <- model_fun( input_shape = input_shape, include_top = FALSE, weights = weights, pooling = pooling ) # , if (!color) { base_model_conf <- keras::get_config(base_model) base_model_conf$layers[[1]]$config$batch_input_shape[[4]] <- 1L base_model <- keras::from_config(base_model_conf) } predictions <- keras::get_layer(base_model, "global_average_pooling2d_1")$output predictions <- keras::layer_dense(predictions, units = num_classes, activation = "softmax") model <- keras::keras_model( inputs = base_model$input, outputs = predictions ) if (compile) { keras::compile( object = model, optimizer = optimizer, loss = loss, metrics = metrics ) } return(model) }
. : get_weights() R- , ( - ), set_weights() . , , .
mobilenet 1 2, resnet34. , SE-ResNeXt. , , ( ).
5.
, docopt :
doc <- ' Usage: train_nn.R --help train_nn.R --list-models train_nn.R [options] Options: -h --help Show this message. -l --list-models List available models. -m --model=<model> Neural network model name [default: mobilenet_v2]. -b --batch-size=<size> Batch size [default: 32]. -s --scale-factor=<ratio> Scale factor [default: 0.5]. -c --color Use color lines [default: FALSE]. -d --db-dir=<path> Path to database directory [default: Sys.getenv("db_dir")]. -r --validate-ratio=<ratio> Validate sample ratio [default: 0.995]. -n --n-gpu=<number> Number of GPUs [default: 1]. ' args <- docopt::docopt(doc)
docopt http://docopt.org/ R. Rscript bin/train_nn.R -m resnet50 -c -d /home/andrey/doodle_db ./bin/train_nn.R -m resnet50 -c -d /home/andrey/doodle_db , train_nn.R ( resnet50 128128 , /home/andrey/doodle_db ). , . , mobilenet_v2 keras R - R- — , .
RStudio ( tfruns ). , RStudio.
6.
. R- .
« », . , NVIDIA, CUDA+cuDNN — , tensorflow/tensorflow:1.12.0-gpu , R-.
- :
Dockerfile FROM tensorflow/tensorflow:1.12.0-gpu MAINTAINER Artem Klevtsov <aaklevtsov@gmail.com> SHELL ["/bin/bash", "-c"] ARG LOCALE="en_US.UTF-8" ARG APT_PKG="libopencv-dev r-base r-base-dev littler" ARG R_BIN_PKG="futile.logger checkmate data.table rcpp rapidjsonr dbi keras jsonlite curl digest remotes" ARG R_SRC_PKG="xtensor RcppThread docopt MonetDBLite" ARG PY_PIP_PKG="keras" ARG DIRS="/db /app /app/data /app/models /app/logs" RUN source /etc/os-release && \ echo "deb https://cloud.r-project.org/bin/linux/ubuntu ${UBUNTU_CODENAME}-cran35/" > /etc/apt/sources.list.d/cran35.list && \ apt-key adv --keyserver keyserver.ubuntu.com --recv-keys E084DAB9 && \ add-apt-repository -y ppa:marutter/c2d4u3.5 && \ add-apt-repository -y ppa:timsc/opencv-3.4 && \ apt-get update && \ apt-get install -y locales && \ locale-gen ${LOCALE} && \ apt-get install -y --no-install-recommends ${APT_PKG} && \ ln -s /usr/lib/R/site-library/littler/examples/install.r /usr/local/bin/install.r && \ ln -s /usr/lib/R/site-library/littler/examples/install2.r /usr/local/bin/install2.r && \ ln -s /usr/lib/R/site-library/littler/examples/installGithub.r /usr/local/bin/installGithub.r && \ echo 'options(Ncpus = parallel::detectCores())' >> /etc/R/Rprofile.site && \ echo 'options(repos = c(CRAN = "https://cloud.r-project.org"))' >> /etc/R/Rprofile.site && \ apt-get install -y $(printf "r-cran-%s " ${R_BIN_PKG}) && \ install.r ${R_SRC_PKG} && \ pip install ${PY_PIP_PKG} && \ mkdir -p ${DIRS} && \ chmod 777 ${DIRS} && \ rm -rf /tmp/downloaded_packages/ /tmp/*.rds && \ rm -rf /var/lib/apt/lists/* COPY utils /app/utils COPY src /app/src COPY tests /app/tests COPY bin/*.R /app/ ENV DBDIR="/db" ENV CUDA_HOME="/usr/local/cuda" ENV PATH="/app:${PATH}" WORKDIR /app VOLUME /db VOLUME /app CMD bash
; . /bin/bash /etc/os-release . .
-, . , , , :
#!/bin/sh DBDIR=${PWD}/db LOGSDIR=${PWD}/logs MODELDIR=${PWD}/models DATADIR=${PWD}/data ARGS="--runtime=nvidia --rm -v ${DBDIR}:/db -v ${LOGSDIR}:/app/logs -v ${MODELDIR}:/app/models -v ${DATADIR}:/app/data" if [ -z "$1" ]; then CMD="Rscript /app/train_nn.R" elif [ "$1" = "bash" ]; then ARGS="${ARGS} -ti" else CMD="Rscript /app/train_nn.R $@" fi docker run ${ARGS} doodles-tf ${CMD}
- , train_nn.R ; — "bash", . : CMD="Rscript /app/train_nn.R $@" .
, , , .
7. GPU Google Cloud
(. , @Leigh.plt ODS-). , 1 GPU GPU . GoogleCloud ( ) - , $300. 4V100 SSD , . , . K80. — SSD c, dev/shm .
, GPU. CPU , :
with(tensorflow::tf$device("/cpu:0"), { model_cpu <- get_model( name = model_name, input_shape = input_shape, weights = weights, metrics =(top_3_categorical_accuracy, compile = FALSE ) })
( ) GPU, :
model <- keras::multi_gpu_model(model_cpu, gpus = n_gpu) keras::compile( object = model, optimizer = keras::optimizer_adam(lr = 0.0004), loss = "categorical_crossentropy", metrics = c(top_3_categorical_accuracy) )
, , , GPU .
tensorboard , :
# log_file_tmpl <- file.path("logs", sprintf( "%s_%d_%dch_%s.csv", model_name, dim_size, channels, format(Sys.time(), "%Y%m%d%H%M%OS") )) # model_file_tmpl <- file.path("models", sprintf( "%s_%d_%dch_{epoch:02d}_{val_loss:.2f}.h5", model_name, dim_size, channels )) callbacks_list <- list( keras::callback_csv_logger( filename = log_file_tmpl ), keras::callback_early_stopping( monitor = "val_loss", min_delta = 1e-4, patience = 8, verbose = 1, mode = "min" ), keras::callback_reduce_lr_on_plateau( monitor = "val_loss", factor = 0.5, # lr 2 patience = 4, verbose = 1, min_delta = 1e-4, mode = "min" ), keras::callback_model_checkpoint( filepath = model_file_tmpl, monitor = "val_loss", save_best_only = FALSE, save_weights_only = FALSE, mode = "min" ) )
8.
, , :
- keras (
lr_finder fast.ai ); , R , , ; - , GPU;
- , imagenet-;
- one cycle policy discriminative learning rates (osine annealing , skeydan ).
:
- ( ) . data.table in-place , , . .
- R C++ Rcpp . RcppThread RcppParallel , , R .
- Rcpp C++, . xtensor CRAN, , R C++. — ++ RStudio.
- docopt . , .. . RStudio , IDE .
- , . .
- Google Cloud — , .
- , R C++, bench — .
, .