 Experimentelles Protokoll und Implementierung eines Sortieralgorithmus auf einem programmierbaren DNA-Computer
Experimentelles Protokoll und Implementierung eines Sortieralgorithmus auf einem programmierbaren DNA-ComputerWissenschaftler experimentieren seit langem damit, Informationen in DNA zu speichern und diese Informationen zu verarbeiten. Beispielsweise haben Wissenschaftler der Washington University und von Microsoft kürzlich das
"weltweit erste DNA Winchester" gebaut (
Foto ). Dieses Design ist zum ersten Mal in der Lage, Informationen in der DNA-Speicherung ohne menschliches Eingreifen aufzuzeichnen und zu lesen. Eine sehr bedeutende Leistung, da DNA Informationen mit einer Dichte von
2,2 Petabyte pro Gramm aufzeichnen kann. DNA ist ein kompakter Behälter mit einer Aufzeichnungsdichte, die tausendfach größer ist als die vorhandenen Träger.
Alle vorhandenen DNA-Systeme haben jedoch ein Problem: All dies sind einzigartige proprietäre Entwicklungen, denen jegliche Flexibilität fehlt. Wenn wir mit der Siliziumtechnologie vergleichen, entwickelt jede Gruppe von Forschern von Grund auf eine neue Computerarchitektur, für die Sie neue Software schreiben müssen. Dank des
ersten programmierbaren DNA-Computers, der an der UC Davis, dem California Institute of Technology und der University of Maynooth entwickelt wurde, können sich die Dinge ändern.
Der erste programmierbare DNA-Computer wird in einem
wissenschaftlichen Artikel beschrieben, der am 20. März 2019 in der Zeitschrift Nature veröffentlicht wurde. Die Autoren zeigten, dass mit Hilfe eines einfachen Auslösers derselbe Grundsatz von DNA-Molekülen viele verschiedene Algorithmen implementieren kann. Obwohl es sich bei der Studie um ein reines Laborexperiment handelt, können in Zukunft programmierbare molekulare Algorithmen verwendet werden, um beispielsweise DNA-Roboter zu programmieren, die bereits erfolgreich
Medikamente an Krebszellen abgeben .
„Dies ist eines der wegweisenden Werke in diesem Bereich“,
sagt Torsten-Lars Schmidt, Assistenzprofessor für experimentelle Biophysik an der University of Kent, der nicht an der Forschung beteiligt war. "Früher haben sie die algorithmische Selbstorganisation demonstriert, aber nicht so komplex."
In elektronischen Computern sind Bits binäre Informationseinheiten. Sie repräsentieren den diskreten physikalischen Zustand der Grundausstattung, beispielsweise das Vorhandensein oder Fehlen eines elektrischen Stroms. Diese Bits bzw. elektrischen Signale werden durch Schaltungen geleitet, die aus Logikelementen bestehen, die eine Operation an einem oder mehreren Eingangsbits ausführen und ein Bit als Ausgang erzeugen.
Computer, die diese einfachen Bausteine immer wieder kombinieren, können erstaunlich komplexe Programme ausführen. Die Idee des DNA-Computing besteht darin, elektrische Signale durch chemische Bindungen und Silizium durch Nukleinsäuren zu ersetzen, um biomolekulare Software zu erstellen.
 Abstrakte Architekturhierarchie und praktische Implementierung der vollständigen 6-Bit-IBC-Logikschaltung (Iterated Boolean Circuit)
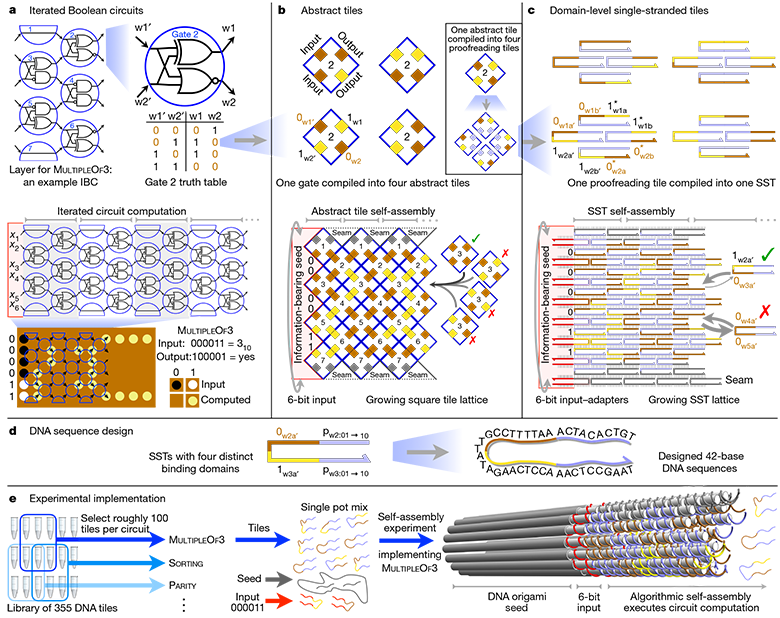
Abstrakte Architekturhierarchie und praktische Implementierung der vollständigen 6-Bit-IBC-Logikschaltung (Iterated Boolean Circuit)Laut Eric Winfrey, Wissenschaftler am California Institute of Technology und Mitautor des Artikels, nutzen molekulare Algorithmen die natürlichen Möglichkeiten der Informationsverarbeitung in DNA, aber anstatt der Natur zu erlauben, die Zügel selbst in die Hand zu nehmen, werden die Berechnungen in DNA nach einem vom Menschen geschriebenen Programm durchgeführt.
In den letzten 20 Jahren wurden mehrere erfolgreiche Experimente mit molekularen Algorithmen durchgeführt, beispielsweise zum Spielen von Tic-Tac-Toe oder zum Zusammensetzen von Molekülen verschiedener Formen. In jedem Fall war eine sorgfältige Entwicklung der DNA-Sequenz erforderlich, um einen spezifischen Algorithmus auszuführen, der die DNA-Struktur erzeugen würde. In diesem Fall besteht der Unterschied darin, dass die Forscher ein System entwickelt haben, in dem dieselben grundlegenden DNA-Fragmente angeordnet werden können, um
völlig unterschiedliche Algorithmen zu erstellen - und damit völlig unterschiedliche Ergebnisse zu erzielen.
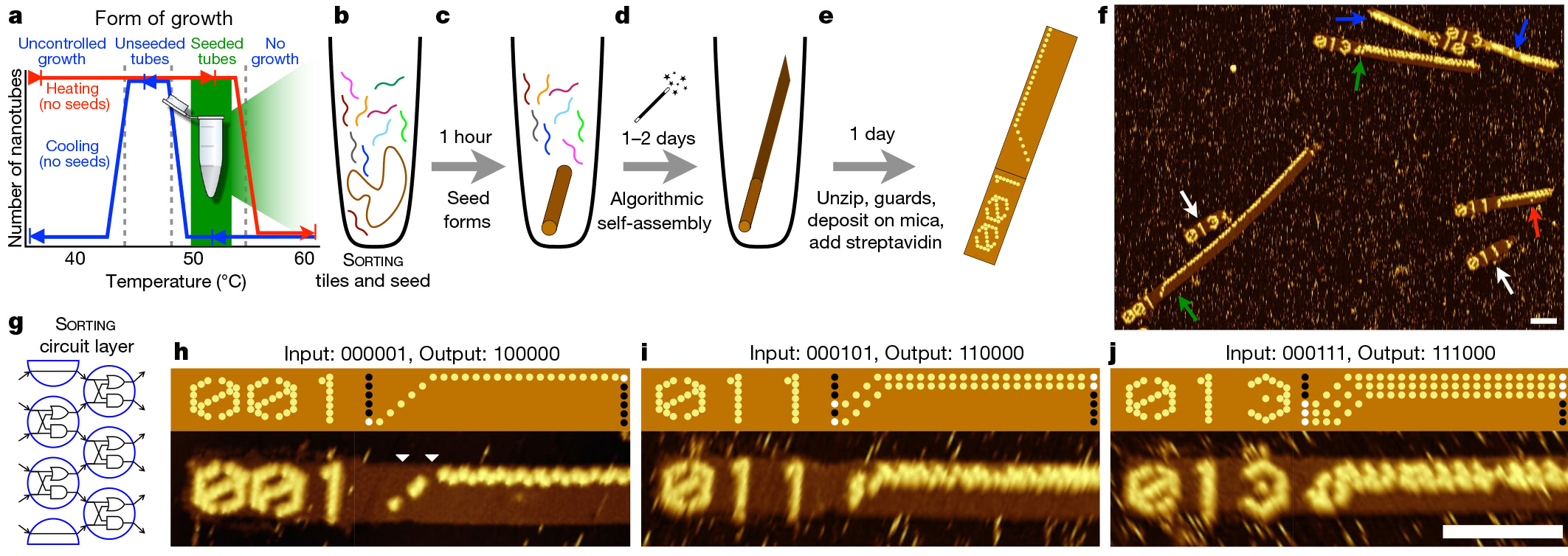
Der Prozess beginnt mit der DNA-Origami-Technik, dh dem Falten einer langen DNA-Kette in die gewünschte Form. Diese gefaltete Scheibe funktioniert wie ein Startwert, der eine algorithmische Montagelinie ausführt. Das Saatgut bleibt unabhängig vom Algorithmus praktisch unverändert. Für jedes Experiment werden nur kleine Änderungen in mehreren Sequenzen vorgenommen.
 Neuprogrammierung der Logikschaltung
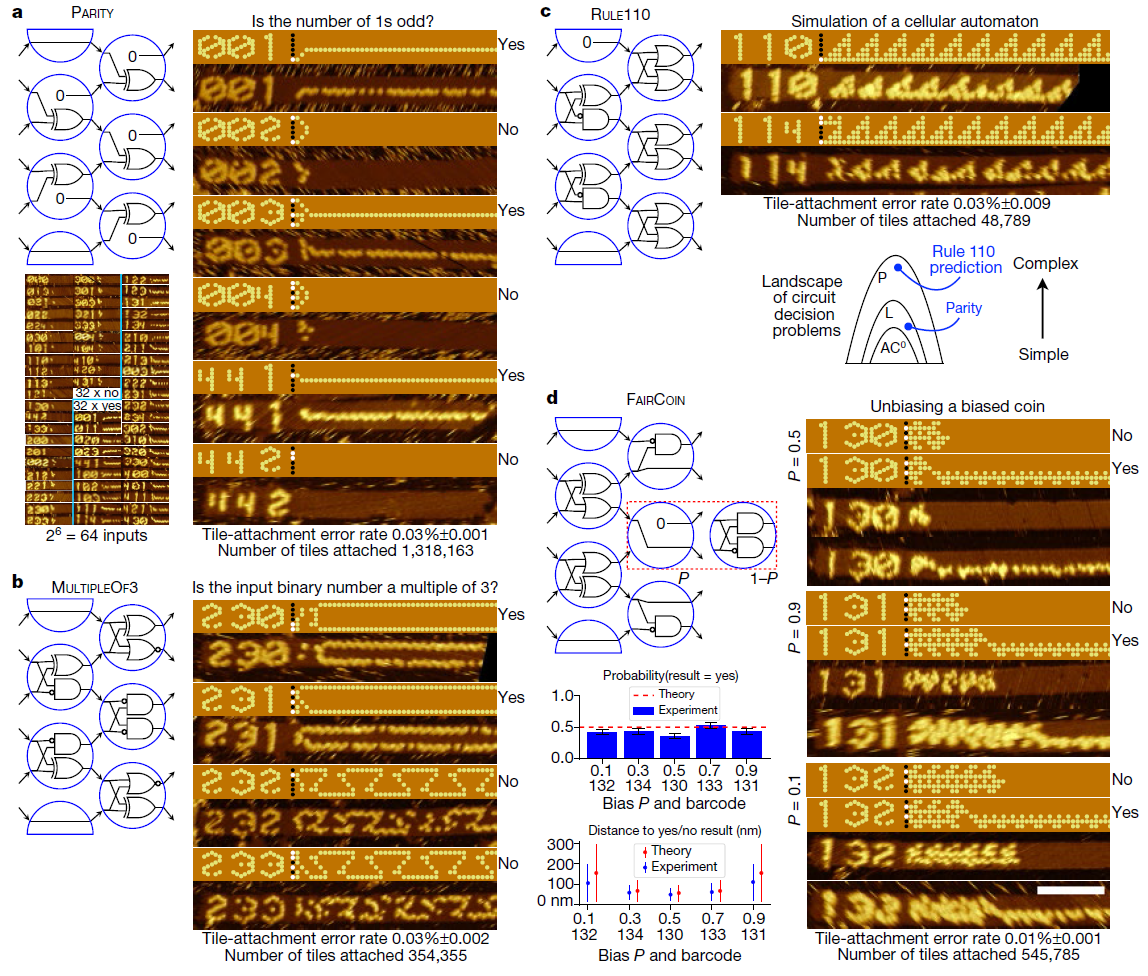
Neuprogrammierung der LogikschaltungNach dem Erstellen des „Samens“ wird es mit Hunderten anderer DNA-Stränge, die als DNA-Kacheln bekannt sind, zur Lösung hinzugefügt. Wissenschaftler haben 355 dieser Kacheln entwickelt. Jedes hat eine einzigartige Anordnung von stickstoffhaltigen Basen. Dementsprechend wählen die Forscher für jeden Algorithmus einfach einen anderen Satz von Startkacheln aus. Da diese DNA-Fragmente während des Assemblierungsprozesses verbunden werden, bilden sie eine Schaltung, die den ausgewählten molekularen Algorithmus auf den vom "Keim" bereitgestellten Eingabebits implementiert.
Mit diesem System entwickelten und testeten die Forscher 21 Algorithmen, um Aufgaben wie das Erkennen einer Division durch drei, das
Auswählen eines Leiters , das Generieren von Mustern und das Zählen von 0 bis 63 auszuführen. Alle diese Algorithmen werden unter Verwendung verschiedener Kombinationen derselben 355 DNA-Kacheln implementiert.
Natürlich ist es nicht einfach, Code zu schreiben, indem man DNA-Fragmente in ein Reagenzglas fallen lässt, aber wenn der Prozess automatisiert ist, müssen zukünftige molekulare Programmierer nicht einmal über Biomechanik nachdenken, da heutige Programmierer die Physik von Transistoren nicht verstehen müssen, um gute Programme zu schreiben.