Hallo nochmal! Wir teilen eine Publikation, deren Übersetzung speziell für Studenten des Kurses
"Neuronale Netze in Python" erstellt wurde .

Heute werden wir über das erste wichtige Ereignis in der Geschichte der Entwicklung von DeepMind sprechen, um zu zeigen, wie Forschung mit künstlicher Intelligenz die Entstehung wissenschaftlicher Entdeckungen stimulieren kann. Aufgrund des interdisziplinären Charakters unserer Arbeit brachte DeepMind Experten aus den Bereichen Strukturbiologie, Physik und maschinelles Lernen zusammen, um fortschrittliche Methoden zur Vorhersage der dreidimensionalen Struktur eines Proteins zu verwenden, die ausschließlich auf seiner genetischen Sequenz basiert.
Das AlphaFold-System, an dem wir in den letzten zwei Jahren gearbeitet haben, basiert auf langjähriger Forschungserfahrung, bei der umfangreiche Genomdaten zur Vorhersage der Proteinstruktur verwendet wurden. Die dreidimensionalen Proteinmodelle, die AlphaFold generiert, sind viel genauer als die zuvor erhaltenen. Dies war ein bedeutender Fortschritt in einer der Hauptaufgaben der Biologie.
Was ist das Problem der Proteinfaltung?
Proteine sind große und komplexe Moleküle, die zur Erhaltung des Lebens benötigt werden. Fast alle Funktionen unseres Körpers, sei es Muskelkontraktion, Lichtwahrnehmung oder die Umwandlung von Nahrung in Energie, können auf ein oder mehrere Proteine zurückgeführt werden und wie sie sich bewegen und verändern. Rezepte für diese Proteine, Gene genannt, sind in unserer DNA kodiert.
Die Eigenschaften eines Proteins hängen von seiner einzigartigen dreidimensionalen Struktur ab. Zum Beispiel sind die Antikörperproteine, aus denen unser Immunsystem besteht, „Y-förmig“ und sehen aus wie spezielle Haken. Antikörperproteine klammern sich an Viren und Bakterien und können Krankheitserreger für die anschließende Zerstörung nachweisen und markieren. In ähnlicher Weise liegen Kollagenproteine in Form von Schnüren vor, die Spannungen zwischen Knorpel, Bändern, Knochen und Haut übertragen. Andere Arten von Proteinen umfassen Cas9, das, geleitet von CRISPR-Sequenzen, als Schere fungiert, die DNA schneidet und neue Stellen einfügt. Frostschutzproteine, deren dreidimensionale Struktur es ihnen ermöglicht, sich an Eiskristalle zu binden und das Einfrieren von Organismen zu verhindern; und Ribosomen, die als programmierter Förderer fungieren, der an der Konstruktion von Proteinen beteiligt ist.
Die dreidimensionale Struktur eines Proteins ausschließlich anhand seiner genetischen Sequenz zu bestimmen, ist eine schwierige Aufgabe, mit der Wissenschaftler seit Jahrzehnten zu kämpfen haben. Das Problem ist, dass DNA nur Informationen über die Sequenz von Bausteinen eines Proteins enthält, die als Aminosäurereste bezeichnet werden und lange Ketten bilden. Die Vorhersage, wie diese Ketten eine komplexe 3D-Proteinstruktur bilden, wird als „Proteinfaltungsproblem“ bezeichnet.
Je größer das Protein ist, desto schwieriger ist es zu modellieren, da mehr Bindungen zwischen Aminosäuren gebildet werden, die berücksichtigt werden müssen. Wie aus
dem Levintal-Paradoxon folgt, wird es mehr Zeit dauern, als das Universum existiert, um alle möglichen Konfigurationen eines gewöhnlichen Proteins aufzulisten, bevor seine korrekte dreidimensionale Struktur erreicht ist.

Warum ist Proteinfaltung wichtig?
Die Fähigkeit, die Form eines Proteins vorherzusagen, ist äußerst nützlich, da sie für das Verständnis der Rolle des Proteins im Körper sowie für die Diagnose und Behandlung von Krankheiten wie Alzheimer,
Parkinson ,
Huntington und
Mukoviszidose , von denen Ärzte glauben, dass sie durch falsch gefaltete Proteine verursacht werden, von grundlegender Bedeutung ist.
Wir freuen uns besonders, dass die Fähigkeit, die Form eines Proteins vorherzusagen, unser Verständnis der Funktionsweise unseres Körpers verbessern kann und es uns ermöglicht, neue Medikamente effizient zu entwickeln. Wenn wir mehr Informationen über die Formen von Proteinen und deren Funktionsweise durch Modellierung erhalten, eröffnen sich neue Möglichkeiten für die Herstellung von Arzneimitteln und die Kosten für Experimente sinken. Letztendlich werden diese Entdeckungen die Lebensqualität von Millionen von Patienten weltweit verbessern.
Das Verständnis des Prozesses der Proteinfaltung kann auch bei der Entwicklung einer Proteinart helfen, die einen wesentlichen Beitrag zur umgebenden Realität leistet. Beispielsweise können Fortschritte bei der Entwicklung von Proteinen auf dem Gebiet der biologisch abbaubaren Enzyme dazu beitragen, mit Verunreinigungen wie Kunststoff und Öl umzugehen und Abfall abzubauen, ohne die Umwelt zu schädigen. Tatsächlich haben Forscher bereits damit begonnen
, Bakterien zu entwickeln, die Proteine absondern, die den Abfall biologisch abbaubar machen und seine Handhabung erleichtern.
Um die Forschung anzuregen und die Fortschritte auf dem Gebiet der neuesten Methoden zur Verbesserung der Prognosegenauigkeit zu bewerten, wurde 1994 ein zweijähriger Großwettbewerb namens CASP
Community Experiment zur kritischen Bewertung von Proteinstrukturmethoden (CASP)
gestartet , der zum Goldstandard für Bewertungsmethoden geworden ist.
Wie wird KI einen Unterschied machen?
In den letzten fünf Jahrzehnten konnten Wissenschaftler die Formen von Proteinen im Labor mithilfe experimenteller Methoden wie
Kryoelektronenmikroskopie ,
Kernspinresonanz oder
Röntgenbeugung erkennen . Jede Methode wurde jedoch durch viele Versuche und Fehler abgeleitet, die Jahre dauerten und Zehntausende von Dollar kosteten. Aus diesem Grund wenden sich Biologen jetzt AI-Methoden als Alternative zum langen und mühsamen Prozess der Erforschung komplexer Proteine zu.
Glücklicherweise verfügt das Gebiet der Genomik aufgrund der raschen Senkung der Kosten für die genetische Sequenzierung über genügend Daten. Infolgedessen sind in den letzten Jahren
Ansätze für das Problem der Vorhersage mithilfe von Deep Learning und basierend auf Genomdaten immer beliebter geworden. Die Arbeit von DeepMind zu diesem Thema führte zum Erscheinen von AlphaFold, das wir CASP in diesem Jahr vorgestellt haben. Wir sind stolz darauf, Teil des Fortschritts zu sein, den CASP-Experten als "beispiellosen Fortschritt bei der Fähigkeit von Berechnungsmethoden zur Vorhersage der Struktur eines Proteins" bezeichneten. Infolgedessen
belegten wir
den ersten Platz in der Rangliste der Teams (wir sind A7D).
Unser Team konzentrierte sich genau auf die Aufgabe, Zielformen von Grund auf neu zu modellieren, ohne zuvor gelöste Proteine als Vorlagen zu verwenden. Wir haben ein hohes Maß an Genauigkeit bei der Vorhersage der physikalischen Eigenschaften der Proteinstruktur erreicht und dann zwei verschiedene Methoden verwendet, um vollständige Proteinstrukturen vorherzusagen.
Verwendung neuronaler Netze zur Vorhersage physikalischer Eigenschaften
Beide Methoden verwendeten tiefe neuronale Netze, die darauf trainiert sind, die Eigenschaften eines Proteins anhand seiner genetischen Sequenz vorherzusagen. Die vom Netzwerk vorhergesagten Eigenschaften sind: (a) der Abstand zwischen Aminosäurepaaren und (b) die Winkel zwischen den chemischen Bindungen, die diese Aminosäuren verbinden. Die erste Entwicklung war ein echter Fortschritt bei der Verwendung gängiger Methoden, die bestimmen, ob Aminosäurepaare nebeneinander liegen.
Wir haben das neuronale Netzwerk trainiert, um eine separate Verteilung der Abstände zwischen jedem Paar von Proteinresten vorherzusagen. Diese Wahrscheinlichkeiten wurden dann zu einer Schätzung kombiniert, die zeigt, wie gut die Proteinstruktur gestaltet ist. Wir haben auch ein anderes neuronales Netzwerk trainiert, das alle Entfernungen insgesamt verwendet, um zu bewerten, wie nahe die vorgeschlagene Struktur an der richtigen Antwort liegt.

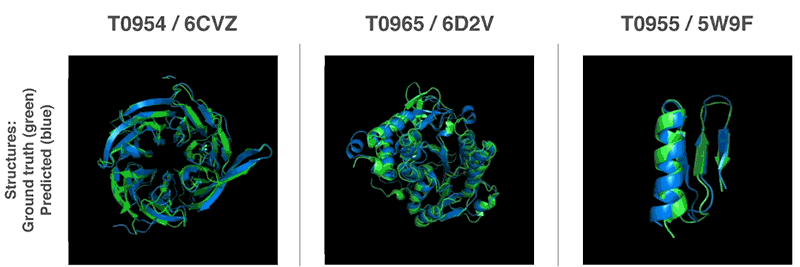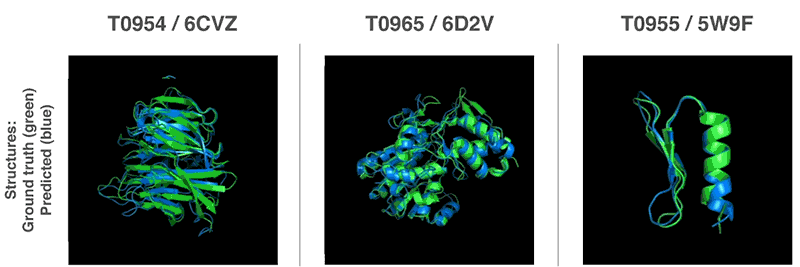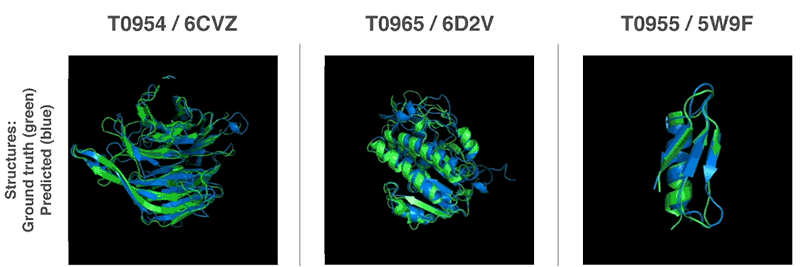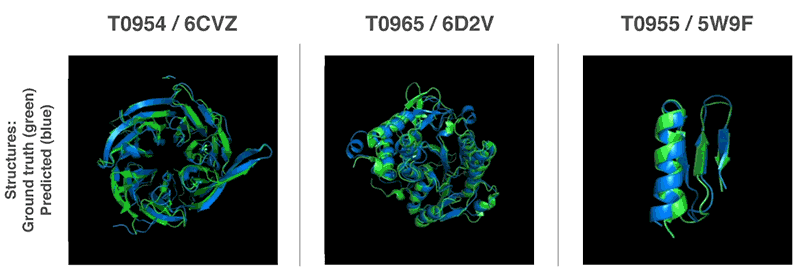
Neue Methoden zur Vorhersage von Proteinstrukturen
Mit diesen Bewertungsfunktionen konnten wir Strukturen finden, die unseren Prognosen entsprechen. Unsere erste Methode basiert auf Methoden, die in der Strukturbiologie weit verbreitet sind, und hat wiederholt Teile der Proteinstruktur durch neue Fragmente ersetzt. Wir haben das generativ-kompetitive neuronale Netzwerk trainiert, um neue Fragmente vorzuschlagen, mit denen die Bewertung der vorgeschlagenen Proteinstruktur kontinuierlich verbessert werden kann.

Die zweite Methode optimierte die Noten mithilfe des Gradientenabfalls (eine mathematische Methode, die üblicherweise beim maschinellen Lernen für kleine inkrementelle Verbesserungen verwendet wird), was zu einer hohen Genauigkeit der Strukturen führte. Diese Methode wurde auf ganze Proteinketten angewendet und nicht auf Teile, die vor dem Zusammenbau separat gestapelt werden müssen, was die Komplexität des Vorhersageprozesses verringert.
Was weiter?
Der Erfolg unseres Proteinkoagulationsstifttests zeigt, dass maschinelle Lernsysteme mehrere Informationsquellen integrieren können, um Wissenschaftlern dabei zu helfen, schnell kreative Lösungen für komplexe Probleme zu entwickeln. Wir haben bereits gesehen, wie KI Menschen hilft, komplexe Spiele durch Systeme wie
AlphaGo und
AlphaZero zu meistern . Wir hoffen auch, dass der Durchbruch der KI der Menschheit hilft, grundlegende wissenschaftliche Probleme zu lösen.
Es ist interessant, die ersten Fortschritte bei der Proteinfaltung zu sehen, die die Nützlichkeit der KI für wissenschaftliche Entdeckungen demonstrieren. Obwohl wir noch viel zu tun haben, verstehen wir klar, dass wir in der Lage sein werden, zur Suche nach der Behandlung verschiedener Krankheiten beizutragen, der Umwelt zu helfen und vieles mehr, denn tatsächlich ist das Potenzial riesig. Mit einem engagierten Team, das sich darauf konzentriert, wie maschinelles Lernen die Welt der Wissenschaft voranbringen kann, werden wir die verschiedenen Möglichkeiten und Methoden untersuchen, mit denen unsere Technologie die Welt um uns herum beeinflussen kann.