Bevor Sie dieses Upgrade lesen, sollten Sie sich mit dem zuvor veröffentlichten Quellcode vertraut machen.Der allererste und beliebteste Nanion-Poren-Sequenzer MinION (in Großbritannien mindestens 1000 US-Dollar, in Russland 150.000 Rubel), der von Oxford Nanopore Technologies (ONT) entwickelt wurde, arbeitet mit Einwegzellen, die jeweils 900 US-Dollar kosten Russland - 135 Tausend Rubel). Mit solchen Zellen können Sie DNA 10 ... 20 Gb in 2 ... 3 Tagen digitalisieren. Dies ist zu wenig für die Sequenzierung des menschlichen Genoms (Sie benötigen> 100 GB), aber zu viel für alle anderen klinischen Diagnosezwecke (<1 GB ist ausreichend).
Vor kurzem wurde mit dem Verkauf produktiverer (und teurerer) Modelle begonnen - PromethION 24/48 (165.000 USD / 285.000 USD), das hauptsächlich auf die Inline-Sequenzierung des gesamten Genoms abzielt. Teurer sind die effizienteren Verbrauchsmaterialien (2.000 USD / Einheit,> 100 GB). Zwar können sie in großen Mengen billiger sein (1.800.000 USD für 2.880 Stück, 625 USD / Stück), aber solche Akquisitionen (200 ... 250 Millionen Rubel pro Charge) sind für das russische Budget nicht zulässig.
Und das nicht nur für Russisch. Daher wurde die größte Aufmerksamkeit der Besucher der jüngsten ONT-Konferenz (22.-24. Mai) durch die Ankündigung des Verkaufsstarts von Flongle erregt, einem Adaptereinsatz für den MinION-Sequenzer, der das Arbeiten mit weniger effizienten (~ 1 GB), aber relativ billigen (90 USD) Einwegzellen ermöglicht.
 https://nanoporetech.com/products
https://nanoporetech.com/productsDas Auftreten von Flongle kann zu einem explosionsartigen Anstieg des Einsatzes von NGS (Next Generation Sequencing) in der klinischen Praxis führen und die konventionelle PCR-Diagnostik verdrängen, deren Marktgröße in Russland eine Milliarde Rubel übersteigt und die weltweit in Milliarden Dollar gemessen wird. Für die meisten russischen Patienten wird die NGS-Diagnostik jedoch weiterhin ein unzulässiger Luxus bleiben, da die Kosten für Zellen für Flongle auf dem heimischen Markt mehr als 10.000 Rubel betragen werden. Und wenn wir die Kosten für verbrauchbare Reagenzien, die Transportkosten und den Overhead hinzufügen, werden die geschätzten Kosten für die Sequenzierung einer DNA / RNA-Probe nicht unter 20.000 Rubel fallen.
Dies impliziert die Notwendigkeit, heimische Nanoporen-Sequenzer zu entwickeln und sie mit billigen Verbrauchsmaterialien und Reagenzien zu versorgen.
Nanorider
Durch die Wiederverwendung von Zellen für Flongle und den Import der Substitution der für die Arbeit mit ihnen erforderlichen Reagenzien können die Kosten für die Nanoporen-Sequenzierung erheblich gesenkt werden. Zunächst benötigen Sie jedoch ein Testgerät, mit dem Sie die Bildung von Doppelschicht-Lipidmembranen (BLM) in 126 Vertiefungen der Arbeitszelle überwachen und die Menge und Qualität der in diese Membranen eingebetteten Nanoporen bewerten können. Ein solches Gerät ("Nanorider"?) Sollte Änderungen der elektrischen Leitfähigkeit (und / oder Impedanz) einzelner Löcher während der Bildung von BLMs auf diesen verfolgen und picoamperöse Ströme registrieren, die durch einzelne Ionenkanäle fließen.
Es ist möglich, ein über USB 2.0 an den Computer angeschlossenes Lesegerät aus Standardelementen zusammenzustellen: FPGA (100 ... 200 $), Stromverstärker (MAX9923FEUB - 200 ... 300 Rubel), ADC (1 ... 2.000 Rubel) usw. ... Es muss entwickelt werden Nur eine Karte mit einem nicht standardmäßigen Kontaktpad (10 x 13) vom Typ LGA (Land Grid Array) zum Andocken an eine Zelle von Flongle. Oder mit seinem vereinfachten Haushaltsgegenstück, das eine kleinere Anzahl (16 ... 32) von Sensorvertiefungen enthält.
 Touch Pad Cell Flongle
Touch Pad Cell FlongleDie Reduzierung der Designproduktivität von Zellen um das 4 ... 8-fache (bis zu 10 ... 20 Mb) ist für die Lösung vieler Probleme (Bestimmung von Krankheitserregern, HLA-Typisierung, Feststellung der Vaterschaft, Identifizierung usw.) nicht entscheidend, ermöglicht jedoch das Sammeln eines Nanoporen-Sequenzers auf herkömmlichen Verstärkern Picoampere-Ströme. Und verzichten Sie auf die Verwendung spezieller 512-Kanal-Chips, in deren Entwicklung die Oxfordianer zig Millionen Dollar investiert haben. Zwar muss jeder Mehrkanal-Nanoreader mehr als ein Dutzend Stromverstärker + ADCs enthalten, aber es kann immer noch ein Vielfaches weniger kosten als die proprietäre MinION + Flongle-Kombination (> 200.000 Rubel). Die geringe Leistung des Nanoreaders kann für russische Benutzer, die keine anständigen Computer haben (Ärzte, Biologen, Biohacker usw.), sogar nützlich sein.
Kilorider
Die von den Oxfordianern entwickelten 512-Kanal-Picoampere-Stromverstärker sind in jeder Einwegzelle für den MinION enthalten. Abfallzellen können weggeworfen werden, aber es ist besser, die darin enthaltenen Chips zu verwenden, um Flongle-Analoga herzustellen, die sich durch eine erhöhte Produktivität auszeichnen. Wenn diese Chips mit Oberflächenkontakten ausgestattet sind, können sie mit Kontaktzellen arbeiten, die 2048 (512 x 4) Sensorvertiefungen enthalten.
Das Entwerfen von Zellen mit solchen LGAs ist nicht einfach, aber Sie müssen berücksichtigen, dass die Kontaktanforderungen hier weniger streng sind als bei Prozessoren und Motherboards, bei denen ein schlechter Kontakt das gesamte System deaktivieren kann. Für einen Sequenzer kann die Inoperabilität sogar der Hälfte der Zellkontakte als akzeptabel angesehen werden. Die verbleibende Hälfte reicht aus, um eine Leistung von 5 ... 10 GB zu gewährleisten.
Die logische Entwicklung dieser Idee wird die Kombination mehrerer ähnlicher Lesegeräte in einem Gerät sein. Auf diese Weise erhalten Sie einen analogen Sequenzer GridION X5, der für die gleichzeitige Arbeit mit fünf (oder mehr) wiederverwendbaren Zellen der heimischen Produktion ausgelegt ist. Ein solcher Sequenzer kann in vielen klinischen Diagnoselabors gefragt sein.
 https://store.nanoporetech.com/devices
https://store.nanoporetech.com/devicesEinweg-PromethION-Sequenzerzellen enthalten 9.000 Sensorvertiefungen auf Chips. Und die Chips selbst sind 3000-Kanal-Verstärker mit Picoampere-Strömen. Der Informationsstrom, den sie mit 24 (PromethION 24) oder 48 (PromethION 48) Informationszellen ausgeben, ist so groß, dass er in Rohform nicht auf einen normalen externen Computer übertragen werden kann. Sequenzer dieses Typs müssen daher mit einem eigenen Supercomputer ausgestattet sein. Wenn der Sequenzer jedoch nur mit einer Zelle funktioniert, kann er an einen normalen Gaming-Laptop mit Thunderbolt 3.0-Anschluss angeschlossen werden.
Die Änderung der freien (ausgeworfenen) Chips von ONT zur Arbeit mit Kontaktzellen ist kein unlösbares Problem. Sequenzer, die auf der Verwendung solcher Chips basieren, können jedoch nur mit ONT-Software arbeiten. Und die Anstrengungen, die für die Neugestaltung dieser Programme aufgewendet werden müssen, können die Vorteile der Verwendung von Standardchips zunichte machen. Und die Entwicklung solcher Chips von Grund auf ist zu teuer.
Mega Rider
In diesem Jahr soll der Beta-Test des Nanoporen-Sequenzers von AXBIO (USA) beginnen. Es werden Chips mit einer Million Stromverstärkungskanälen verwendet, die in Japan von der israelischen Firma Tower Semiconductor Ltd. hergestellt werden. Verbrauchsmaterialien und der Verkauf dieser Sequenzer werden von der chinesischen Niederlassung von AXBIO abgewickelt.

In den USA und in der Europäischen Union sind mehr als 7.000 Patente der Nanoporen-Sequenzierung gewidmet, was für lokale Unternehmen, die versuchen, mit solchen Technologien umzugehen, unvorhersehbare finanzielle Risiken mit sich bringt. Dies ist offenbar der Hauptgrund für eine so hohe Internationalisierung dieser Entwicklung. Außerdem können russische Unternehmen und Labors uneingeschränkt am Testen und Verbessern der AXBIO-Technologie sowie an der Herstellung (oder Sammlung) solcher Sequenzer in ihrem Hoheitsgebiet teilnehmen.
Terabyte an Rohdaten, die von einem Nanoporen-Sequenzer mit Megapixel-Chips (Mega-Reader) erhalten wurden, ist wünschenswert zu filtern. Und übertragen Sie nur die längsten und qualitativ hochwertigsten Lesevorgänge auf den Computer. Dies wird nicht nur die Qualität der Sequenzierung verbessern, sondern auch die Anforderungen an den Computer verringern, was auch für die Massenproduktion und den weit verbreiteten Einsatz von Genomsequenzierern wichtig ist. Darüber hinaus kann die übermäßige Produktivität von Mega-Lesern die Sequenzierungsdauer des menschlichen Genoms von 2 bis 3 Tagen (für ONT) auf mehrere Stunden reduzieren.
Technologiesumme
Der Erfolg von ONT wurde durch die Organisation der parallelen Pipeline-Optimierung der fünf Hauptelemente der Nanoporen-Sequenzierungstechnologie erleichtert - Membranen, Nanoporen, „Motor“ (Helikase), Sequenzierungsbedingungen und Decodierungsalgorithmen für Lesesignale.
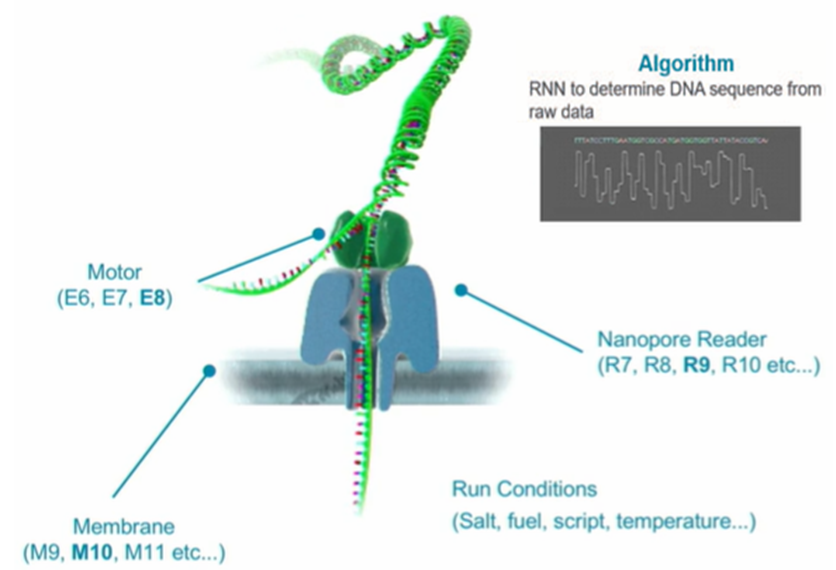 https://nanoporetech.com/resource-centre/videos/sub1000
https://nanoporetech.com/resource-centre/videos/sub1000Die garantierte Haltbarkeit von Zellen für die Nanoporen-Sequenzierung hängt hauptsächlich von der Stabilität der Doppelschicht-Lipidmembran ab. Jetzt sind es 6 Wochen, obwohl solche Membranen normalerweise in wenigen Tagen zerstört werden. Um ihre Haltbarkeit zu erhöhen, werden hydrophobe Polymeradditive, oberflächenaktive Blockcopolymere, Cholesterin, Antioxidationsmittel, Vernetzung von Fettsäuren usw. verwendet, und solche Modifikationen können die Bildung von Ionenkanälen stören. Daher ist es sehr schwierig, eine BLM-Stabilität zu erreichen.
Die radikale Lösung dieses Problems ermöglicht die Bildung von Membranen in den Arbeitszellen des Sequenzers und die Einführung von Nanoporen in diese unmittelbar vor der Verwendung. Die Selbstvorbereitung von Zellen für die Arbeit bietet die Möglichkeit für ihre Regeneration und Wiederverwendung. Die Haltbarkeit trockener Zellen ist nahezu unbegrenzt, und die für ihre Aktivierung und Regeneration erforderlichen Reagenzien können monatelang gelagert werden.
Der Engpass in der Technologie der Nanodrahtsequenzierung ist gleichzeitig der Engpass der Nanopore, von dem die Qualität der gelesenen Informationen abhängt. Daher war eine der Hauptaufgaben von ONT die Suche nach einem Membranprotein, mit dem Ergebnisse von akzeptabler Qualität erzielt werden können.
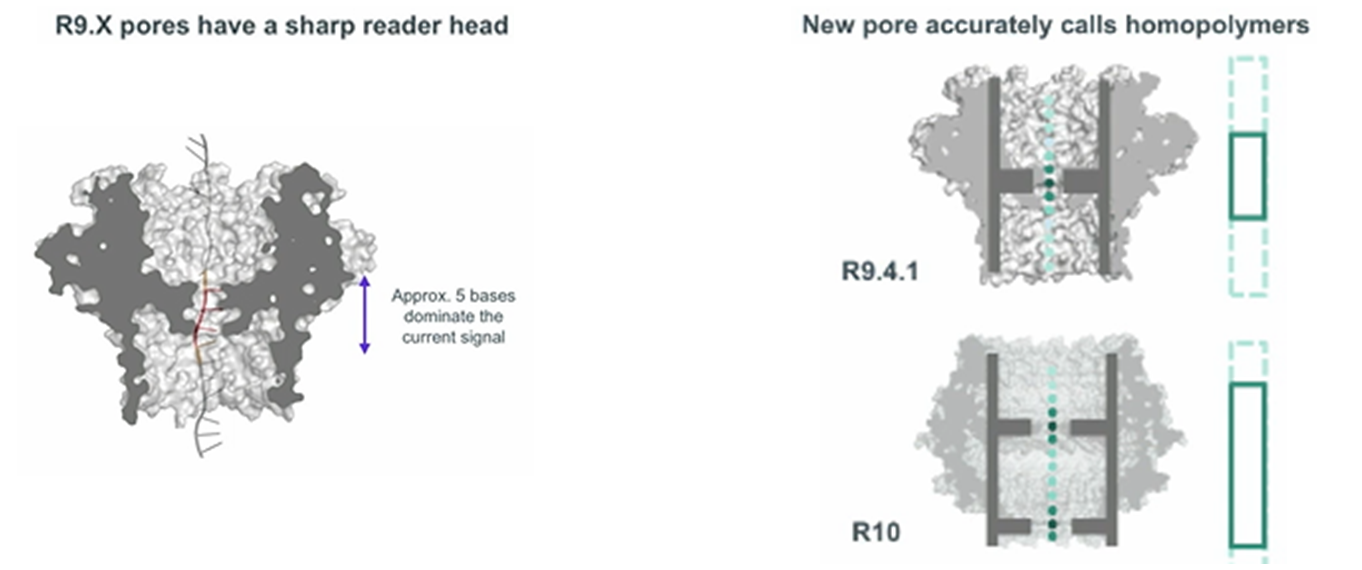
Das erste derartige Protein war CsgG, ein Außenmembranprotein, das in vielen gramnegativen Bakterien gefunden wurde. Neun seiner Untereinheiten bilden einen Ionenkanal in der Membran mit breiten Eingangs- und Ausgangsvorräumen und einem Engpass. Erst nach Aufzählung und Analyse von Hunderten modifizierter CsgG-Varianten konnten jedoch ausreichend hochwertige Nanoporen (R9, R9.4.1, R9.5.1) erhalten werden. In diesem Sommer sollten Zellen mit Poren der nächsten Generation (R10 oder R10b) zum Verkauf angeboten werden, die eine erhöhte Genauigkeit beim Ablesen von Homopolymer-Wiederholungen aufweisen.
 https://nanoporetech.com/about-us/news/london-calling-clive-brown-and-team-plenary
https://nanoporetech.com/about-us/news/london-calling-clive-brown-and-team-plenaryONT ist ein obligatorischer Bestandteil der ONT-Nanoporen-Sequenzierungstechnologie, eine Helikase, ein molekularer Motor, der doppelsträngige DNA abwickelt und den Fortschritt einzelsträngiger DNA durch eine Nanopore hemmt. Die maximale Geschwindigkeit der DNA-Abwicklung durch einen solchen Motor erreicht jetzt 450 Nukleotidpaare pro Sekunde, obwohl die Geschwindigkeit der elektronischen MinION-Komponenten eine Erhöhung auf 1000 Nukleotide pro Sekunde ermöglicht. Theoretisch kann die Beschleunigung der Helikase-Arbeit die Leistung von MinION verdoppeln. In der Praxis kann dies jedoch zu einer schlechten Sequenzierung führen. Daher ist es unwahrscheinlich, dass schnellere und effizientere Helikasen die Leistung von Nanoporen-Sequenzierern wie MinION und PromethION verbessern.
Die Qualität der Sequenzierung hängt von vielen Bedingungen ab - der Größe der an die Membran angelegten Spannung, der elektrischen Leitfähigkeit und der ionischen Zusammensetzung des Reaktionsgemisches, der Menge und Art der Verunreinigungen, die die Poren verstopfen, der Möglichkeit der Reinigung der verstopften Poren durch Ändern der Polarität des Potentials, der Struktur der verwendeten Adapter usw. usw. Die Sequenzierung erfordert eine sorgfältige Optimierung aller Parameter und Zusammensetzungen von Arbeitslösungen (Laufbedingungen), was die Qualität der Digitalisierung von Nukleotidsequenzen nachteilig beeinflussen kann Grannen. Zum Beispiel wurde kürzlich gezeigt, dass eine Abnahme der durch Helikase gespaltenen ATP-Konzentration die Genauigkeit der Sequenzierungsergebnisse allmählich verringert, und jetzt versuchen die Oxfordianer, dieses Problem durch Einführung eines ATP-Regenerationssystems in das Reaktionsgemisch zu beseitigen.
Das fünfte Element der Nanoporen-Sequenzierungstechnologie ist künstliche Intelligenz, die auf wiederkehrenden neuronalen Netzen (RNN) und der Verwendung einiger spezieller Algorithmen und Programme basiert, die die Erkennung homogener Wiederholungen verbessern (Flip-Flop, Medaka). Im Juli soll die nächste Veröffentlichung der Programme Guppi und MinKNOW erscheinen, in der die Oxfordianer versprechen, die Möglichkeit zur Bestimmung methylierter Basen (5 mC und 6 mA) hinzuzufügen. Dies ist nicht nur für epigenomische Studien wichtig, sondern auch, um die Erkennung herkömmlicher Basen zu verbessern.
Die großen Informationsmengen, die von Nanoporen-Sequenzierern gelesen werden, und die Komplexität ihrer Verarbeitung stellen erhöhte Anforderungen an die Hardware solcher Software und Hardwaresysteme. Daher ist nicht jeder Computer für den Anschluss von MinION geeignet. In GridION und PromethION sind Grafikbeschleuniger der neuesten Generation installiert. Oxford Nanopore Technologies war einer der ersten Käufer einer großen Menge von Jetson AGX Xavier (NVIDIA) -Modulen für Geräte mit künstlicher Intelligenz.
In jüngerer Zeit wurde die Nanoporentechnologie als nichts anderes als eine nützliche Ergänzung zu genaueren Fluoreszenz- und Halbleitersequenzierungstechnologien angesehen. Eine allmähliche Erhöhung der Genauigkeit des Lesens von DNA in Kombination mit einer großen Länge lesbarer Sequenzen ermöglichte es ihm jedoch, zumindest den Hauptvektor für die Entwicklung genomischer Sequenzierungstechnologien zu bestimmen, wenn er nicht allen Wettbewerbern voraus war.

Es kann eine Weile dauern, bis alle Elemente der Nanoporen-Sequenzierungstechnologie beherrscht sind, aber dies ist nicht das größte Problem. Das Hauptproblem ist der Mangel an gezielter Finanzierung für solche integrierten Entwicklungen in Russland. Das kürzlich genehmigte wissenschaftliche und technische Programm des Bundes für die Entwicklung genetischer Technologien für 2019–2027 (Abschnitt "Anweisungen zur Programmumsetzung") besagt, dass kurzfristig (3 ... 6 Jahre) ein Prototyp für die Hochleistungs-Genomsequenzierung entwickelt wird. Es ist jedoch nicht so einfach, diese einzelne Zeile auf 30 Textseiten zu finden. Einfachere Verhandlungen mit den Chinesen über den Kauf von AXBIO-Sequenzern.
Noch einfacher ist es, Handwerker zu finden, die den einfachsten Nanorider „am Knie“ zusammenbauen können. Und dann mit ausgestreckter Hand nach Investoren suchen, die sich bereit erklären, die Entwicklung der heimischen Technologie der Nanoporen-Sequenzierung zu finanzieren.