Ich bin auf
einen Artikel im School of Data-Unternehmensblog gestoßen und habe beschlossen, zu überprüfen, wozu die Fast.ai-Bibliothek in demselben Datensatz in der Lage ist, der im Artikel erwähnt wird. Hier finden Sie keine Argumente dafür, wie wichtig es ist, eine Lungenentzündung rechtzeitig und korrekt zu diagnostizieren, ob Radiologen unter den Bedingungen der technologischen Entwicklung benötigt werden, ob die Vorhersage eines neuronalen Netzwerks als medizinische Diagnose angesehen werden kann usw. Das Hauptziel ist zu zeigen, dass maschinelles Lernen in modernen Bibliotheken recht einfach sein kann (erfordert buchstäblich ein paar Codezeilen) und hervorragende Ergebnisse liefert. Erinnern wir uns an das Ergebnis des Artikels (Genauigkeit = 0,84, Rückruf = 0,96) und sehen, was mit uns passiert.
Wir nehmen die Daten für das Training
von hier . Die Daten sind 5856 Röntgenstrahlen, die in zwei Klassen verteilt sind - mit oder ohne Anzeichen einer Lungenentzündung. Die Aufgabe des neuronalen Netzwerks besteht darin, uns einen hochwertigen binären Klassifikator für Röntgenbilder zur Bestimmung der Anzeichen einer Lungenentzündung zu geben.
Wir beginnen mit dem Import der Bibliotheken und einiger Standardeinstellungen:
%reload_ext autoreload %autoreload 2 %matplotlib inline from fastai.vision import * from fastai.metrics import error_rate import os
Bestimmen Sie als Nächstes die Stapelgröße. Wenn Sie auf der GPU lernen, ist es wichtig, diese so zu wählen, dass Ihr Speicher nicht voll ist. Bei Bedarf kann es halbiert werden.
bs = 64
Wichtiges Update:Wie in den Kommentaren unten zu Recht erwähnt, ist es wichtig, die Daten, an denen das Modell trainiert wird und an denen wir seine Wirksamkeit testen, klar zu überwachen. Wir werden das Modell in den Bildern in den Ordnern train und val trainieren und in den Bildern im Testordner validieren, ähnlich wie
hier .
Wir bestimmen die Pfade zu unseren Daten
path = Path('storage/chest_xray') path.ls()
und überprüfen Sie, ob alle Ordner vorhanden sind (der Ordner val wurde zum Trainieren verschoben):
Out: [PosixPath('storage/chest_xray/train'), PosixPath('storage/chest_xray/test')]
Wir bereiten unsere Daten für das „Laden“ in das neuronale Netzwerk vor. Es ist wichtig zu beachten, dass es in Fast.ai verschiedene Methoden gibt, um die Bildbezeichnung abzugleichen. Die from_folder-Methode gibt an, dass Beschriftungen aus dem Namen des Ordners entnommen werden sollen, in dem sich das Bild befindet.
Der Größenparameter bedeutet, dass wir die Größe aller Bilder auf eine Größe von 299 x 299 ändern (unsere Algorithmen arbeiten mit quadratischen Bildern). Die Funktion get_transforms erweitert das Bild, um die Menge der Trainingsdaten zu erhöhen (die Standardeinstellungen belassen wir hier).
np.random.seed(5) data = ImageDataBunch.from_folder(path, train = 'train', valid = 'test', size=299, bs=bs, ds_tfms=get_transforms()).normalize(imagenet_stats)
Schauen wir uns die Daten an:
data.show_batch(rows=3, figsize=(6,6))
Um zu überprüfen, schauen wir uns an, welche Klassen wir haben und welche quantitative Verteilung der Bilder zwischen Zug und Validierung:
data.classes, data.c, len(data.train_ds), len(data.valid_ds)
Out: (['NORMAL', 'PNEUMONIA'], 2, 5232, 624)
Wir definieren ein Trainingsmodell basierend auf der Resnet50-Architektur:
learn = cnn_learner(data, models.resnet50, metrics=error_rate)
und beginnen Sie in 8 Epochen zu lernen, basierend auf der
Ein-Zyklus-Richtlinie :
learn.fit_one_cycle(8)
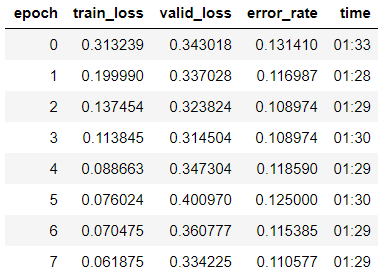
Wir sehen, dass wir bereits eine Genauigkeit von 89% für die Validierungsprobe erhalten haben. Wir werden vorerst die Gewichte unseres Modells aufschreiben und versuchen, das Ergebnis zu verbessern.
learn.save('step-1-50')
Das ganze Modell "auftauen", weil Zuvor haben wir das Modell nur für die letzte Schichtgruppe trainiert, und die Gewichte der übrigen Modelle wurden dem auf Imagenet vorab trainierten und „gefrorenen“ Modell entnommen:
learn.unfreeze()
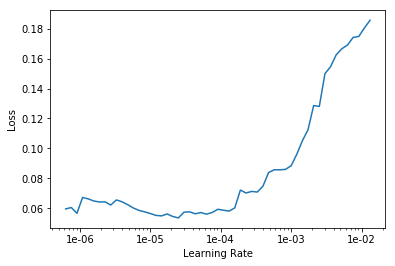
Wir suchen die optimale Lernrate, um weiter zu lernen:
learn.lr_find() learn.recorder.plot()
Wir beginnen mit dem Training für 10 Epochen mit unterschiedlichen Lernraten für jede Schichtgruppe.
learn.fit_one_cycle(10, max_lr=slice(1e-6, 1e-4))
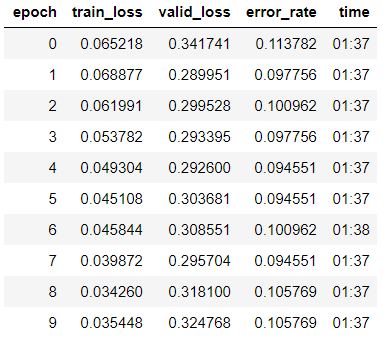
Wir sehen, dass die Genauigkeit unseres Modells in der Validierungsstichprobe leicht auf 89,4% gestiegen ist.
Wir schreiben die Gewichte auf.
learn.save('step-2-50')
Verwirrungsmatrix erstellen:
interp = ClassificationInterpretation.from_learner(learn) interp.plot_confusion_matrix()
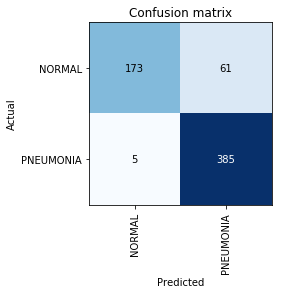
An dieser Stelle erinnern wir uns, dass der Genauigkeitsparameter allein nicht ausreicht, insbesondere für unsymmetrische Klassen. Wenn beispielsweise im wirklichen Leben eine Lungenentzündung nur bei 0,1% derjenigen auftritt, die sich einer Röntgenuntersuchung unterziehen, kann das System in allen Fällen einfach das Fehlen einer Lungenentzündung feststellen, und seine Genauigkeit liegt bei 99,9%, wobei der Nutzen absolut Null ist.
Hier kommen Präzisions- und Rückrufmetriken ins Spiel:
- TP - echte positive Vorhersage;
- TN - wahre negative Vorhersage;
- FP - falsch positive Vorhersage;
- FN - Falsch negative Vorhersage.
Präzision=TP/(TP+FP)=385/446=0,863
Recall=TP/(TP+FN)=385/390=$0,98
Wir sehen, dass das Ergebnis, das wir erhalten haben, sogar etwas höher ist als das im Artikel erwähnte. Bei der weiteren Arbeit an der Aufgabe ist zu beachten, dass der Rückruf ein äußerst wichtiger Parameter bei medizinischen Problemen ist, weil Falsch Negative Fehler sind aus diagnostischer Sicht am gefährlichsten (was bedeutet, dass wir eine gefährliche Diagnose einfach „übersehen“ können).