← Artículo anterior en el cicloLa lógica de incluir operones de diferentes vías metabólicas en E. coli está cerca de los elementos AND y OR. Por lo tanto, en base a ellos, puede hacer una construcción genética que implemente un esquema lógico arbitrario (no demasiado complicado). Hay programas, por ejemplo, Cello, que, a partir de la descripción de la lógica en Verilog, hacen una secuencia de una construcción genética que implementa esta lógica.
Dinámica de cambio de genes
En el último artículo, modelamos el funcionamiento del operón lactosa con un modelo simple con cuatro parámetros. El tiempo no era parte de este modelo. Por lo tanto, de hecho, solo describe el estado de equilibrio, cuando los niveles de la señal de entrada permanecen constantes durante mucho tiempo. Para modelar procesos rápidos al activar y desactivar genes, se deben considerar factores adicionales.En primer lugar, hay un retraso transcripcional: desde el comienzo del trabajo de la ARN polimerasa hasta el final de la síntesis de ARNm, y luego las proteínas, pasa un tiempo notable, aproximadamente 10 minutos en Escherichia coli y de una hora o más en humanos. Por lo tanto, la tasa de aparición de nuevas moléculas de proteínas no depende del estado del promotor de su gen en este momento, sino del estado del promotor hace unos minutos.En segundo lugar, cada proteína no solo se sintetiza, sino que también se descompone. En el caso más simple, la descomposición de la proteína no se controla y cada minuto una fracción fija de las moléculas de esta proteína se descompone en la célula. Cuanto más son, más se rompen. Es decir, la proteína tiene una vida media, durante la cual su concentración se reduce a la mitad.Más detalles para los más curiosos.En las células en crecimiento, hay un segundo factor en la disminución de la concentración de proteínas: la dilución debido a un aumento en el volumen celular. Se describe con la misma fórmula que la descomposición: la concentración disminuye dos veces en el tiempo de una división celular a la siguiente.
Para describir la dinámica de activar y desactivar el gen, necesitamos escribir un diffur:
donde A es la actividad del gen (la misma función de entrada del artículo anterior), tau es el tiempo de demora de la transcripción yk es la tasa de descomposición de proteínas. Si A es primero igual a cero, y luego aumenta bruscamente y se mantiene en un nivel constante, los cambios en la cantidad de proteína a lo largo del tiempo se verán así:Motivos en las redes de genes y sus funciones.
El motivo más simple característico de Escherichia coli, el circuito de retroalimentación, consiste en un gen que se regula a sí mismo. Muy a menudo, el gen es un represor por sí mismo, es decir, la retroalimentación en el bucle es negativa. Si este es el principal aporte regulatorio, esta retroalimentación generalmente sirve para estabilizar el nivel de la proteína codificada por el gen. Tal circuito de retroalimentación, por ejemplo, mantiene un nivel constante de represor de lactosa en la célula. Si, además de la retroalimentación negativa, hay otras entradas reguladoras, entonces la retroalimentación acelera la respuesta del gen a una señal externa.(las líneas discontinuas son curvas teóricas, las líneas continuas son el resultado del experimento. Las líneas verdes con retroalimentación negativa, las líneas azules sin ella)La retroalimentación positiva (una proteína activa su propio gen) generalmente conduce a un comportamiento desencadenante. Tal gen tiene dos estados estables: o no funciona en absoluto, o funciona a plena capacidad. Una señal externa fuerte pero corta puede transferir un gen de un estado estable a otro en el que permanece hasta la próxima señal. Tal motivo puede servir como un elemento de memoria. Si un gen "capta" ruido en una entrada reguladora, entonces una retroalimentación positiva conducirá a una fuerte propagación de la actividad génica entre las células vecinas que crecen en las mismas condiciones. Esto se utiliza para mantener la heterogeneidad de las bacterias que crecen juntas de modo que el estrés repentino no mate a todos a la vez.Por ejemplo, muchos antibióticos interfieren con la síntesis de proteínas. Si la célula no crece y no sintetiza proteínas, puede sobrevivir fácilmente a la adición de antibióticos al medio y volver a crecer más tarde. En E. coli, incluso en condiciones ideales, hasta el 0,01% de las células están en hibernación en caso de envenenamiento repentino del medio ambiente. Estas células latentes (se denominan "persistores") no difieren del resto en el ADN; entran y salen de la hibernación bajo el control de un gen con retroalimentación positiva y entrada de ruido.Otro motivo común de las redes de genes es un poco más complicado. Se llama un "ciclo de alimentación hacia adelante" y consta de tres genes: el gen X regula los genes Y y Z, el gen Y regula Z. Cada uno de los tres enlaces en el ciclo puede ser activación o represión, por lo tanto, en principio, son posibles 8 tipos tales bucles En realidad, generalmente se encuentran dos tipos de ocho: los genes X e Y son activadores (bucle coherente tipo 1), o X es el activador Y y Z, Y es el represor Z (bucle incoherente tipo 1).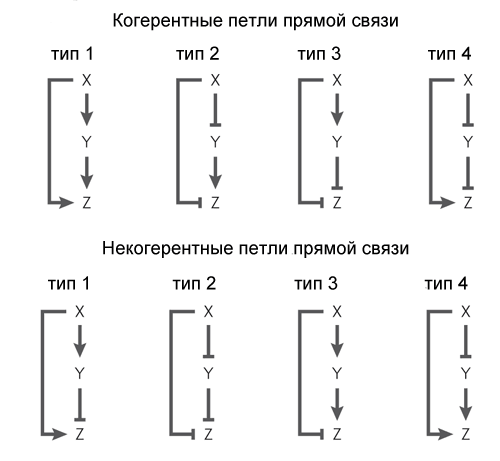
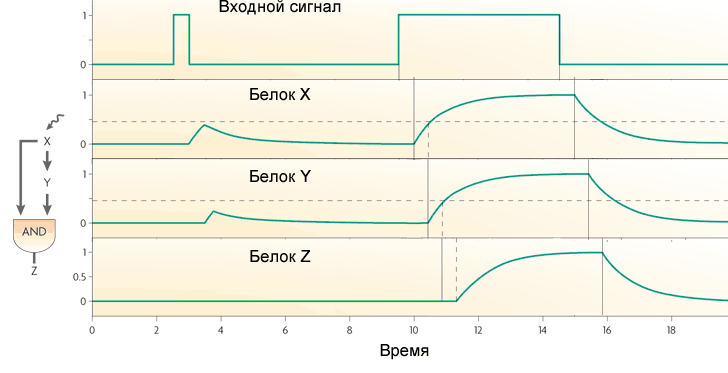 Si el gen Z funciona como OR, entonces la función del bucle se convierte en lo opuesto: pasa todos los pulsos a la salida y se convierte en cortos en largos. Pero ella no pierde pausas cortas entre pulsos.El bucle directo con el activador X y el represor Y actúa como un filtro de paso alto. Produce pulsos de pequeña longitud fija (igual al retraso transcripcional del gen Y) en la salida en respuesta a los bordes de las señales de entrada.El tercer motivo frecuente en las redes de genes de Escherichia coli es un módulo de entrada única. Es simplemente un grupo de genes impulsados por el mismo gen regulador. Por lo general, los genes en este grupo tienen diferentes umbrales de activación, por lo que un aumento suave en la concentración del factor de transcripción incluye genes en un cierto orden. Tal esquema se usa, por ejemplo, en el manejo de respuestas al estrés.
Si el gen Z funciona como OR, entonces la función del bucle se convierte en lo opuesto: pasa todos los pulsos a la salida y se convierte en cortos en largos. Pero ella no pierde pausas cortas entre pulsos.El bucle directo con el activador X y el represor Y actúa como un filtro de paso alto. Produce pulsos de pequeña longitud fija (igual al retraso transcripcional del gen Y) en la salida en respuesta a los bordes de las señales de entrada.El tercer motivo frecuente en las redes de genes de Escherichia coli es un módulo de entrada única. Es simplemente un grupo de genes impulsados por el mismo gen regulador. Por lo general, los genes en este grupo tienen diferentes umbrales de activación, por lo que un aumento suave en la concentración del factor de transcripción incluye genes en un cierto orden. Tal esquema se usa, por ejemplo, en el manejo de respuestas al estrés.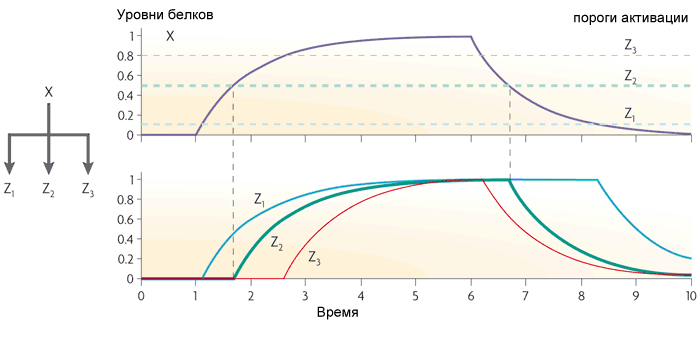 El cuarto motivo más complejo es el módulo de entradas múltiples o regulón denso y superpuesto. Se compone de varios genes reguladores que controlan varios genes de "salida".Dependiendo de la lógica de regulación de cada gen de salida, este motivo puede funcionar de manera muy diferente. Se puede comparar con un conjunto de elementos lógicos que, dependiendo de las conexiones, pueden ser un decodificador, contador u otra cosa. Todavía no existen descripciones completas de la lógica del trabajo de tales motivos.Con estos cuatro motivos, la estructura de las redes de genes de Escherichia coli o levadura está prácticamente agotada. Sobre esta base, ya se están realizando varios esquemas artificiales. Por ejemplo, bacterias con un generador de oscilación, que también se sincroniza en las células vecinas, y la salida de proteínas luminiscentes.- Un análogo completo de "parpadea una bombilla" para microcontroladores y "Hello World" para otros programadores. Al agregar una "sensación de quórum" a este sistema (cuando las bacterias perciben cuántos primos los rodean), y al cambiar la producción de luminiscencia a toxinas, los científicos han recolectado bacterias que se arrastran hacia tumores cancerosos y se acumulan en grandes cantidades, mueren juntas, liberan toxinas y matan tumor ( más detalles aquí ).Pero estas son bacterias y levaduras. Las redes de genes animales son mucho más complejas. En la siguiente parte, analizaremos una de las redes genéticas animales bien estudiadas y los problemas que surgen con su estudio y modelado.
El cuarto motivo más complejo es el módulo de entradas múltiples o regulón denso y superpuesto. Se compone de varios genes reguladores que controlan varios genes de "salida".Dependiendo de la lógica de regulación de cada gen de salida, este motivo puede funcionar de manera muy diferente. Se puede comparar con un conjunto de elementos lógicos que, dependiendo de las conexiones, pueden ser un decodificador, contador u otra cosa. Todavía no existen descripciones completas de la lógica del trabajo de tales motivos.Con estos cuatro motivos, la estructura de las redes de genes de Escherichia coli o levadura está prácticamente agotada. Sobre esta base, ya se están realizando varios esquemas artificiales. Por ejemplo, bacterias con un generador de oscilación, que también se sincroniza en las células vecinas, y la salida de proteínas luminiscentes.- Un análogo completo de "parpadea una bombilla" para microcontroladores y "Hello World" para otros programadores. Al agregar una "sensación de quórum" a este sistema (cuando las bacterias perciben cuántos primos los rodean), y al cambiar la producción de luminiscencia a toxinas, los científicos han recolectado bacterias que se arrastran hacia tumores cancerosos y se acumulan en grandes cantidades, mueren juntas, liberan toxinas y matan tumor ( más detalles aquí ).Pero estas son bacterias y levaduras. Las redes de genes animales son mucho más complejas. En la siguiente parte, analizaremos una de las redes genéticas animales bien estudiadas y los problemas que surgen con su estudio y modelado.