
El experimento a gran escala (cuantitativa y cualitativamente) de modificación génica de un organismo eucariota continúa.
Un grupo internacional de más de 200 científicos
publicó en la revista Science los últimos resultados del
Synthetic Yeast Project (Sc2.0) , cuyo objetivo final es crear
levadura de panadería con un genoma completamente sintético. En la actualidad, se han sintetizado 6 cromosomas, que reemplazaron a sus contrapartes naturales. Los organismos resultantes de Saccharomyces cerevisiae son bastante viables y tienen varias propiedades de diseño complejas predeterminadas.
Eucariotas - "nuclear normal" - organismos en los que el material genético está encerrado en un orgánulo separado rodeado por una membrana de membrana - el núcleo. En términos simplificados, todos los seres vivos que no son bacterias (no tienen núcleo) son eucariotas. La levadura es un organismo eucariota ejemplar tanto para una persona común (solo recuerde cómo se hacen el pan y la cerveza) como para un biotecnólogo. Siendo el llamado organismo modelo, la levadura posee las mejores propiedades para estudiar los procesos básicos de la vida de las células eucariotas. No es sorprendente, respectivamente, la elección del cuerpo por parte del equipo Sc2.0 para tales intervenciones sin precedentes.
El principio de diseño básico del nuevo genoma, declarado por el equipo, es lograr un equilibrio entre el mantenimiento del fenotipo de "especies silvestres" (características externas de la variante natural de la subespecie S288C), la introducción de la "motilidad" genómica inducida y la eliminación de las fuentes de inestabilidad genómica.
 Cromosomas designados para la síntesis de subcomandos
Cromosomas designados para la síntesis de subcomandosLa conservación del fenotipo se logra preservando los genes mismos. Decidieron no cambiar su orden y número en los cromosomas, con la excepción de ciertos grupos específicos importantes.
La inducción de la "movilidad" genómica se logra a través del sistema de recombinación SCRaMbLE, que baraja las regiones cromosómicas cuando se aplica una señal específica a las regiones loxPsym introducidas especialmente en el genoma en sitios estratégicos. Esto hace posible permitir la simulación de uno de los mecanismos de evolución "grandes" globales en la escala de todo el genoma. Al mismo tiempo, los científicos trataron de eliminar una fuente de evolución genómica tan grande como elementos móviles, que, bajo ciertas condiciones, se "pegan" o se "pegan" con resultados impredecibles. Podemos decir que los científicos tomaron la macroevolución del genoma de la levadura de la casualidad a la suya.
Otros cambios en el código en sí incluyen la asignación de genes de ARN de transferencia a un neocromosoma separado, la eliminación de muchos intrones, la inclusión de etiquetas especiales en el genoma para la conveniencia de distinguir cromosomas sintéticos y naturales en las células, y la inclusión de secciones especiales para simplificar el ensamblaje de los cromosomas. La distancia promedio entre las regiones que distinguen el genoma sintético del genoma natural es pequeña: alrededor de 400 pares de bases de ADN.
La síntesis, a pesar de todo su revolucionismo, sin embargo no tuvo lugar desde cero, sino mediante la eliminación y fijación de nuevas regiones a los cromosomas naturales (recombinación jerárquica). Para acelerar el proceso, se asignaron diferentes cromosomas para la síntesis de subcomandos en diferentes países. El ensamblaje de varios cromosomas artificiales en una célula también ocurre jerárquicamente.
Utilizando métodos de fenotipado, genómica estructural y funcional, los científicos estaban convencidos del funcionamiento normal de la levadura sintética. Uno de estos métodos para monitorear el éxito de un proyecto es el análisis de contacto de los cromosomas. El método moderno Hi-C nos permite analizar la estructura interna del núcleo calculando las probabilidades de contactos cromosómicos entre sí y entre sí en diferentes áreas. La versión tridimensional de tal visualización mostró claramente que los cromosomas artificiales, a pesar de su acortamiento (intrones eliminados) e insertos de diseño, ocupan posiciones en el núcleo similares a sus contrapartes naturales. En la siguiente ilustración, cada cromosoma es un par de ramas de diferentes longitudes que cuelgan de un cierto punto a lo largo de su longitud: el centrómero (círculos blancos en la parte superior de A). Los cromosomas artificiales resaltados en color en B, C y D generalmente corresponden únicamente a sus contrapartes en A en posición y orientación. Cabe señalar que esta visualización es precisamente el promedio probabilístico sobre miles de millones de celdas, por lo que no se muestran las "posiciones" de las ramas, sino algo remotamente asociado con el cuadrado del módulo de función psi en mecánica cuántica.
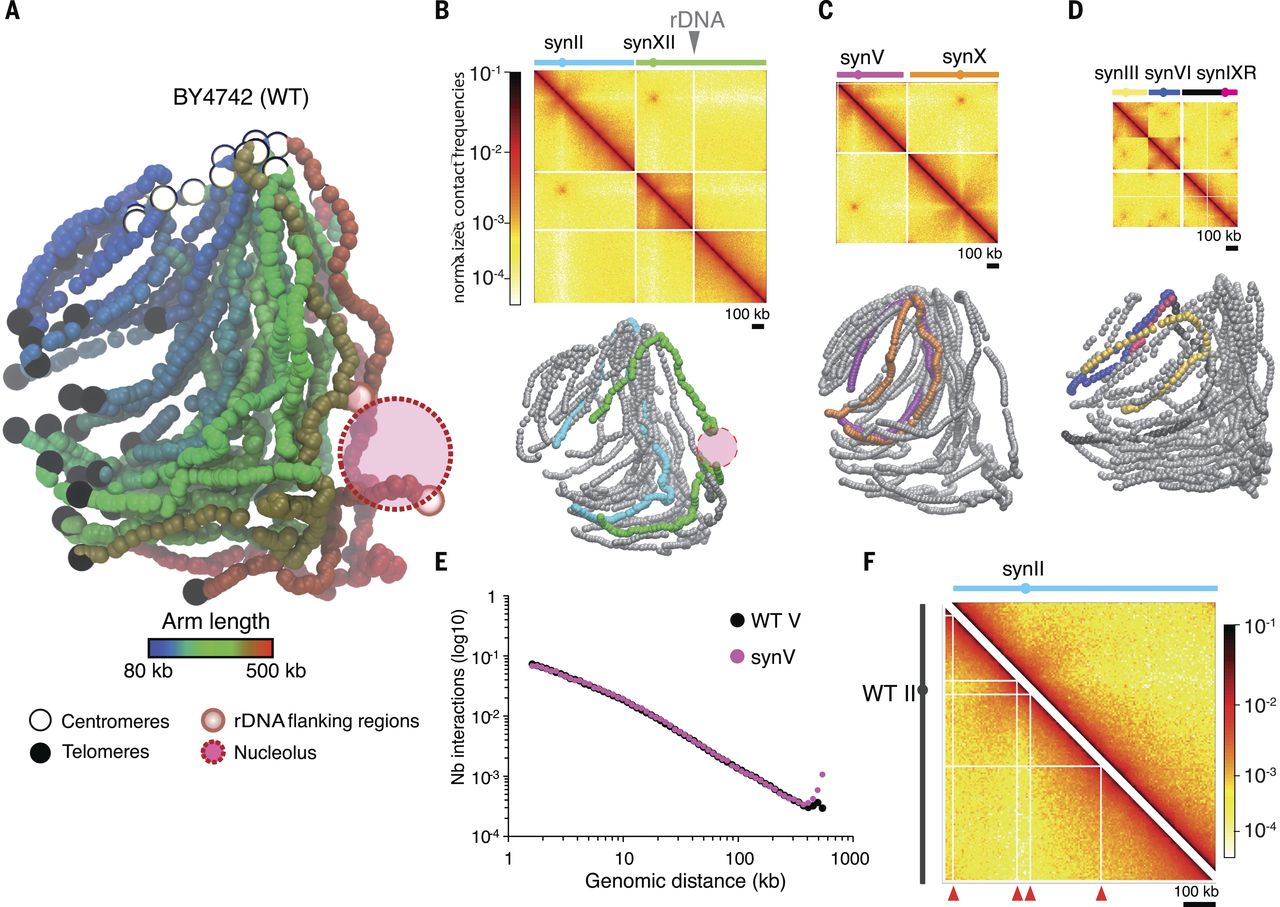
El trabajo de ensamblaje se realizó y se lleva a cabo "de abajo hacia arriba", con simulación completa por computadora, síntesis paso a paso de secciones con depuración posterior. Por el momento, el equipo no ha encontrado serios errores fundamentales que cuestionen la posibilidad de un proyecto o la base teórica del diseño.
Proyecto de levadura sintética: el "acto de creación" más significativo de una persona viva en la actualidad. La levadura ahora se convertirá no solo en el eucariota más estudiado, sino también en el punto de apoyo, gracias al cual puedes aprender creando. El libro de la vida difícilmente se puede entender sin tratar de escribirlo.
El equipo planea completar el ensamblaje del cuerpo para fines de 2017.