Hola a todos, mi nombre es Alexander Sokolov, y quiero contarles cómo hice un secuenciador en casa, un dispositivo para decodificar el ADN. El precio de mercado de dicho dispositivo es de aproximadamente 10 millones de rublos.

Una breve excursión a la genética. Si recuerdas de repente, en 2003 se hizo una declaración sensacional: los científicos finalmente descifraron el genoma humano. El genoma está construido a partir de ADN, y el ADN es el código fuente del cuerpo. El ADN es una cadena doble que consta de 4 tipos de nucleótidos, que se repiten en el genoma humano aproximadamente 3 mil millones de veces. Así como toda la información en su computadora está encriptada en bits, las instrucciones para ensamblar todas las proteínas del cuerpo humano están encriptadas en nucleótidos. Es decir, sabiendo en qué secuencia se ubican los nucleótidos en el ADN, teóricamente podemos recolectar todas las proteínas necesarias y obtener un modelo humano. Entonces, en el sentido estándar, los científicos no descifraron el ADN, sino que simplemente tradujeron la secuencia química en un conjunto de ceros y unos en la computadora. Lo que debe hacer a continuación es una conversación separada. Por ejemplo, en este momento tenemos claro la función de solo el 5% de toda la matriz del genoma (esto es la codificación de proteínas). Lo que hace el 95% restante, solo se puede especular.
En 2003, el costo de la secuenciación del ADN humano fue de aproximadamente $ 100 millones. Con el tiempo, esta cifra ha disminuido y ahora se acerca a los mil dólares. Usted paga, su ADN se secuencia y se le entrega un disco duro con 3 GB de información: su genoma en forma digital.
Hay tres secuenciadores principales en el mercado hoy. El más productivo, Hiseq, y su receptor NovaSeq, proporcionan la secuenciación más barata (fluorescente). Uno de sus lanzamientos dura varios días, y durante este tiempo los genomas de varias personas se procesan a la vez. Sin embargo, el lanzamiento en sí cuesta unos diez mil dólares. Por cierto, el dispositivo en sí cuesta alrededor de $ 1 millón, y dado que se vuelve obsoleto en aproximadamente 3 años, para que valga la pena, debe traerle $ 1000 por día.
El segundo dispositivo apareció en el mercado el verano pasado. Se llama Nanopore y se basa en una tecnología muy interesante cuando el ADN se secuencia pasando a través de un nanopore. La variante Nanopore más barata se posiciona como un secuenciador casero desechable y cuesta $ 1000.
El tercer dispositivo es PGM, un secuenciador de semiconductores que cuesta $ 50,000 en su tierra natal y unos 10 millones de rublos (con entrega, despacho de aduanas, etc.) en Rusia. El proceso de secuenciación lleva varias horas.
Bueno, no tenía diez millones, pero quería PGM. Tuve que hacerlo yo mismo. Primero, brevemente sobre cómo ocurre la secuenciación de semiconductores. Toda la cadena de ADN se divide en fragmentos de 300-400 nucleótidos de longitud, llamados lecturas. Luego, las lecturas se unen a esferas pequeñas y se copian muchas veces, como resultado, un montón de fragmentos de ADN idénticos "cuelgan" en cada esfera. La copia es necesaria para amplificar la señal de cada lectura particular. Un conjunto de áreas diferentes se llama biblioteca de ADN.
El corazón de PGM es un chip de una sola vez: una matriz similar a la matriz de la cámara, pero en lugar de píxeles que responden a la luz, aquí hay transistores de pH que responden a los cambios en el equilibrio ácido-base. La biblioteca de ADN resultante se carga en un chip que contiene 10 millones de pocillos; en la parte inferior de cada uno de ellos hay un transistor de pH. Solo una esfera cabe en un agujero y, por lo tanto, lee de un solo tipo (con una secuencia de nucleótidos específica). Luego, los reactivos se alimentan al chip para que el ADN comience a copiarse. Y se copia linealmente, es decir, los nucleótidos se unen a la cadena recién creada en el orden en que se encuentran en la cadena madre. Por lo tanto, se alimenta un tipo de nucleótidos al chip, y el cambio de pH en algunos pocillos se registra de inmediato (esto significa que el nucleótido se unió a ellos). Luego se alimenta un tipo diferente de nucleótidos y se registran los cambios de pH en los pocillos, etc. Por lo tanto, al aplicar al chip los 4 tipos de nucleótidos muchas veces, podemos obtener información sobre la secuencia de nucleótidos en cada lectura. Luego, por métodos matemáticos, los segmentos cortos leídos se recopilan en una computadora en una sola cadena. Para recolectarlo más o menos con confianza, cada lectura debe leerse unas 100 veces.
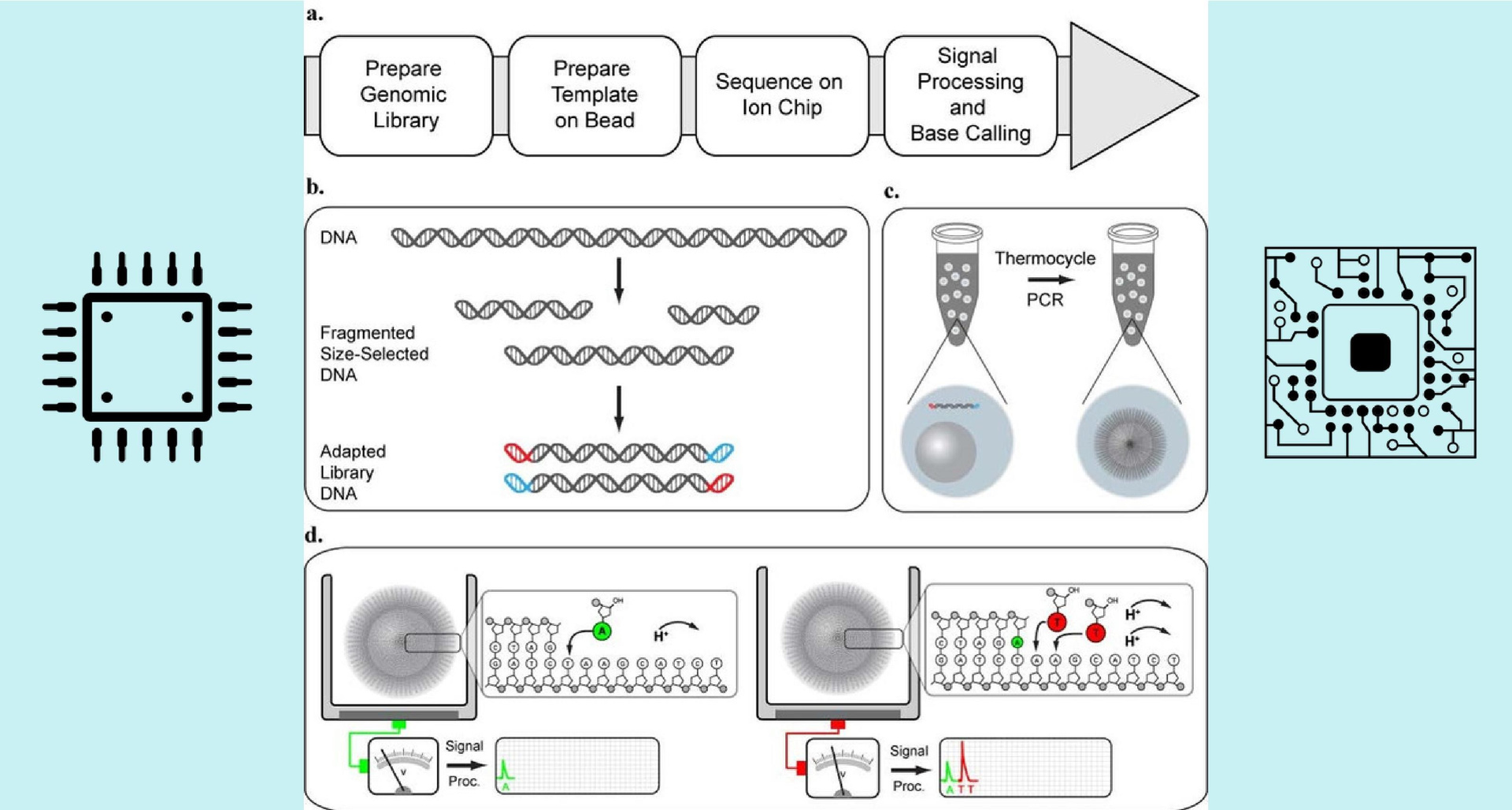 Fig.1. Secuenciación de semiconductores
Fig.1. Secuenciación de semiconductoresAhora veamos en qué consiste el dispositivo. Hay, como ya sabemos, un chip, así como un sistema de suministro de reactivos y una placa base. Toda la secuencia se lleva a cabo con precisión en el chip: el resto del dispositivo solo le transmite ciertas señales, entrega reactivos, lee señales analógicas, las digitaliza y conduce el flujo de información recibido a una computadora, donde los datos se acumulan y procesan.
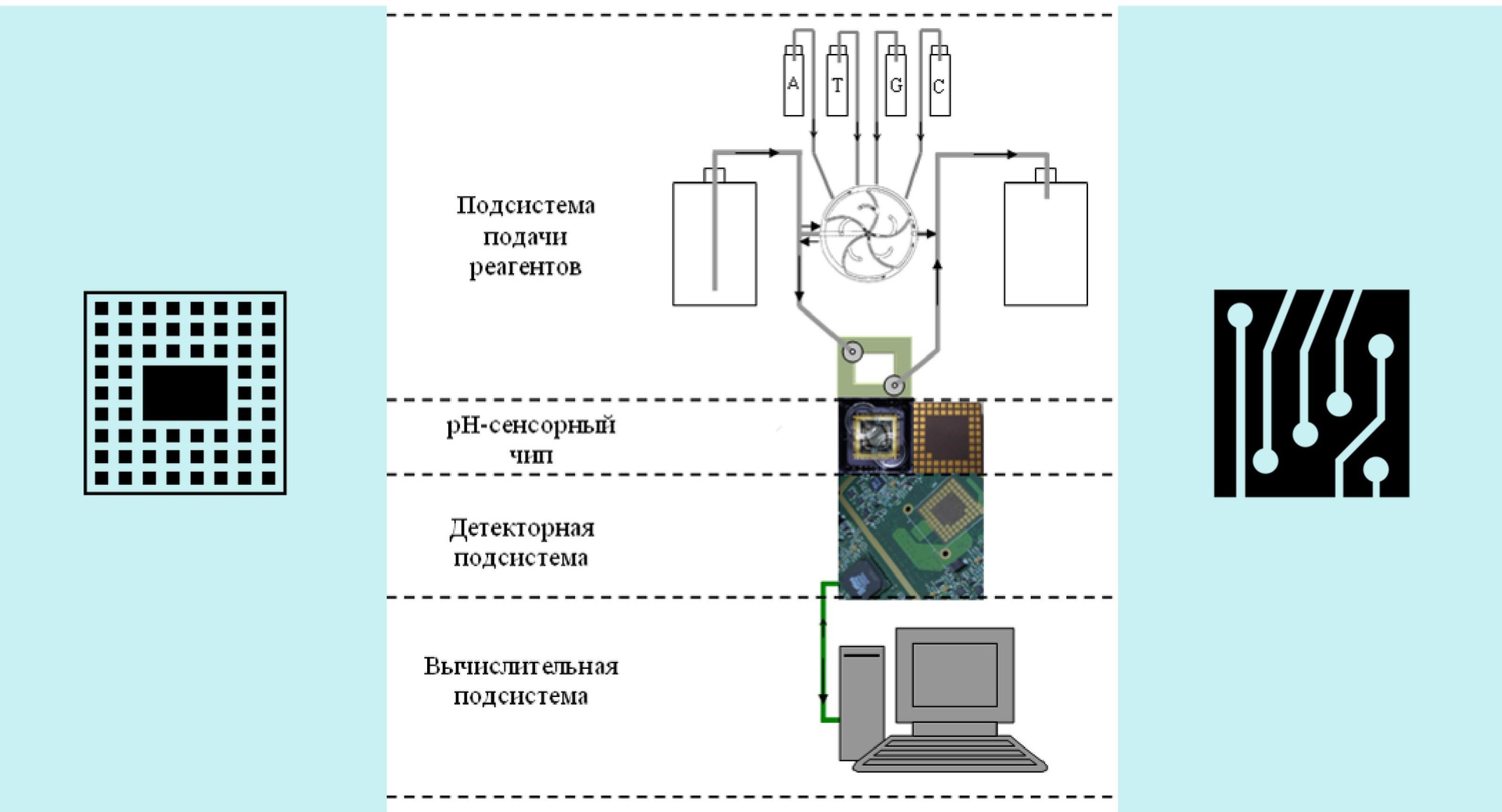 Fig. 2. Dispositivo secuenciador
Fig. 2. Dispositivo secuenciadorEl chip se posiciona una vez y se tira después de su uso. En consecuencia, donde PGM funciona, tales chips se pueden obtener de forma gratuita en cualquier cantidad. ¿Por qué conseguirlos, preguntas? El hecho es que ya he logrado usar el chip repetidamente. De hecho, es eterno: enjuáguelo lo suficientemente bien y puede usarse una y otra vez. En precisión, no diferirá de la nueva. Mi idea era crear un dispositivo para este chip shareware.
Entonces, me enfrenté a la tarea de ingeniería inversa del chip. Por supuesto, no se pudo encontrar documentación para el codiciado microcircuito: el fabricante no iba a compartir los secretos de la producción, pero quería vender tranquilamente sus dispositivos por $ 50,000. Para comenzar, hice lo más obvio y simple: los contactos sonaron con un probador. Quedó claro dónde se encuentran las E / S digitales y analógicas, la potencia y más. Parte de la información se obtuvo de las patentes en el chip. Pero todo esto, por supuesto, no fue suficiente para crear un producto completo. Todavía jugueteaba con el chip, revisé mis varias conjeturas, experimenté dando señales, pero básicamente no avancé a ninguna parte. Tuve que pausar el proyecto.
 Fig. 3. Chip
Fig. 3. ChipY luego, de repente, en Habrahabr, me encontré
con un artículo del famoso blogger
BarsMonster sobre cómo hace ingeniería inversa de chips. Se inspiró, le escribió, escribió a otros entusiastas, envió una solicitud a Kiev, donde se dedicaba a fotografiar chips. Respondieron desde Kiev que no saben cómo pulir en capas, solo pueden disparar la capa superior, y dado que mi chip es multicapa, no estará claro dónde van las pistas de los contactos. Luego conoció a un estadounidense que también se dedica a la ingeniería inversa de chips, le envió sus microcircuitos, pero incluso aquí, no fue más que fotografiar la capa superior. Luego me encontré con un artículo en Internet sobre aquellos que pudieron revertir el chip Sony PlayStation, etc. ("¡Gloria a los héroes!" Y eso es todo, si alguien lo sabe). Decidí escribirlos con preguntas, encontré sus apodos y luego me di cuenta de que uno de ellos me era familiar. Recientemente, un amigo me trajo a un amigo que también está "involucrado en la genética a nivel amateur", hablamos con este amigo en Skype y terminamos este diálogo. Y ahora entiendo que mi nuevo amigo es un maestro genial de chips de ingeniería inversa. Le escribí allí mismo. Sin embargo, resultó que, aunque estaba listo para ayudar, no tenía un microscopio. Callejón sin salida de nuevo.
Unos meses más tarde, ¡se encontró el microscopio necesario en un laboratorio cercano! Es cierto, la cámara incorporada era terrible, tomé fotos en un teléfono móvil a través de un ocular y recibí fotos de esta calidad:
 Fig. 4. El chip bajo el microscopio.
Fig. 4. El chip bajo el microscopio.Luego, en el último año nuevo, apareció un excelente microscopio para 130 mil en mi trabajo (soy especialista en criptografía cuántica). Los sueños se hacen realidad. Finalmente, pude fotografiar normalmente el chip desde arriba.
 Fig. 5. Mi microscopio de trabajo
Fig. 5. Mi microscopio de trabajoY luego ... Entonces aún tenía que dominar la técnica de pulirlo yo mismo. La dificultad del pulido es eliminar capas de metal con un grosor del orden de 1 micra, mientras que el ancho del chip es de 1 centímetro. A modo de comparación, diré que es casi lo mismo que permitir un error de no más de 10 cm por 1 km. Lo intenté mucho. Los resultados de mi trabajo se presentan en la siguiente foto:
 Fig. 6. Ingeniería inversa bajo un microscopio óptico.
Fig. 6. Ingeniería inversa bajo un microscopio óptico.La capa inferior de silicio, la capa superior con transistores, la primera, segunda, tercera y cuarta capas de metal son claramente visibles.
El chip consta de zonas repetidas (como registros de desplazamiento), y a partir de esas imágenes fue muy conveniente analizarlo: inmediatamente quedó claro lo que estaba sucediendo en las diferentes capas. "Invertí" las secciones más "rellenas" con una gran cantidad de lógica, que se repitieron muchas veces. Pero lo más difícil fue rastrear las pistas que corren a lo largo de todo el chip, para entender qué contacto externo se refiere a qué. Desde las vacaciones de Año Nuevo hasta finales de febrero, yo, armado con un nuevo y hermoso microscopio, estudié detenidamente esta tarea: me senté en el trabajo hasta las diez de la noche, "invertido", pensé. Y luego ocurrió un nuevo milagro: un amigo pudo organizar fotografías gratuitas del chip en capas en un microscopio electrónico en MIREA. "Photosession" se desmorona en el 1er trimestre cm eran 50 GB de fotografías en blanco y negro.
Ahora, todas estas fotografías individuales tenían que combinarse de alguna manera en una imagen completa. Casi el mismo día escribí un programa en Python que generaba un archivo HTML; cuando lo abrí en un navegador, obtuve lo que necesitaba. (Por cierto, la décima Opera más antigua lo hizo mejor, ¡lo recomiendo!) Luego escribí otro programa en javascript que le permite comparar capas, hacer una transición suave entre ellas, alinearlas, seleccionar la escala, etc. Finalmente, en mis manos existían todas las herramientas para resolver los principales problemas. Seguí los caminos que perforaron el chip y restauré toda su estructura hasta el último transistor.
Otra fotografía de una rodaja de chip tomada bajo una radiografía (en MIREA):
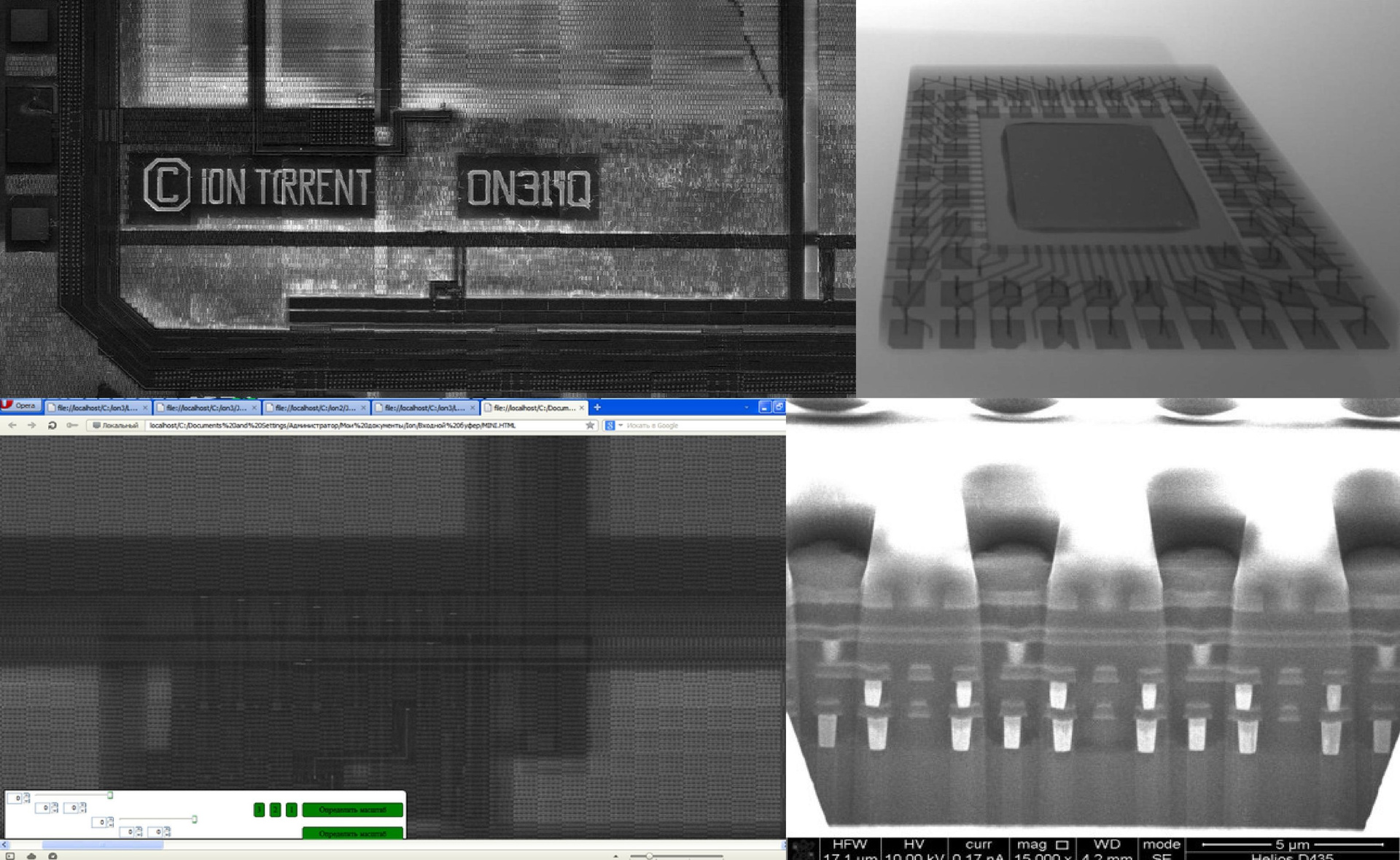 Fig. 7. Microscopía electrónica.
Fig. 7. Microscopía electrónica.Los pozos son visibles donde caen las esferas con lecturas. Debajo hay tres capas de metal, e incluso más abajo, una capa con transistores.
La siguiente etapa en la lucha por un futuro más brillante fue la creación de una placa base para el chip. Lo diseñó y envió una orden de producción. Mientras tanto, el tribunal y el caso utilizaron la placa Mars Rover-2 con FPGA para trabajar con el chip. (FPGA es, en términos generales, una matriz de 10,000 elementos lógicos universales; al programar FPGA, podemos obtener cualquier lógica que pueda procesar fácilmente flujos de información de gigabits). Escribí el firmware para FPGA y, además, escribí software para el control dinámico del sistema. que establece toda la configuración para FPGA. Luego reapareció un descanso de medio año (se fue de viaje de negocios al lago Baikal, preparó una planta en el laboratorio, que se lo demostró a Putin). Pero al final las estrellas se unieron: tuve tiempo, llegaron las tablas preparadas y ensamblé mi sistema.
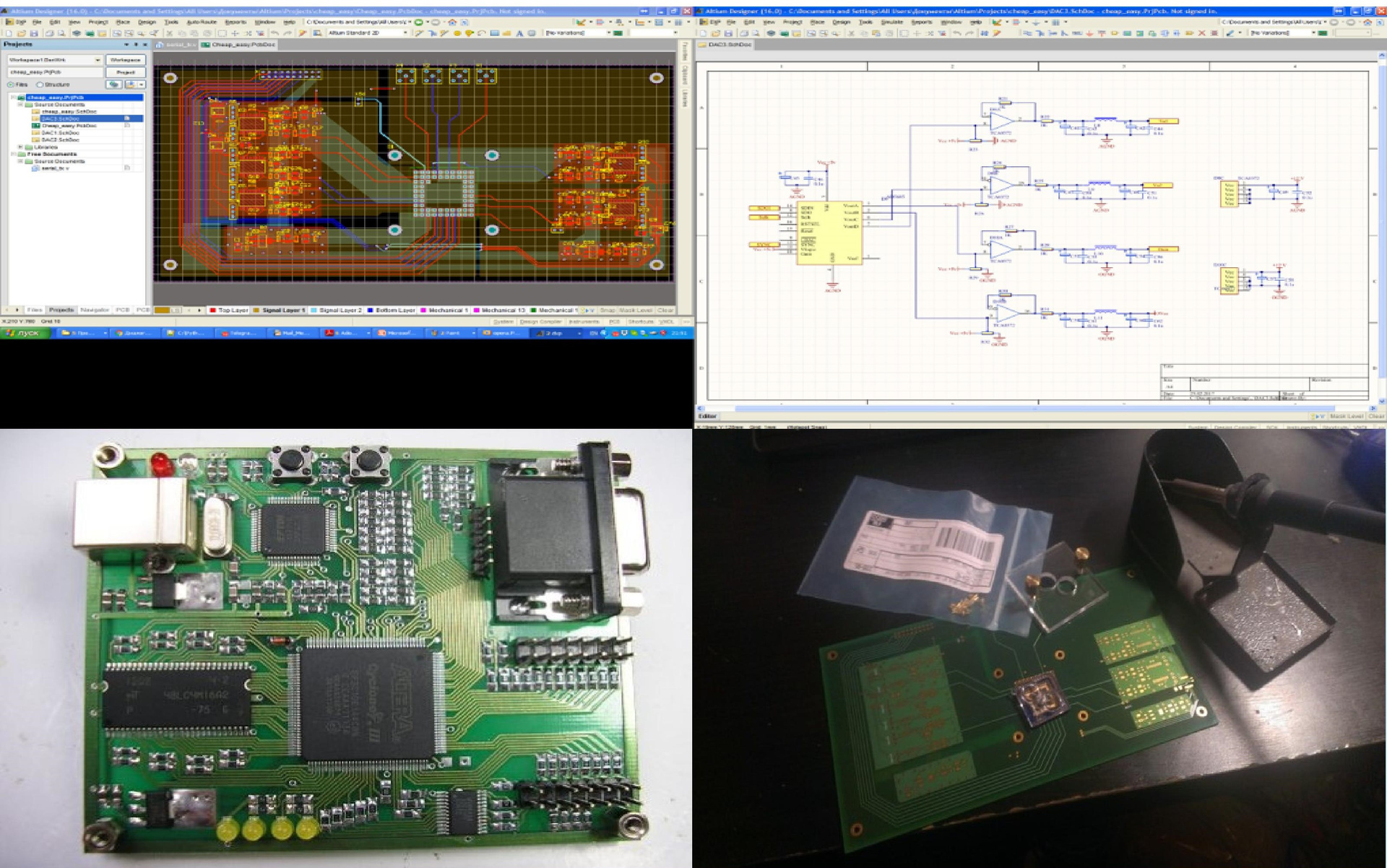 Fig. 8. La creación del "hierro"
Fig. 8. La creación del "hierro"Dio todas las señales necesarias y, ¡oh, un milagro! - Vi una señal del chip en el osciloscopio. (Una vez compré un osciloscopio por 6,000 rublos en eBay, otros 1,000 costos de firmware para él). Las manchas son claramente visibles en la imagen: gotas de algún reactivo.
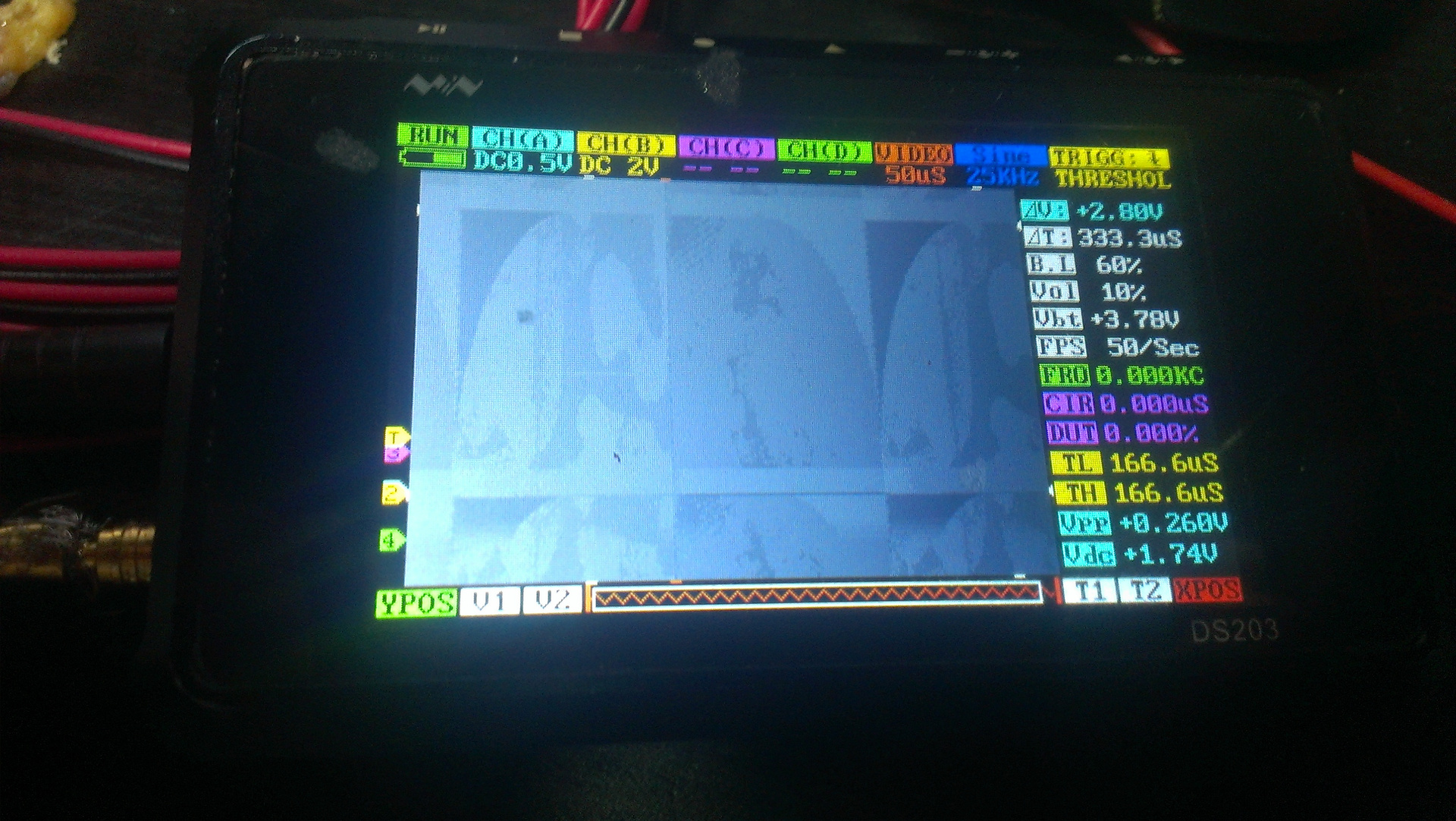 Fig. 9. La señal del chip en el osciloscopio.
Fig. 9. La señal del chip en el osciloscopio.Ahora necesitaba descubrir cómo digitalizar esta imagen y transferirla a una computadora. Arme esta configuración:
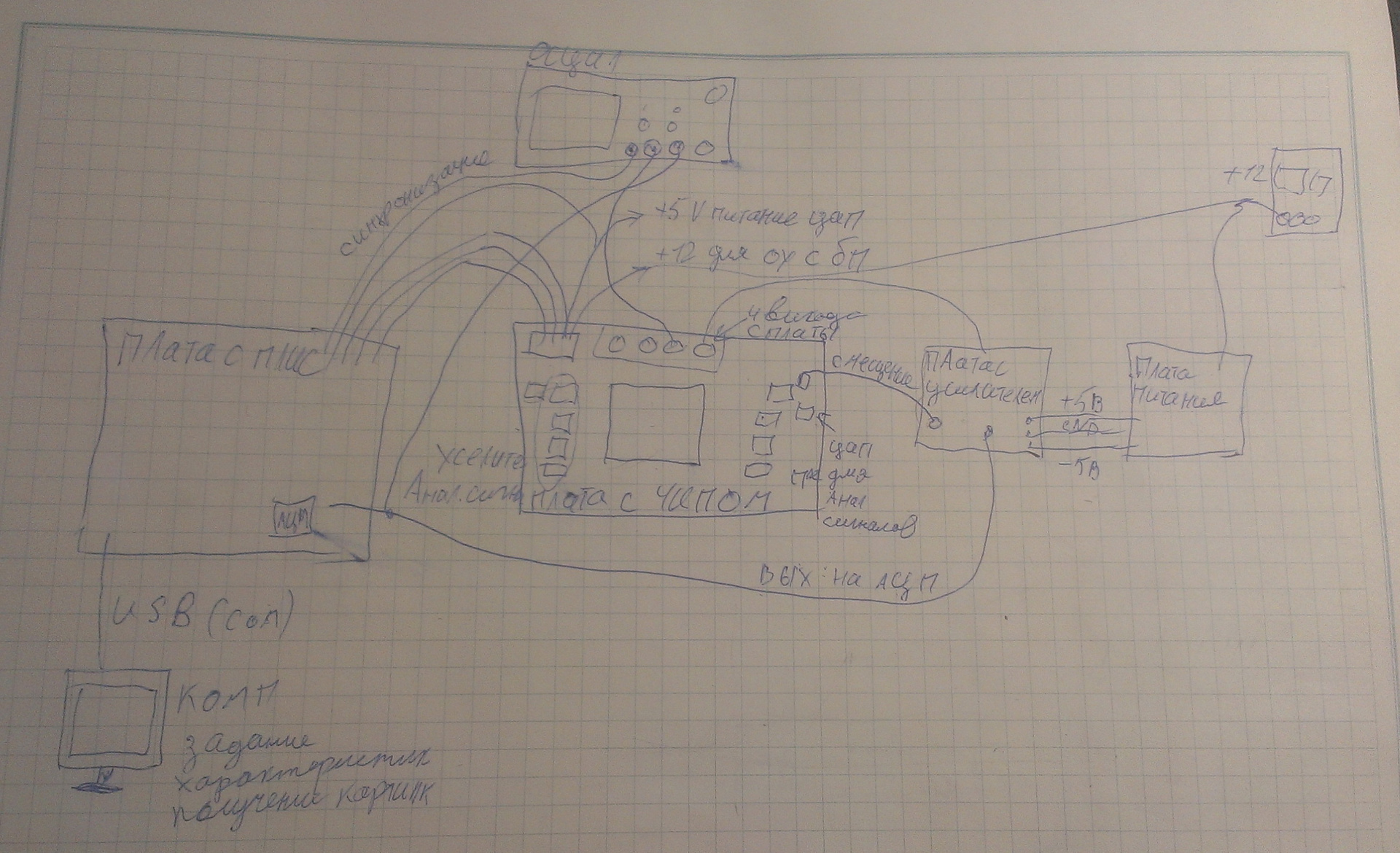 Fig.10. Circuito de instrumentos
Fig.10. Circuito de instrumentos Fig. 11. Instalación lista
Fig. 11. Instalación listaHay una computadora que suministra datos de control a la placa con FPGA. La placa genera señales digitales y las envía al chip. La señal del chip va al amplificador, luego al ADC en la placa, se digitaliza y se transmite a través del puerto COM a la computadora. En general, el ancho de banda del puerto COM es pequeño: 15 kilobits por segundo (ya que hay de 1 millón a 10 millones de "píxeles" en un chip, y la velocidad máxima de transmisión es de 115200 baudios). Sin embargo, la imagen finalmente llega a la computadora.
 Fig. 12. La señal procesada en la computadora.
Fig. 12. La señal procesada en la computadora.La foto de arriba muestra que cuando se suministra una biblioteca de ADN al chip usado usado, el chip se llena de manera desigual: hasta los bordes, en menor medida. Los diferentes colores se deben a diferentes tensiones en los transistores de pH. Es decir, podemos distinguir claramente aquellos agujeros donde golpean las esferas con lecturas; más adelante esto nos ayudará a controlar el enjuague del chip.
En consecuencia, la siguiente tarea fue vaciar el chip. Era necesario asegurarse de que se volviera como nuevo. Afortunadamente, tenía un chip completamente nuevo como muestra de referencia. En la fig. Y se puede ver que en la región activa dicho chip es casi del mismo color (las rayas repetitivas verticales son solo ruido, interferencia).
 Fig. 13. Enjuagar el chip
Fig. 13. Enjuagar el chipEn la fig. 13 B El chip no se lava bien, es multicolor. En la Figura 13, D es un chip usado pero bien lavado. Se ve que el gradiente a lo largo de los bordes ha desaparecido. Sin embargo, valdría la pena demostrar que está realmente limpio y puede reutilizarse.
Dado que las bibliotecas de ADN se unen al revestimiento de tantalio del chip en un entorno ácido y se separan en alcalinas (es decir, a un pH alto), el chip se lava con pipetas semiautomáticas especiales con soluciones con pH diferente. Hasta la fecha, logré lograr una limpieza casi completa del chip.
Me interesaba saber por qué, cuando entendí completamente la estructura del chip, no ordené su fabricación, sino que preferí continuar buscando y acostumbrarme a ellos, jugar con su lavado, etc. Sí, porque el desarrollo del microcircuito Cuesta mucho dinero, millones de dólares, y una parte sustancial de esta cantidad se destina a la depuración física del producto resultante: ajuste, ajuste de todos los parámetros de los transistores, etc. Es decir, simplemente copiar el circuito lógico no es suficiente. Por lo tanto, tomo microcircuitos condicionalmente gratuitos, listos para usar, diseñados, fabricados, depurados y, por lo tanto, ahorro dinero significativo, abaratando seriamente el proyecto.
Mi siguiente tarea fue ensamblar un dispositivo más avanzado, lo que permitiría una transferencia de información más rápida a una computadora y al mismo tiempo no consistiría en una gran cantidad de placas separadas.
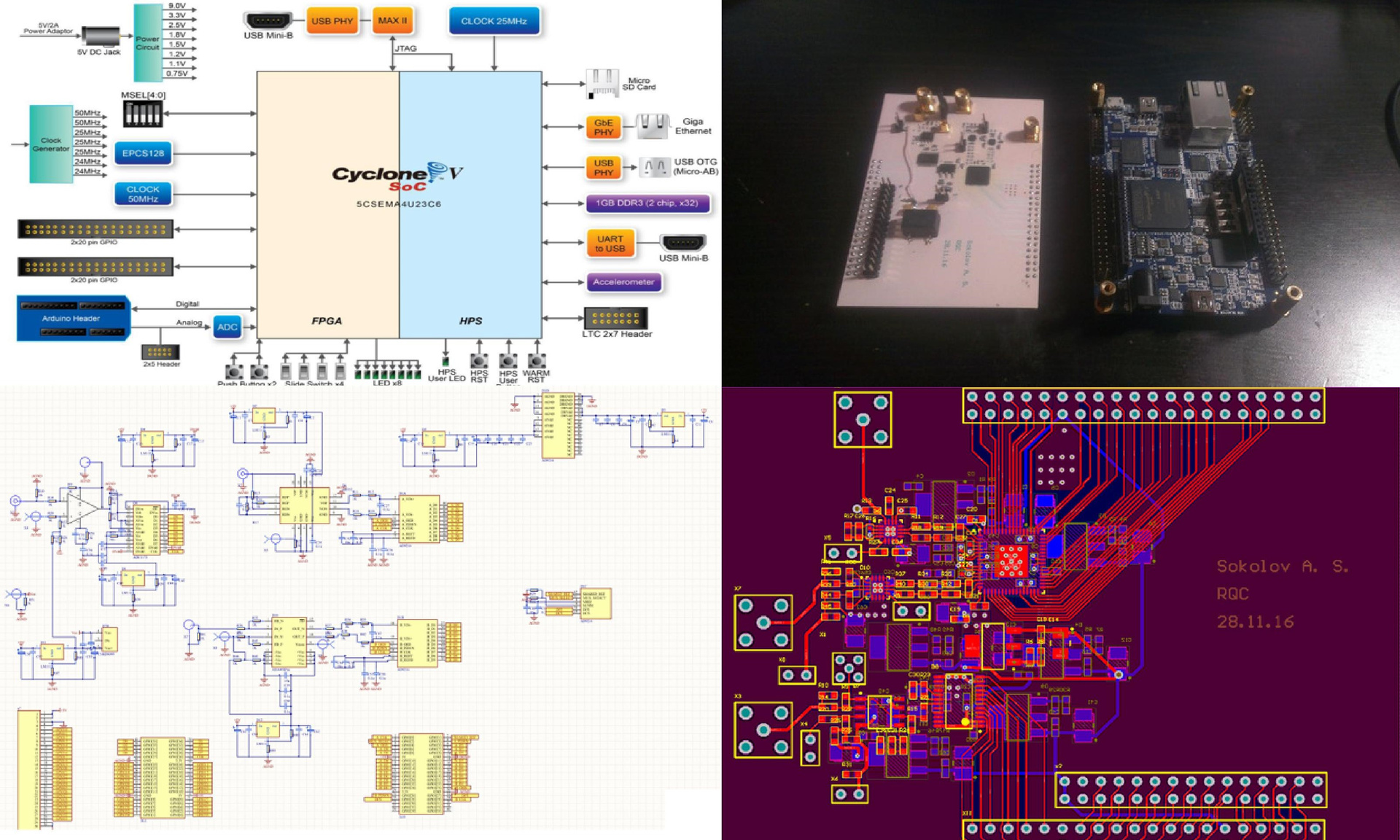 Fig. 14 Desarrollo de la próxima versión del dispositivo.
Fig. 14 Desarrollo de la próxima versión del dispositivo.Tomé una nueva placa base con FPGA: en el mismo chip había 2 núcleos ARM con Linux, Gigabit Ethernet y otras "ventajas", pero, a diferencia de la versión anterior, no había ADC. Más tarde, diseñó otra placa, con ADC de alta velocidad y todos los demás elementos necesarios. Lanzado: todo funcionó.
¿Qué queda por hacer para la aparición del dispositivo final? Solo tres cosas.
El primero Necesita gigabit de internet, transferencia de datos rápida a una computadora. Me di cuenta de esto ayer.
El segundo Sistema de suministro de reactivos. El diseño de una válvula especial ya está en progreso.
El tercero Software para procesar información del chip. Todavía tengo preguntas con el software, por lo que invito a los programadores a colaborar.
El dispositivo final cuesta 10 millones de rublos. El costo de la secuencia es de varios miles de dólares. Los chips cuestan de $ 100 a $ 1,000, dependiendo de la cantidad de "píxeles" que contengan. (Por cierto, la restauración de chips en sí misma puede ser una buena forma de ganar dinero, especialmente teniendo en cuenta que solo necesita hacer un par de clics para descargar). Los reactivos también se compran, pero en el futuro se crearán.
En general, todo esto es muy interesante, pero lo principal es el futuro. Hoy, la biotecnología ocupa el mismo lugar en el progreso científico y tecnológico mundial que la tecnología informática en los años 80. siglo pasado Además, la secuenciación es una de las áreas clave para la biología y la medicina modernas. Y, por supuesto, la biotecnología es muy rentable.
Recientemente, el secuenciador de semiconductores S5 ha aparecido en el mercado, y en un futuro próximo planeo cambiarlo.
¡Estaré encantado de conversar con todos los que quieran participar de una forma u otra en el desarrollo de este proyecto!
El proyecto no hubiera sido posible sin la formación teórica de
Vladimir Zubov . Le expreso mi gratitud.
Gracias por su atencion!