
Al escribir un artículo sobre el desarrollo de un detector de anomalías, implementé uno de los algoritmos llamado Gas neuronal de crecimiento incremental.
En Literatura soviética En el segmento ruso de Internet, este tema está cubierto bastante mal, y solo hubo un artículo , e incluso entonces, con la aplicación de este algoritmo.
Entonces, ¿qué es exactamente un algoritmo de gas neuronal en crecimiento incremental?
Introduccion
IGNG, como GNG , es un algoritmo de agrupamiento adaptativo.
El algoritmo en sí mismo se describe en un artículo de Prudent y Ennadji para 2005 .
Al igual que GNG, hay muchos vectores de datos. X o función generadora f(t) , que proporciona vectores a partir de datos distribuidos aleatoriamente (parámetro t - hora o número de muestra en la muestra).
El algoritmo no impone restricciones adicionales en estos datos.
Pero por dentro es muy diferente de GNG.
Este algoritmo también es interesante porque es un poco más preciso que los modelos GNG de neurogénesis .
Descripción del algoritmo
El algoritmo divide muchos datos en grupos.
En comparación con GNG, su ventaja es una mayor tasa de convergencia.
Ideas en las que se basa el algoritmo:
- Teoría de resonancia adaptativa : Primero, se busca la neurona más cercana, y si la diferencia no excede el umbral (el "parámetro de vigilancia"), se ajustan los pesos o, de lo contrario, se cambian las coordenadas de las neuronas en el espacio de datos. Si no se ha superado el umbral, se crean nuevas neuronas que se aproximan mejor al valor de la muestra de datos.
- Tanto las conexiones como las neuronas tienen un parámetro de edad (GNG solo tiene conexiones), que al principio es cero, pero aumenta a medida que aprende.
- Una neurona no aparece inmediatamente: primero aparece un embrión (o neurona germinal), cuya edad aumenta con cada iteración, hasta que madura. Después del entrenamiento, solo las neuronas maduras participan en la clasificación .
Ciclo principal
El trabajo comienza con un gráfico vacío. Parámetro sigma Inicializado por la desviación estándar de la muestra de entrenamiento:
sigma= sqrt frac1N sum limitsNi=1 left(xi− barx right)2
Donde: barx - el promedio entre las coordenadas de la muestra.
El bucle principal en cada paso disminuye el valor sigma , que es el umbral de proximidad, y calcula la diferencia entre el nivel anterior de calidad de agrupación y el nivel que se obtuvo después de la agrupación mediante el procedimiento IGNG .
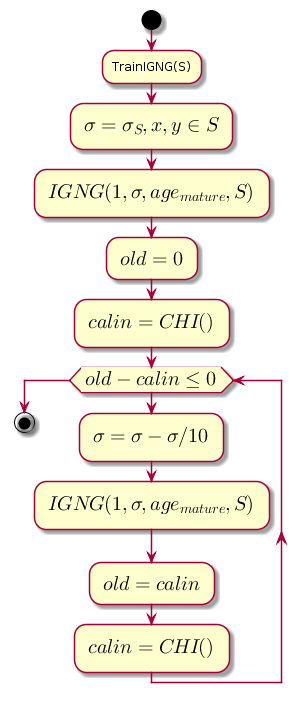
Código gráfico@startuml start :TrainIGNG(S); :<latex>\sigma = \sigma_S,x,y \in S</latex>; :<latex>IGNG(1, \sigma, age_{mature}, S)</latex>; :<latex>old = 0</latex>; :<latex>calin = CHI()</latex>; while (<latex>old - calin \leq 0</latex>) :<latex>\sigma=\sigma - \sigma / 10</latex>; :<latex>IGNG(1, \sigma, age_{mature}, S)</latex>; :<latex>old = calin</latex>; :<latex>calin = CHI()</latex>; endwhile stop @enduml
CHI es el índice Kalinsky-Kharabaz, que muestra la calidad de la agrupación:
CHI= fracB/(c−1)W/(n−c)
Donde:
- n - el número de muestras de datos.
- c - el número de grupos (en este caso, el número de neuronas).
- B - matriz de dispersión interna (la suma de las distancias al cuadrado entre las coordenadas de las neuronas y el promedio de todos los datos).
- W - matriz de dispersión externa (la suma de las distancias al cuadrado entre todos los datos y la neurona más cercana).
Cuanto mayor sea el valor del índice, mejor, porque si la diferencia entre los índices después de la agrupación y antes de que sea negativa, es posible suponer que el índice se volvió positivo y excedió el anterior, es decir. agrupación completada con éxito.
Procedimiento IGNG
Este es el procedimiento básico del algoritmo.
Se divide en tres etapas mutuamente excluyentes:
- No se encontraron neuronas.
- Se encontró una neurona satisfactoria.
- Encontramos dos que satisfacen las condiciones de la neurona.
Si una de las condiciones pasa, los otros pasos no se realizan.
Primero, se busca en una neurona la mejor muestra de datos aproximados:
c1=min(dist( xi, omegac))
Aqui dist(x omega,x xi) - función de cálculo de distancia, que suele ser una métrica euclidiana .
Si no se encuentra la neurona, o está demasiado lejos de los datos, es decir no cumple el criterio de proximidad dist( xi, omegac) leq sigma , se crea una nueva neurona embrionaria con coordenadas iguales a las coordenadas de la muestra en el espacio de datos.

Si la verificación de proximidad ha pasado, se busca una segunda neurona de la misma manera y se verifica la proximidad a la muestra de datos.
Si no se encuentra la segunda neurona, se crea.

Si se encuentran dos neuronas que satisfacen la condición de proximidad a la muestra de datos, sus coordenadas se corrigen de acuerdo con la siguiente fórmula:
epsilon(t)hc,ci= begincases epsilonb, ifc=ci epsilonn, ifhaylaconexiónentrec=ci0,inothercase endcases
Delta omegac= epsilon(t)hc,c1 parallel xi− omegac parallel omegac= omegac+ Delta omegac
Donde:
- epsilon(t) - paso de adaptación.
- ci Es el número de la neurona.
- hc,c1 - Función de Neuron Neighborhood c con la neurona ganadora (en este caso, devolverá 1 para vecinos directos, 0 de lo contrario, porque el paso de adaptación, para el cálculo omega será distinto de cero solo para vecinos directos).
En otras palabras, la coordenada (peso) de la neurona ganadora se cambia a epsilonb∗ Delta omegai , y todos sus vecinos directos (aquellos conectados con él por un borde del gráfico) en epsilonn∗ Delta omegai donde omegai - la coordenada de la neurona correspondiente antes del cambio.
Luego se crea una conexión entre las dos neuronas ganadoras, y si ya se ha creado, su edad se restablece.
La edad de todas las demás relaciones está aumentando.
Todas las comunicaciones cuya edad excedió la constante agemax son eliminados
Después de eso, se eliminan todas las neuronas maduras aisladas (aquellas que no tienen conexión con otras).
La edad de las neuronas inmediatas vecinas a la neurona ganadora está aumentando.
Si la edad de cualquiera de las neuronas de la línea germinal excede agemature Se convierte en una neurona madura.
El gráfico final contiene solo neuronas maduras.
Una condición para completar el procedimiento IGNG a continuación puede considerarse un número fijo de ciclos.
El algoritmo se muestra a continuación (se puede hacer clic en la imagen):
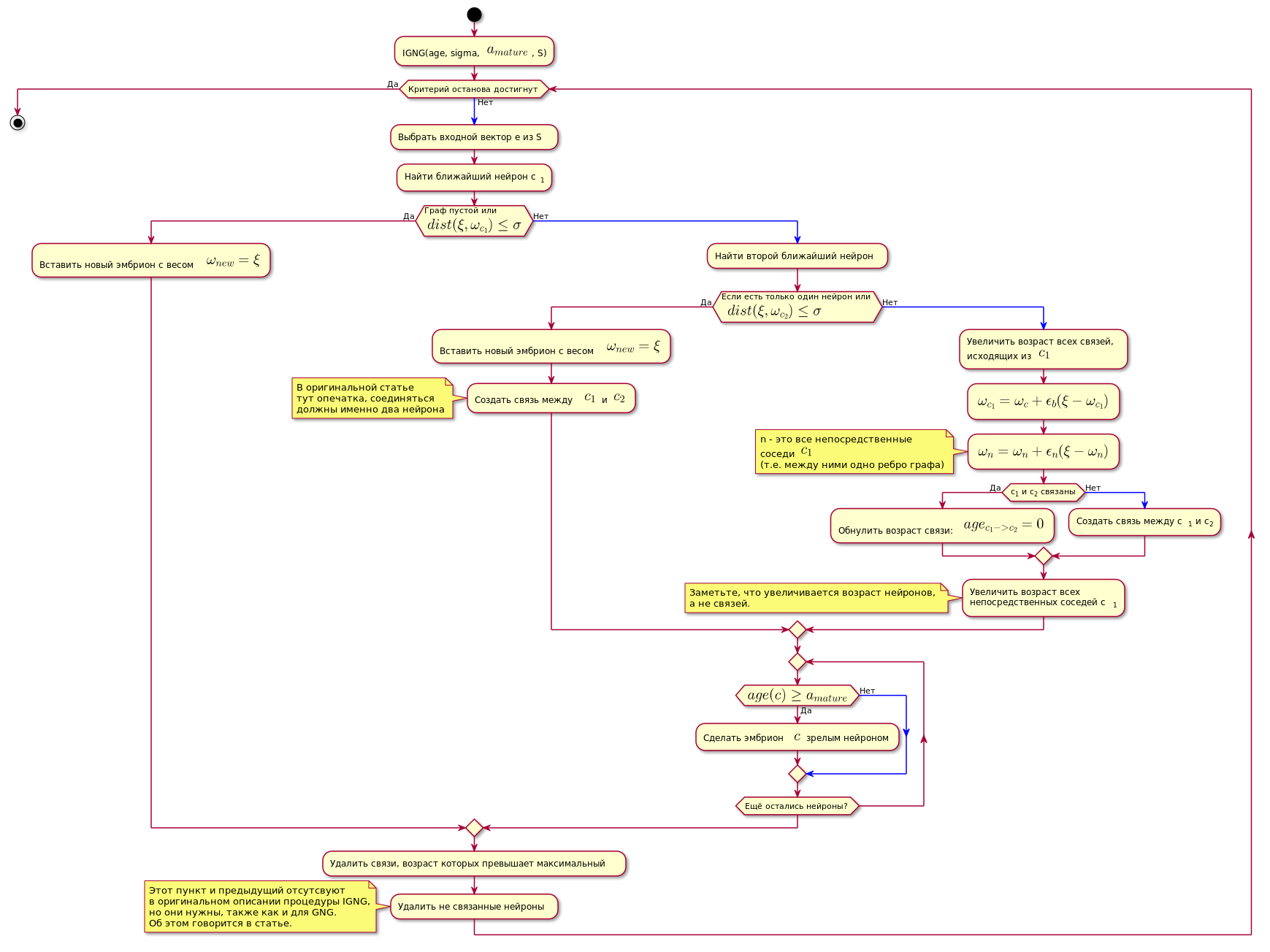
Código gráfico @startuml skinparam nodesep 10 skinparam ranksep 20 start :IGNG(age, sigma, <latex>a_{mature}</latex>, S); while ( ) is () -[#blue]-> : e S; : c<sub>1</sub>; if ( \n<latex>dist(\xi, \omega_{c_1}) \leq \sigma</latex>) then () : <latex>\omega_{new} = \xi</latex>; else () -[#blue]-> : ; if ( \n <latex>dist(\xi, \omega_{c_2}) \leq \sigma</latex>) then () : <latex>\omega_{new} = \xi</latex>; : <latex>c_1</latex> <latex>c_2</latex>; note , end note else () -[#blue]-> : ,\n <latex>c_1</latex>; :<latex>\omega_{c_1} = \omega_c + \epsilon_b(\xi - \omega_{c_1})</latex>; :<latex>\omega_n = \omega_n + \epsilon_n(\xi - \omega_n)</latex>; note n - <latex>c_1</latex> (.. ) end note if (c<sub>1</sub> c<sub>2</sub> ) then () : : <latex>age_{c_1 -> c_2} = 0</latex>; else () -[#blue]-> : c<sub>1</sub> c<sub>2</sub>; endif : \n c<sub>1</sub>; note , , . end note endif repeat if (<latex>age(c) \geq a_{mature}</latex>) then () : $$ ; else () -[#blue]-> endif repeat while ( ?) endif : , ; : ; note IGNG, , GNG. . endnote endwhile () stop @enduml
Implementación
La red se implementó en Python usando la biblioteca de gráficos NetworkX . A continuación, se muestra el corte del código del prototipo en el artículo anterior . También hay breves explicaciones para el código.
Si alguien está interesado en el código completo, aquí hay un enlace al repositorio .
Un ejemplo del algoritmo:

La mayor parte del código class NeuralGas(): __metaclass__ = ABCMeta def __init__(self, data, surface_graph=None, output_images_dir='images'): self._graph = nx.Graph() self._data = data self._surface_graph = surface_graph