Hola habr Hoy continuamos la última historia sobre el ADN. En él, hablamos sobre cuánto
sucede, cómo se almacena el ADN y por qué es tan importante . Hoy comenzaremos con un trasfondo histórico y terminaremos con los conceptos básicos de la codificación de información en el ADN.
La historia
El ADN en sí fue aislado en 1869 por Johann Friedrich Mischer de los glóbulos blancos que recibió del pus. Los glóbulos blancos son glóbulos blancos que realizan una función protectora. Hay bastantes de ellos en el pus, porque tienden a dañar los tejidos, donde las células bacterianas "comen". Aisló una sustancia que contiene nitrógeno y fósforo. Al principio se llamaba nucleina, sin embargo, cuando descubrió propiedades ácidas, el nombre se cambió a ácido nucleico. La función biológica de la sustancia recién descubierta no estaba clara, y durante mucho tiempo se creyó que el fósforo estaba almacenado en ella. Incluso a principios del siglo XX, muchos biólogos creían que el ADN no tenía nada que ver con la transferencia de información, ya que la estructura de la molécula, como parecía entonces, era demasiado monótona y no podía codificar tanta información.
Para 1901, Albrecht Kossel aisló y describió las cinco bases nitrogenadas que forman el ADN y el ARN. Y un poco más tarde, Peter Leuven descubrió que el componente de carbohidratos de los ácidos nucleicos son la desoxirribosa y la ribosa. Los ácidos nucleicos, que incluyen ribosa, se denominaron ácidos ribonucleicos o, en resumen, ARN, y los que contenían desoxirribosa, ácidos desoxirribonucleicos o ADN.
Ahora, surgió la pregunta de cómo se interconectan los enlaces individuales. Para esto, la cadena de ADN necesitaba ser destruida y ver qué sucedería después de la destrucción. Para esto, el polímero de ADN se hidrolizó. Sin embargo, Leuven cambió el método de hidrólisis. Ahora, en lugar de hervir durante muchas horas en un ambiente ácido, usaba enzimas. Esta vez, no solo se aislaron adenina, guanina, timina, citosina, desoxirribosa y ácido fosfórico individuales de los hidrolizados, sino también fragmentos más grandes, por ejemplo, compuestos de bases nitrogenadas con carbohidratos o carbohidratos con ácido fosfórico. Al mismo tiempo
, los compuestos que consisten en dos bases nitrogenadas o compuestos del tipo ácido base-fosfórico no se encontraron en los hidrolizados de ácido nucleico . Es decir, quedó claro que el ácido fosfórico se combina con el azúcar y, a su vez, con una base nitrogenada. Se propuso que los compuestos de bases nitrogenadas con un carbohidrato se llamaran nucleósidos, y los ésteres fosfóricos de nucleósidos se llamaran nucleótidos.
Como resultado de estos trabajos, Leuven concluyó que los ácidos nucleicos son polímeros. Los nucleótidos sirven como monómeros. El contenido de cada uno de los cuatro nucleótidos en el ADN, o ARN, según el análisis químico de esa época, parecía igual para Leven. Por lo tanto, Leuven propuso la siguiente teoría de la estructura de los ácidos nucleicos: son polímeros cuyos monómeros son bloques de cuatro nucleótidos conectados en serie.
La teoría de la estructura de tetranucleótidos en ese momento parecía bastante justificada, después de haber ingresado a todos los libros de texto del período anterior a la guerra. Sin embargo, la cuestión de la función del ADN seguía sin estar clara. Me llevó casi medio siglo aclarar este problema.
Llegó un período durante el cual los biólogos acumularon información sobre la distribución de ácidos nucleicos en varios tipos de tejidos animales y vegetales, en bacterias y virus, en algunos organismos unicelulares.
En ese momento, la comunidad científica creía seriamente que las proteínas eran las responsables del almacenamiento de la información genética. La idea tradicional del papel principal de las proteínas en el proceso de la vida no nos permitió pensar que una sustancia tan importante como una sustancia de la herencia podría ser cualquier cosa menos proteína. Las proteínas tenían una estructura extremadamente diversa, que entonces no podía decir sobre los ácidos nucleicos. El famoso genetista-citólogo soviético N.K. Koltsov calculó que al variar la secuencia de 20 aminoácidos que componen la molécula de proteína, se pueden crear billones de proteínas diferentes.
Si quisiéramos imprimir de la forma más simplificada cómo se imprimen las tablas logarítmicas, este trillón de moléculas proporcionaría todas las imprentas existentes en el mundo para este plan, produciendo 50,000 volúmenes de 100 hojas impresas por año, entonces habría pasado tanto tiempo antes del final del trabajo realizado, cuánto ha pasado desde el período Arqueano de nuestros días.
Realmente mucho ... 20 en el 20 ... Pero las secuencias son mucho más largas que 20 aminoácidos.
Y aquí está lo que A. R. Kizel escribe sobre esto, uno de los bioquímicos más eruditos de la época.
A partir de las opiniones que se acaban de dar sobre el papel del ácido nucleico ... se deduce que no está involucrado en la estructura de los genes y se deduce que los genes están formados por algún otro material. Todavía no conocemos este material de manera confiable, a pesar del hecho de que en la mayoría de los casos se llama directamente proteína.
El primer éxito vino de la microbiología. En 1944, se publicaron los resultados de los experimentos de Avery y empleados (EE. UU.) Sobre la transformación de bacterias. Algunas palabras sobre la transformación.
La transformación en sí fue descubierta en 1928 por el microbiólogo Griffiths.
Griffith trabajó con cultivos de neumococo (Streptococcus pneumoniae), el agente causal de una de las formas de neumonía. Algunas cepas de esta bacteria son virulentas y causan neumonía. Sus células están recubiertas con una cápsula de polisacárido que protege a la bacteria de la acción del sistema inmune. En cultivo, tales bacterias forman grandes colonias lisas de forma esférica regular. Debido a esto, se llaman cepas S (del inglés liso - liso).
Hay varias cepas virulentas de neumococo, que difieren en los anticuerpos que se producen en el cuerpo cuando las bacterias ingresan. Se llaman IS, IIS, IIIS, etc. De vez en cuando, algunas células de cepas virulentas de S mutan, perdiendo la capacidad de sintetizar la membrana de polisacárido y se vuelven avirulentas. En la cultura, forman pequeñas colonias rugosas de forma irregular, por eso se les llama cepas R (del inglés rugoso - rugoso). A veces se producen mutaciones inversas, restaurando la capacidad de sintetizar la membrana de polisacárido, pero solo en grupos de las cepas correspondientes:
IIS - IIR
IIIS - IIIR
Esto sugiere que las cepas R avirulentas siempre corresponden a la cepa S virulenta original.
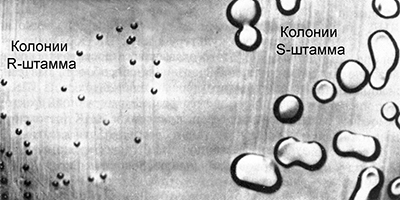
Griffith presentó a diferentes grupos de ratones de laboratorio una cepa virulenta y avirulenta de neumococo. En el primer grupo de control, la inyección de una cepa virulenta de IIIS condujo a la muerte de los animales. Los animales del segundo grupo de control permanecieron vivos después de la inyección de la cepa avirulenta IIR. Después de eso, Griffith calentó la solución con el cultivo de la cepa virulenta IIIS a una temperatura de 60 ° C, lo que condujo a la muerte de las bacterias. Introdujo bacterias muertas por calentamiento al tercer grupo de ratones experimentales. Los animales permanecieron vivos, lo que, en principio, se esperaba. Sin embargo, esto no es todo. Introdujo porciones de ratones supervivientes a las bacterias de la cepa avirulenta IIR.
Parecía que esto no podía tener consecuencias terribles para los ratones. Sin embargo, contrario a las expectativas, los animales murieron. Cuando las bacterias se aislaron de sus cuerpos y se sembraron en cultivo, resultó que pertenecían a la cepa virulenta IIIS.

El hecho de que las células que causaron la muerte de los ratones sintetizaron una membrana de polisacárido de tipo III en lugar de II indicó que no podrían haber surgido como resultado de la mutación inversa IIR - IIS. A partir de esto, Griffith hizo una conclusión muy importante. Las bacterias avirulentas de la cepa IIR pueden transformarse en virulentas de alguna manera interactuando con las bacterias muertas por calor de la cepa IIIS que aún permanecían en el cuerpo de los ratones. En otras palabras, las bacterias avirulentas de la cepa IIR reciben un factor de las bacterias muertas de la cepa IIIS que las convierte en virulentas. Sin embargo, cuál era este factor, Griffith no lo sabía.
En realidad, este fenómeno se llamó transformación bacteriana. Es una transferencia unidireccional de rasgos hereditarios de una célula bacteriana a otra.
Ahora volvamos a las experiencias de Avery. El diseño de sus experimentos es algo similar a los experimentos de Griffiths. Avery y el personal se propusieron la tarea de descubrir la naturaleza química del agente transformador. Destruyeron la suspensión de neumococos y eliminaron proteínas, polisacáridos capsulares y ARN del extracto, sin embargo, la actividad transformadora del extracto permaneció. La actividad transformadora del fármaco no se perdió durante su tratamiento con tripsina cristalina o quimotripsina (proteínas destructoras), ribonucleasa (destruye ARN). Estaba claro que el fármaco no era ni proteína ni ARN. Sin embargo, la actividad transformadora del fármaco se perdió por completo cuando se trató con desoxirribonucleasa (que daña el ADN), y cantidades insignificantes de la enzima causaron la inactivación completa del fármaco. Por lo tanto, se descubrió que el factor transformante en las bacterias es el ADN puro. Esta conclusión fue un descubrimiento significativo, y Avery lo sabía muy bien. Escribió que esto es exactamente con lo que la genética siempre ha soñado, es decir, la sustancia del gen. Aquí parece ser una prueba. Pero demasiado fuerte era la creencia en la proteína, como sustancia de la herencia. Algunos creían que la transformación puede causar y esas impurezas proteicas insignificantes que permanecieron en el medicamento.
La nueva evidencia del papel genético directo del ADN fue la experiencia de los virólogos Hershey y Chase. Trabajaron con el bacteriófago T2 (Bacteriófagos - virus de bacterias), que infecta la bacteria
Escherichia coli (E. coli).
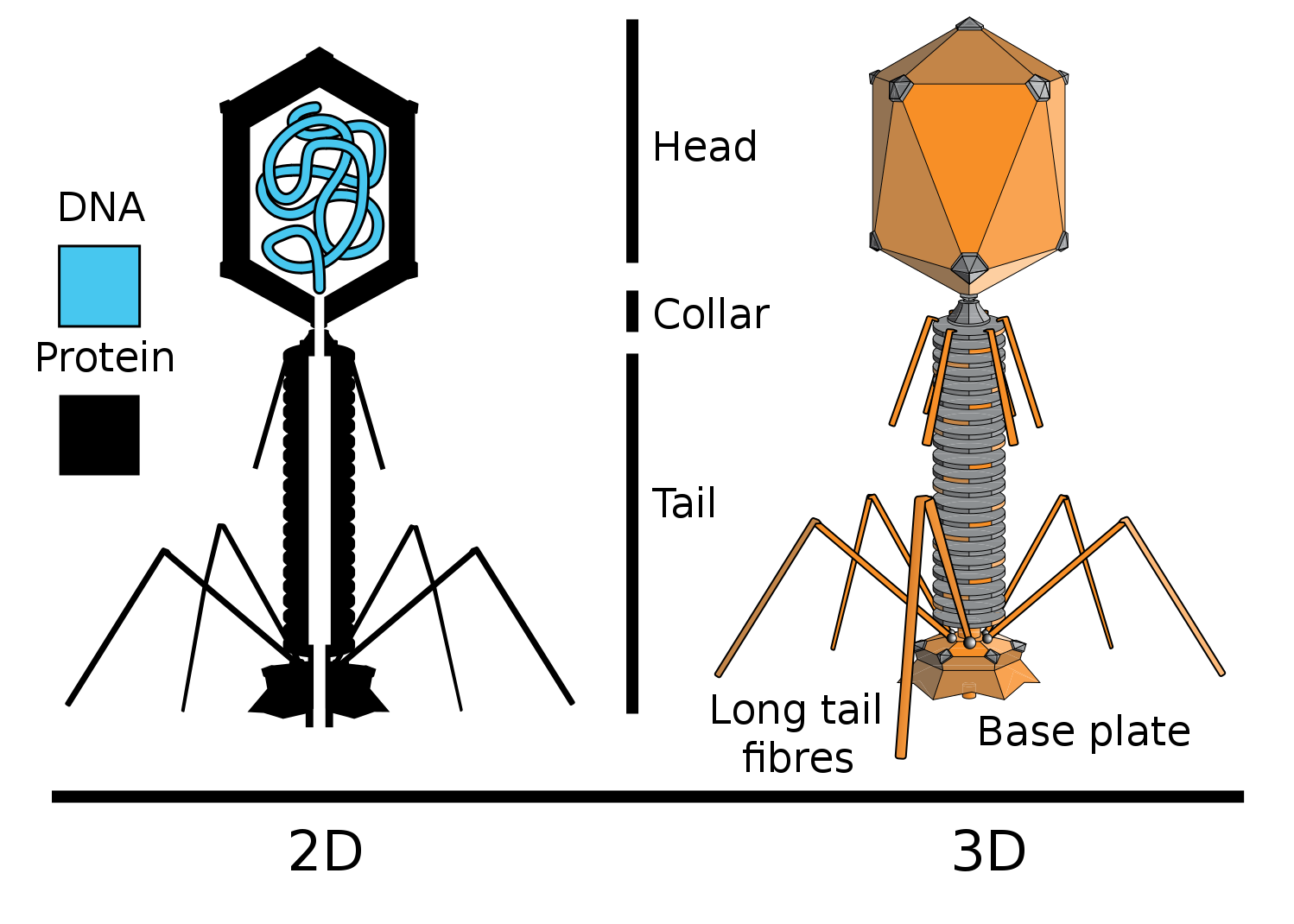
En realidad lo que hicieron. Incluyeron fósforo radiactivo en la composición del ADN de algunos bacteriófagos (P32), y el isótopo de azufre en la composición de las proteínas de otros (S35). Para esto, algunas bacterias se cultivaron en un medio con la adición de fósforo radiactivo en el ion fosfato, mientras que otras se cultivaron en un medio con la adición de azufre radiactivo en el ion sulfato. Luego, se añadió el bacteriófago T2 a estas bacterias, que, multiplicándose en células bacterianas, incluyó una etiqueta radiactiva en su ADN (P está en el ADN, pero no en las proteínas), o proteínas (S está en las proteínas, pero no en el ADN).
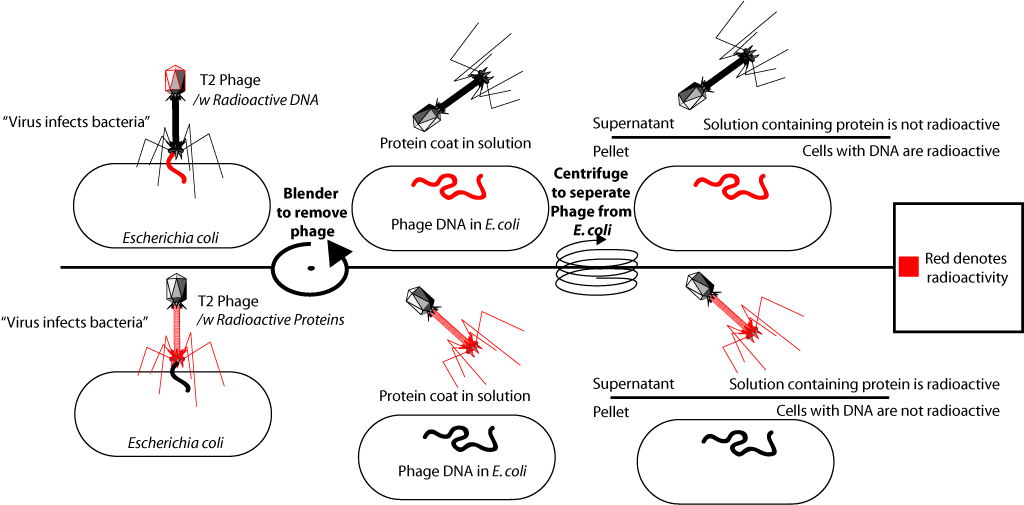
Después del aislamiento de los bacteriófagos marcados radiactivamente, se agregaron al cultivo de bacterias frescas (sin isótopos). Lo que condujo a la infección de estas bacterias. El bacteriófago se une a la célula bacteriana e "inyecta" su ADN. Después de esto, el medio con la bacteria se agitó vigorosamente en un mezclador especial (se demostró que las membranas del fago se separaron de la superficie de las células bacterianas), y luego las bacterias infectadas se separaron del medio. Cuando en el primer experimento se agregaron bacteriófagos marcados con fósforo 32 a las bacterias, resultó que el marcador radiactivo estaba en las células bacterianas. Cuando en el segundo experimento se agregaron bacteriófagos marcados con azufre-35 a las bacterias, la etiqueta se encontró en una fracción del medio con recubrimientos de proteínas, pero no estaba en las células bacterianas. Esto confirmó que el material que infectaba la bacteria era el ADN. Dado que las partículas virales completas que contienen proteínas virales se forman dentro de las bacterias infectadas, este experimento se ha convertido en una de las pruebas decisivas del hecho de que la información genética (información sobre la estructura de las proteínas) está contenida en el ADN.
Estos descubrimientos influyeron mucho en muchos biólogos de la época. En particular sobre el famoso por sus reglas, Chargaff. Él creía que Avery esencialmente descubrió un 'nuevo lenguaje', o al menos mostró dónde buscarlo.
Charguff comenzó a buscar una diferencia en la composición de nucleótidos y la disposición de los nucleótidos en las preparaciones de ADN obtenidas de diversas fuentes. Y, dado que no había métodos para dar con precisión la caracterización química del ADN, en ese momento ... tuvo que idearlos. Se le mostró que la antigua teoría de los tetranucleótidos de la estructura de los ácidos nucleicos es incorrecta. El ADN en diferentes organismos es muy diferente en composición y estructura. Al mismo tiempo, se descubrieron nuevos hechos que no se habían establecido previamente para otros polímeros naturales, a saber, la regularidad en la proporción de bases individuales en la composición de todo el ADN estudiado. Ahora, incluso los escolares los conocen como las reglas de Chargaff.
- La cantidad de adenina es igual a la cantidad de timina y guanina a citosina: A = T, G = Ts.
- El número de purinas es igual al número de pirimidinas: A + G = T + C.
- Fluye desde el primero y el segundo. El número de bases con grupos amino en la posición 6 es igual al número de bases con grupos ceto en la posición 6: A + C = T + G.
Nos referimos al mecanismo
en el último artículo , así que no me detendré aquí.
Poco a poco nos acercamos a dos personas legendarias que descubrieron la estructura del ADN. Francis Crick y James Watson se conocieron en 1951. Watson decidió entonces abordar la estructura del ADN. Como biólogo, entendió que al elegir una estructura de ADN específica, uno debe tener en cuenta la existencia de algún principio simple de duplicar la molécula de ADN incrustada en su estructura. De hecho, una de las propiedades más importantes de los genes es la transmisión de información hereditaria.
Crick creó la teoría de la difracción de rayos X por espirales, que permite determinar si la estructura en estudio tiene una conformación en espiral o no. En ese momento, ya existían radiografías de ADN. Fueron recibidos en Londres por Maurice Wilkins y Rosalind Franklin.
Por la naturaleza de los rayos X del ADN, Watson y Crick se dieron cuenta de que la estructura en estudio tenía una conformación en espiral. También sabían que una molécula de ADN es una cadena de polímero lineal larga que consiste en monómeros de nucleótidos. La cadena principal de fosfodesoxirribosa de este polímero es continua, y las bases nitrogenadas están unidas al lado de los residuos de desoxirribosa. Para construir los modelos, quedaba por resolver la cuestión de cuántas cadenas de polímero lineal se apilaban en una estructura compacta.
Con base en la radiografía del ADN en forma de B, Watson y Crick sugirieron que la molécula de ADN consta de dos cadenas de polinucleótidos lineales con un esqueleto de fosfodesoxirribosa fuera de la molécula y bases nitrogenadas en su interior. Lo que posteriormente se confirmó. Solo quedaba resolver la cuestión de la disposición de las bases nitrogenadas de las dos cadenas dentro de la espiral.
Considerando las posibles combinaciones de pares de bases nitrogenadas, Watson descubrió que los pares adenina - timina y guanina - citosina son del mismo tamaño y están estabilizados por enlaces de hidrógeno. Las reglas de Chargaff se explicaron de inmediato: si en el ADN de doble hélice, la adenina de una cadena siempre se conecta con la timina de otra cadena, y la guanina siempre se combina con la citosina, entonces la adenina en el ADN siempre debe ser tanto como la timina, y tanto la guanina cuánta citosina También estaba claro cómo debería producirse la duplicación de la molécula de ADN. Cada cadena es complementaria de la otra, y en el proceso de replicación del ADN, las cadenas de doble hélice deben separarse y cada cadena complementaria debe completarse en cada cadena de polinucleótidos. También hubo varias teorías, pero sobre ellas en una semana, en el siguiente artículo.
Información de codificación
Entonces, sabemos que el ADN es portador de información, sabemos en qué consiste. Pero la forma en que se codifica la información aún no está clara.
Vayamos de la tarea. El ADN codifica 20 aminoácidos (podemos decir que 21, pero hasta ahora no tocamos selenocystenin). Hay 4 opciones para los nucleótidos. Es decir, un nucleótido puede codificar 4 variantes, 2 - 16, 3 - 64. Es lógico suponer que el código es triplete (es decir, tres bases codifican un aminoácido). Puede leer sobre la confirmación experimental
aquí . Me temo que ya hay mucha historia ...
En realidad, tenemos 64 variantes y 20 aminoácidos. Los aminoácidos pueden ser codificados por diferentes codones. También hay codones de inicio y parada a partir de los cuales comienza la lectura.
No olvides que primero el ADN se lee en el ARN, con el que ya está leyendo en la proteína.
La siguiente tabla muestra la correspondencia de los codones de ARN con los aminoácidos. Recuerde que no hay timina en el ARN, sino que se usa uracilo.

Si no encontró el codón de inicio en la tabla, busque AUG. Codifica la metionina y también es la de partida. Al traducir los genes de procariotas, plastidios y genes mitocondriales, el aminoácido inicial es N-formilmetionina (esto es solo para referencia).
Si pinta desde el ADN hasta la proteína, obtenemos algo como esto.
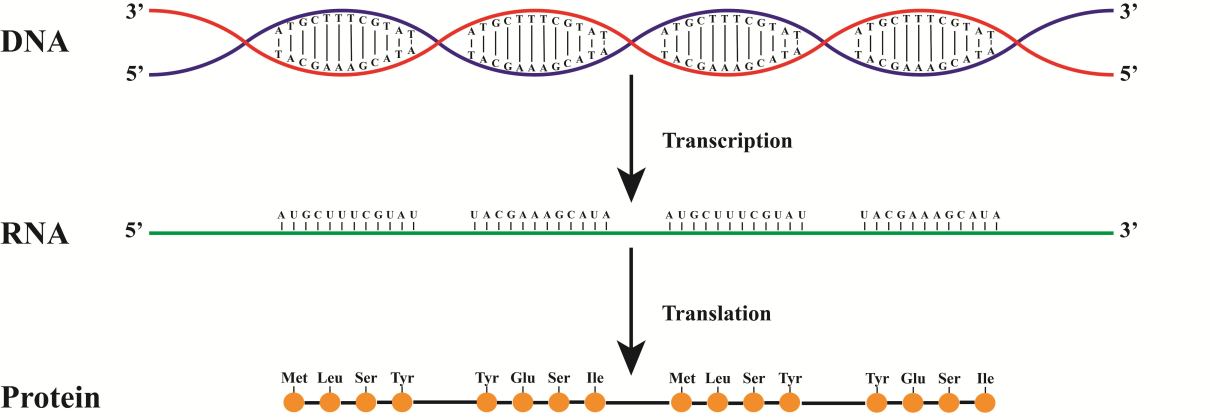
En esta figura, la síntesis es de la cadena roja. Como resultado, el ARN coincidirá con la cadena azul (no se olvide de reemplazar T con Y)
Como dije, varios codones pueden codificar cada aminoácido. A primera vista, esto no parece ser un efecto secundario particularmente necesario de la redundancia de codones. Pero en realidad tiene un papel bastante importante.
Aquí tocaremos un poco las mutaciones. Vienen en diferentes tipos. Desde los cromosomas, cuando se eliminan piezas enteras de los cromosomas del genoma, se intercambian, se duplican, para señalarlas, cuando una base nitrogenada se reemplaza por otra. Centrarse en mutaciones puntuales.
¿A qué pueden conducir las mutaciones puntuales?
El codón puede comenzar a codificar otro aminoácido, que no siempre da miedo. Dichas mutaciones se llaman mutatsimi sin sentido (es decir, con un cambio de significado). Esto puede afectar la estructura de la proteína.
Por ejemplo, si un aminoácido con carga positiva se reemplaza por uno con carga negativa, esto puede hacer que la proteína sea inestable o que se pliegue en otra conformación (sí, la secuencia lineal de aminoácidos generalmente se pliega en una determinada forma) y no puede cumplir sus funciones (o comienza a hacerlo). mejor, ya huele a evolución).Específicamente, la hemoglobina S tiene un cambio de nucleótido único (A a T) en el gen codificador. Como resultado, el triplete GAG que codifica el glutamato se reemplaza por la valina que codifica THG. La hemoglobina S también puede transportar oxígeno, pero lo hace peor que la hemoglobina regular.En la molécula de hemoglobina Hikari, la asparagina se sustituye por la lisina, sin embargo, todavía es bueno transferir oxígeno.Como ejemplo con la pérdida de función, considere la hemoglobina M. Otro punto de mutación en el gen de la hemoglobina conduce a la pérdida completa de la función (la histidina cambia a tirosina en el centro activo).Por cierto, el plegamiento de proteínas se ve así si omites todos los matices. ¿Qué más podría pasar?Reemplazar una base nitrogenada también puede conducir a la aparición de un codón de parada en el centro de la secuencia, o viceversa, el codón de parada al final desaparece. La salida será un circuito incompleto o un circuito extremadamente largo, que en cualquier caso no podrá funcionar normalmente. Tales mutaciones se llaman tonterías.
¿Qué más podría pasar?Reemplazar una base nitrogenada también puede conducir a la aparición de un codón de parada en el centro de la secuencia, o viceversa, el codón de parada al final desaparece. La salida será un circuito incompleto o un circuito extremadamente largo, que en cualquier caso no podrá funcionar normalmente. Tales mutaciones se llaman tonterías.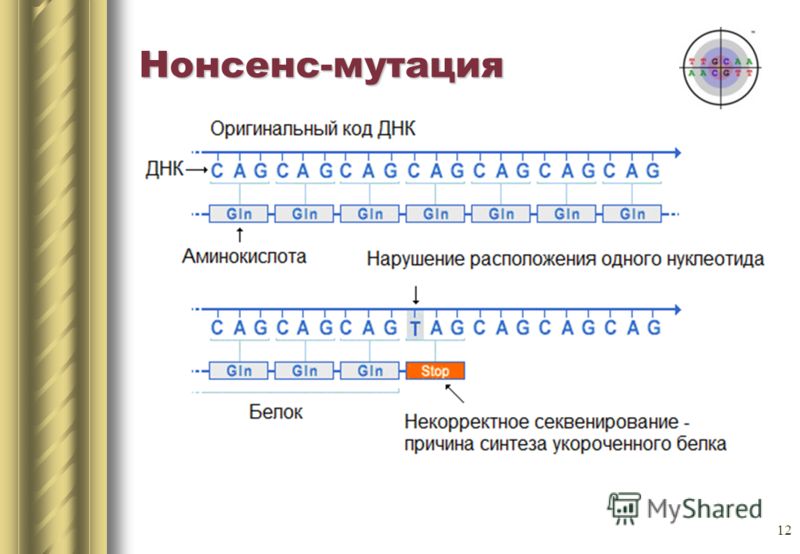 Hay un tercer tipo de mutación: la mutación silenciadora. De hecho, el codón se reemplaza por otra codificación para el mismo aminoácido. Las propiedades de la proteína no cambian.Para resumir el esquema general.
Hay un tercer tipo de mutación: la mutación silenciadora. De hecho, el codón se reemplaza por otra codificación para el mismo aminoácido. Las propiedades de la proteína no cambian.Para resumir el esquema general.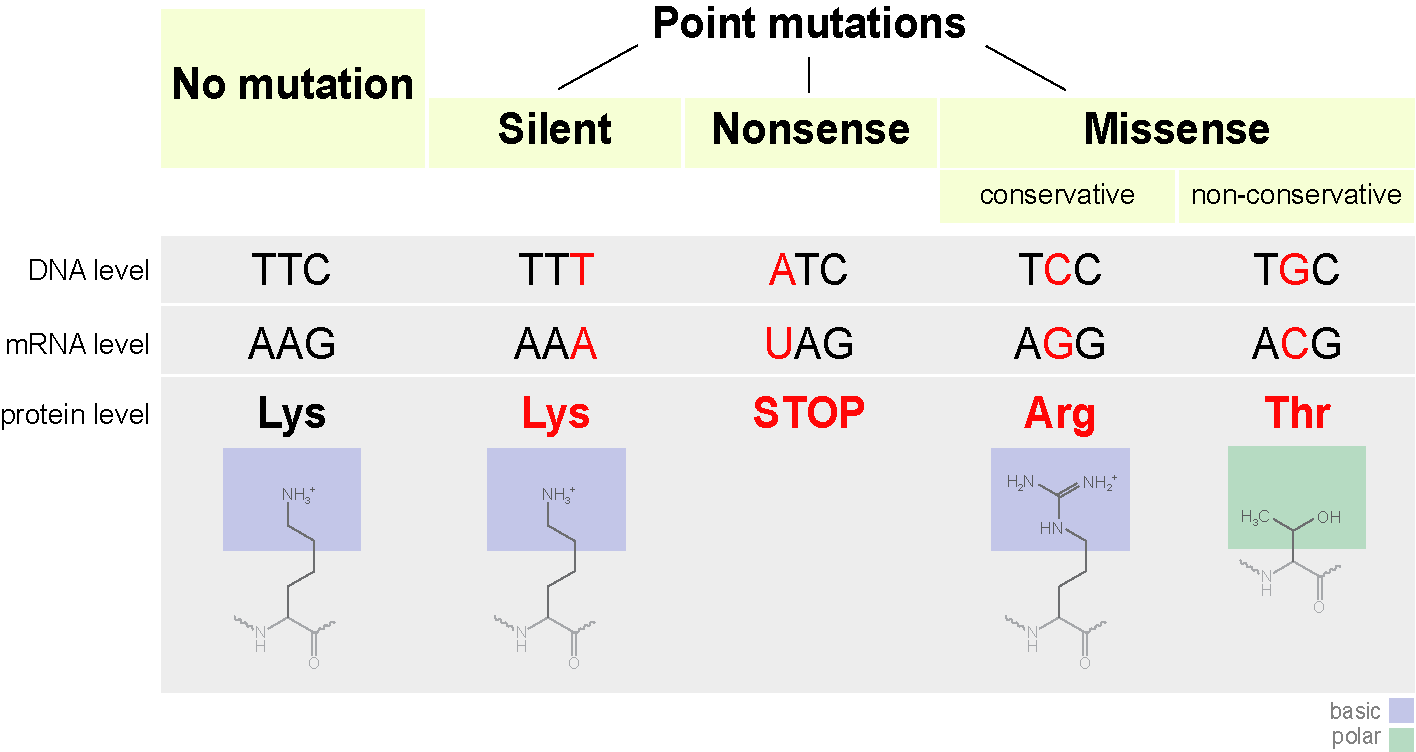 En conclusión, también me gustaría hablar sobre una característica interesante. Un solo aminoácido puede ser codificado por varios codones. Esto lo sabemos. ¿Pero qué significa eso? El cuerpo usa todos los codones a la vez para la codificación. Pero algunos más a menudo, otros menos.Compare humanos y ... E. coli ( Escherichia coli ) en la frecuencia de uso de codones que codifican cisteína.Está codificado por dos codones UGU y UGC. UGUhumana10.6UGC 12.6Escherichia coli (cepa O127: H6)UGU 19.1UGC 0.0Dígitos es la ocurrencia de un triplete por mil. Se puede ver que usamos ambos codones con aproximadamente la misma frecuencia, mientras que E. coli casi no usa codones UGC.Debe recordarse esta característica, especialmente cuando se dedica a la ingeniería genética y desea producir el producto genético de un organismo en otro. Si intenta insertar un gen humano con la aparición frecuente del codón UGC en la E. coli de esta cepa, se sentirá decepcionado. En una célula, los aminoácidos están asociados con ARN de transporte, cada uno de los cuales corresponde a su propio codón. Entonces, el ARNt correspondiente al codón UGC será extremadamente pequeño, lo que ralentizará en gran medida la síntesis.Si está interesado, aquí puede ver las diferencias en la composición de codones de diferentes organismos.La composición del codón puede variar mucho entre organismos de diferentes especies y diferentes cepas. Así tiene Escherichia coliO157: H7 EDL933 es cada vez menos incluso en términos de UGC y UGU. O aquí hay otro ejemplo. Las cepas de bacilo tuberculoso aisladas en diferentes países también tienen una composición de código diferente .
En conclusión, también me gustaría hablar sobre una característica interesante. Un solo aminoácido puede ser codificado por varios codones. Esto lo sabemos. ¿Pero qué significa eso? El cuerpo usa todos los codones a la vez para la codificación. Pero algunos más a menudo, otros menos.Compare humanos y ... E. coli ( Escherichia coli ) en la frecuencia de uso de codones que codifican cisteína.Está codificado por dos codones UGU y UGC. UGUhumana10.6UGC 12.6Escherichia coli (cepa O127: H6)UGU 19.1UGC 0.0Dígitos es la ocurrencia de un triplete por mil. Se puede ver que usamos ambos codones con aproximadamente la misma frecuencia, mientras que E. coli casi no usa codones UGC.Debe recordarse esta característica, especialmente cuando se dedica a la ingeniería genética y desea producir el producto genético de un organismo en otro. Si intenta insertar un gen humano con la aparición frecuente del codón UGC en la E. coli de esta cepa, se sentirá decepcionado. En una célula, los aminoácidos están asociados con ARN de transporte, cada uno de los cuales corresponde a su propio codón. Entonces, el ARNt correspondiente al codón UGC será extremadamente pequeño, lo que ralentizará en gran medida la síntesis.Si está interesado, aquí puede ver las diferencias en la composición de codones de diferentes organismos.La composición del codón puede variar mucho entre organismos de diferentes especies y diferentes cepas. Así tiene Escherichia coliO157: H7 EDL933 es cada vez menos incluso en términos de UGC y UGU. O aquí hay otro ejemplo. Las cepas de bacilo tuberculoso aisladas en diferentes países también tienen una composición de código diferente .Para resumir
Esta vez había mucha historia y relativamente poca biología. Esto no volverá a suceder. Hablamos sobre cómo quedó claro que el ADN es un portador de información, cómo se almacena en el ADN mismo. Hablamos sobre la redundancia del código genético y hacia qué conduce. Las mutaciones y la diferencia en la frecuencia de uso de ciertos codones se vieron ligeramente afectadas.La próxima vez hablaremos sobre la replicación del ADN.PD: también habrá historia, pero mucho menos. Intentaré no hacer esas pausas por escrito nunca más.