
Los científicos realizaron una fiesta en tres en raya usando ADN. Suena divertido, pero esto es solo la punta del iceberg, sacudido por un toque de humor de los propios científicos. De hecho, el estudio de hoy revela nuevos métodos para monitorear la cinética de la interacción de las nanoestructuras de ADN, que juntas pueden realizar tareas complejas. Usar el ADN como las partes principales del sistema no es una tarea fácil, pero esto no detiene a los científicos cuyas mentes están llenas de ideas, sino de corazones de entusiasmo. Y entonces, ¿cómo lograron mover los bloques de ADN de acuerdo con un patrón dado, como lo mostró el estudio, y cuáles son las perspectivas de esta técnica en el futuro? Para obtener respuestas, recurrimos al informe del grupo de investigación. Vamos
Base de estudioEn este estudio, los científicos demostraron una nueva técnica para mover mosaicos de ADN, que se basa en los principios de las articulaciones de soporte y las compensaciones de ramas. Estos principios son similares al desplazamiento estándar de las cadenas de ADN, pero están dirigidos a objetos más grandes, más precisamente a estructuras de ADN.
Esta técnica hizo posible obtener el control sobre la cinética de los mosaicos de ADN y realizar la cadena de reacciones deseada dentro de los sistemas de múltiples estructuras. El juego del tic-tac-toe no se realizó por diversión, sino para demostrar la posibilidad de cambiar el ADN del azulejo en cualquier orden y posición.
Para comprender los mecanismos de los sistemas biológicos, es necesario considerar sus componentes principales, entre los cuales las estructuras moleculares son una de las más importantes. El ADN, como molécula transportadora, se considera uno de los mejores materiales para el estudio y la creación de nanoestructuras autoorganizadas.
Entre las técnicas basadas en el ADN, la técnica de origami de ADN ha ganado cada vez más popularidad en los últimos años, lo que le permite crear estructuras de ADN de cualquier forma y configuración deseadas.
Los componentes de un solo origami de ADN se pueden torcer y torcer a su gusto, pero hasta ahora no ha sido posible lograr una interacción controlada entre varios origami de ADN como componentes de una estructura más grande y, por lo tanto, más compleja. Por el momento, solo era posible conectar / desconectar varios origami de ADN.
En el estudio de hoy, los científicos han demostrado una nueva técnica de interacción de origami de ADN. Hay una estructura en la que se integra el mosaico de origami de ADN, que desplaza a otro mosaico de la matriz general y se conecta a la estructura debido al
dominio de unión * en el borde del mosaico (punto de apoyo / punto de conexión).
Enlace / dominio de enlace * : un dominio de proteínas que forma parte de la cadena de proteínas, pero que puede funcionar por separado.
Como ejemplo, los científicos crearon tres opciones de reconfiguración: competitiva, secuencial y cooperativa. La totalidad de estos "movimientos" (reconfiguración paso a paso) se demostró como un juego de tres en raya, cuando cada jugador tenía 9 origami de ADN únicos que se pueden mover en cualquier orden en un campo de 264x264 nm.
Formación de azulejos de ADN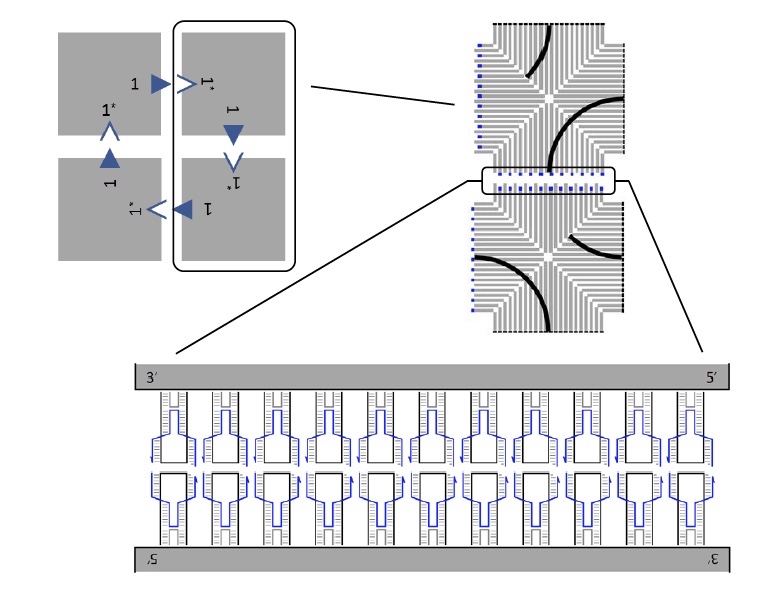 Estructuras de mosaico de ADN.
Estructuras de mosaico de ADN.Anteriormente, antes de realizar este estudio, los científicos desarrollaron una técnica para crear mosaicos de ADN individuales. Cada mosaico en dos bordes tiene 11 grapas, cada una de las cuales participa en el apilamiento.
La imagen de arriba muestra una matriz de 4 mosaicos (arriba a la izquierda), cada uno de los cuales está conectado al adyacente a través de paréntesis.
Los investigadores consideraron varias opciones posibles para crear matrices más grandes con una gran cantidad de mosaicos. Para esto, en teoría, era necesario que la conexión entre las fichas fuera lo suficientemente débil. Esto le permitiría romper y reconstruir la matriz. Sin embargo, las observaciones mostraron que es posible crear matrices reconfigurables de múltiples mosaicos incluso a temperaturas cuando la conexión entre los mosaicos debe ser permanente.
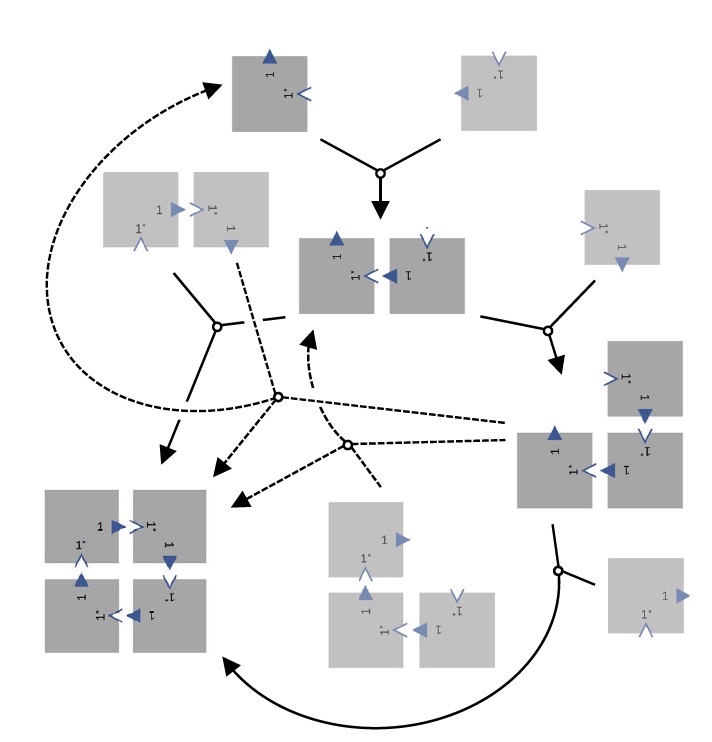 Reconfiguración de azulejos.
Reconfiguración de azulejos.Este fenómeno tiene dos posibles explicaciones. Primero, la formación de enlaces ocurre a una temperatura suficientemente alta, mientras cae dentro del rango de temperatura de la reversibilidad del proceso. El segundo: el dímero y el trímero sufren una reacción de desplazamiento, por lo tanto, se forma una matriz de 2x2 cuando se libera el monómero. También se pueden hacer 2 copias de recortadores, formando una matriz de 2x2 liberando un dímero.
Si durante las reacciones de desplazamiento no hay desconexión espontánea de las baldosas, entonces no habrá trampas cinéticas, por lo que las baldosas se recogerán en una configuración de 2x2.
Era necesario verificar si un origami de ADN desplazaría a otro en la estructura general, sin unirse espontáneamente dentro de la estructura misma. En otras palabras, si el origami de ADN mostrará iniciativa o no. Para esto, se realizaron dos experimentos.
 Experiencia práctica: 2 mosaicos de ADN (cuadrados) + 1 triángulo de ADN, que deberían formar una estructura común.
Experiencia práctica: 2 mosaicos de ADN (cuadrados) + 1 triángulo de ADN, que deberían formar una estructura común.En el primer experimento, participaron 2 mosaicos de ADN y 1 triángulo de ADN; el experimento en sí se realizó a temperatura constante. Estructuralmente, un triángulo podría tener el mismo dominio de conexión que los cuadrados, o podría tener un dominio de conexión adicional que complemente el cuadrado adyacente.
En la primera versión del experimento, 2 cuadrados permanecen conectados entre sí, mientras que el triángulo se conecta a uno de los cuadrados. En la segunda versión, un triángulo reemplazó uno de los cuadrados, desplazándolo, por así decirlo. Esto en la práctica ha demostrado que es posible modificar origami de ADN complejo. Pero ahora debemos asegurarnos de que este proceso sea controlado por los científicos y que no ocurra por sí solo debido a las fuerzas naturales.
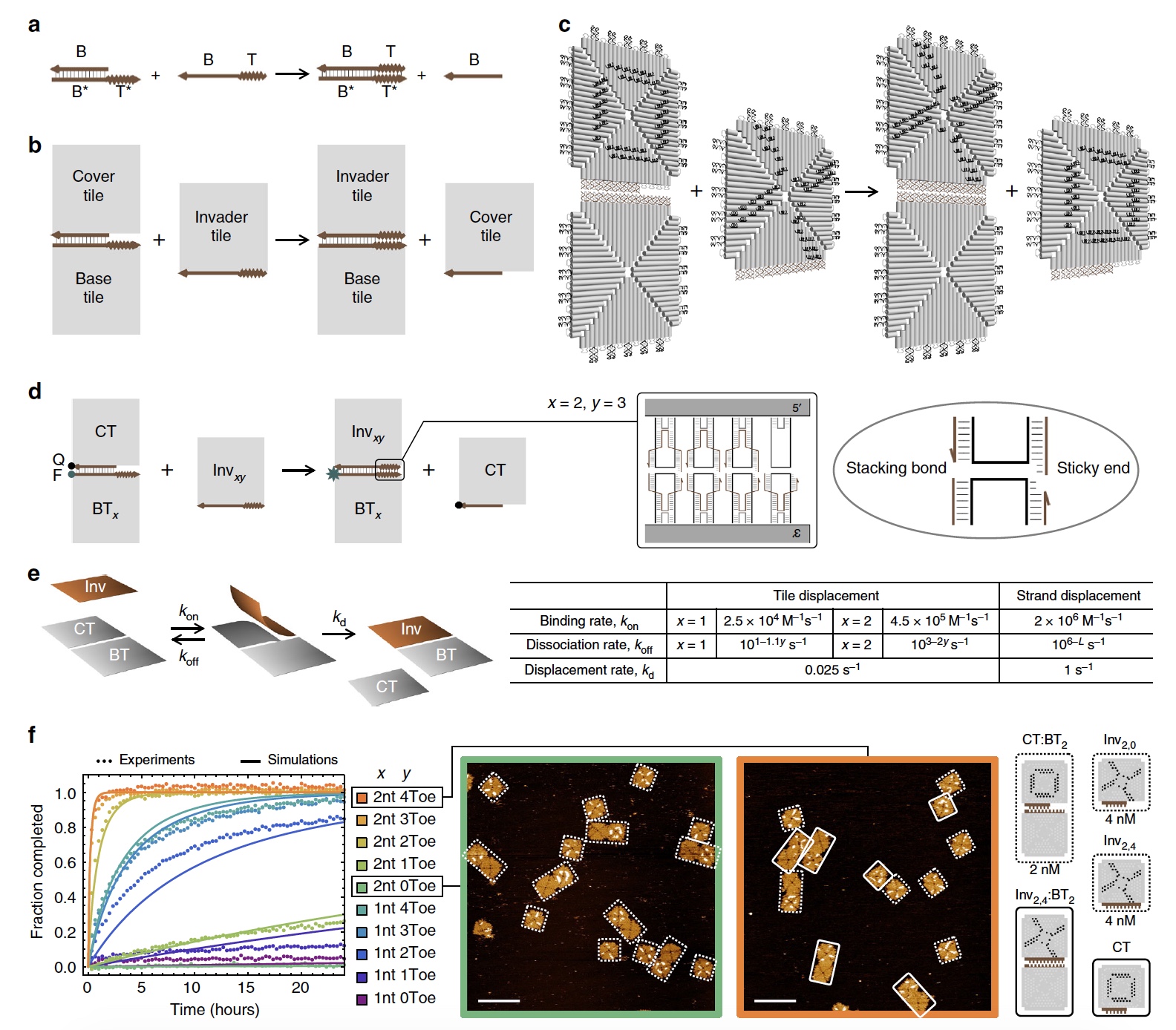 Imagen No. 1
Imagen No. 1Si hablamos de reacciones de desplazamiento en cadenas de ADN, entonces aquí tenemos una cadena con un dominio de unión que se conecta a un dominio complementario "libre". Entonces obtenemos una estructura bicatenaria en la cual el proceso de migración de ramas ocurre cuando las cadenas se mueven a través del punto de conexión (
1a ). Un proceso similar ocurre en las reacciones de desplazamiento de ADN de los azulejos (
1b y
1c ).
De la imagen
1c también podemos ver que cada mosaico de ADN consta de 4 triángulos isósceles, conectados por llaves, que ya conocemos.
En cuanto al juego del tic-tac-toe: para una visualización más conveniente, el proceso de desplazamiento y reconfiguración de los participantes se marcó con una cruz (X) y un dedo del pie (O) mediante corchetes en dos colores diferentes. Eche un vistazo más de cerca a la imagen No. 1, X (negro) y O (blanco) son visibles en los mosaicos.
Cinética de las reacciones de desplazamiento del ADN del azulejoEn palabras simples, obtener el control sobre la cinética de un proceso necesita hacer que algunas reacciones sean más rápidas y otras más lentas. De esta forma, se puede unir, desconectar y reconfigurar los mosaicos. Pero debe determinar el rango de cinética que permite que esto se haga. Para hacer esto, los científicos crearon puntos de conexión (entre mosaicos) con diferentes fuerzas, por así decirlo, de acoplamiento.
El número de grapas en el conector en el dominio de migración de rama fue el mismo para todos los mosaicos. Pero aquí el dominio de dominio de los puntos de referencia (empate) varió de 0 a 4 (
1d ).
El experimento involucró el mosaico principal (los 4 puntos de comunicación son "activos") y los mosaicos accesorios (el número de puntos de conexión varió de 0 a 4). Al final del dominio de migración de rama, se modificaron 2 pares de grapas con un fluoróforo y un desactivador. Si el azulejo accesorio permanece en contacto con el azulejo principal, entonces el fluoróforo se extinguirá, lo que conducirá a una señal fluorescente baja. Pero si el mosaico accesorio (con el amortiguador) está desconectado, la señal fluorescente se amplificará. Por lo tanto, será posible determinar cómo procede el proceso.
Un día después, fue posible sacar las primeras conclusiones de las observaciones. Los científicos señalan que las trayectorias fluorescentes de los azulejos accesorios con 0 soportes de conexión permanecieron prácticamente sin cambios. También se observó que las baldosas con la misma longitud de la costilla de conexión, pero con una gran cantidad de puntos de conexión, exhiben una cinética mucho más rápida. Es curioso que los puntos de conexión con las grapas con 1 nucleótido y 2 nucleótidos muestren tasas muy diferentes de saturación (saturación).
La imagen
1e muestra el modelo matemático utilizado para la estimación numérica de la cinética. Los propios científicos dicen que el modelo es muy simple y que a menudo se usa para evaluar la cinética en las reacciones de desplazamiento de la cadena de ADN y, por lo tanto, también es adecuado para los mosaicos de ADN.
Un análisis comparativo de las simulaciones (
1f ) (reacciones simuladas) y los experimentos permitieron determinar los parámetros que afectan el proceso de recombinación de los mosaicos de ADN.
En primer lugar, los científicos señalan que la velocidad de unión de los mosaicos es de 10 a 100 veces (dependiendo de la longitud del borde del mosaico de unión) menor que la velocidad de unión de las cadenas de ADN. Una cosa común entre los procesos de desplazamiento de azulejos e hilos es que con un aumento en el número de nucleótidos en los puntos de conexión, la tasa de desintegración disminuye exponencialmente. Al mismo tiempo, esta velocidad es 40 veces menor para los mosaicos que para las cadenas de ADN. El desplazamiento total máximo para las tejas de ADN (velocidad de reacción) fue de 4.5x10
5 M
-1 * s
-1 . Si la concentración es baja (<50 nM), entonces la tasa de unión bimolecular limitará la tasa de desplazamiento de las losetas. Si la concentración es más de 50 nanomoles, entonces la tasa de desplazamiento monomolecular limitará la tasa de desplazamiento de las baldosas.
Además, los científicos realizaron varios experimentos que describen 3 tipos principales de reconfiguración: competitiva, secuencial y cooperativa. Y ahora veamos los resultados de los experimentos para cada uno de ellos con más detalle.
Reconfiguración competitivaPara lograr una reconfiguración competitiva, los científicos propusieron usar una función sigmoidea en respuesta a la concentración de la señal, obteniendo así un sistema de reconfiguración basado en la información. Esta función es un elemento importante de los cálculos de lógica digital de los procesos de desplazamiento de cadena de ADN, por lo tanto, también se puede aplicar a los mosaicos de ADN.
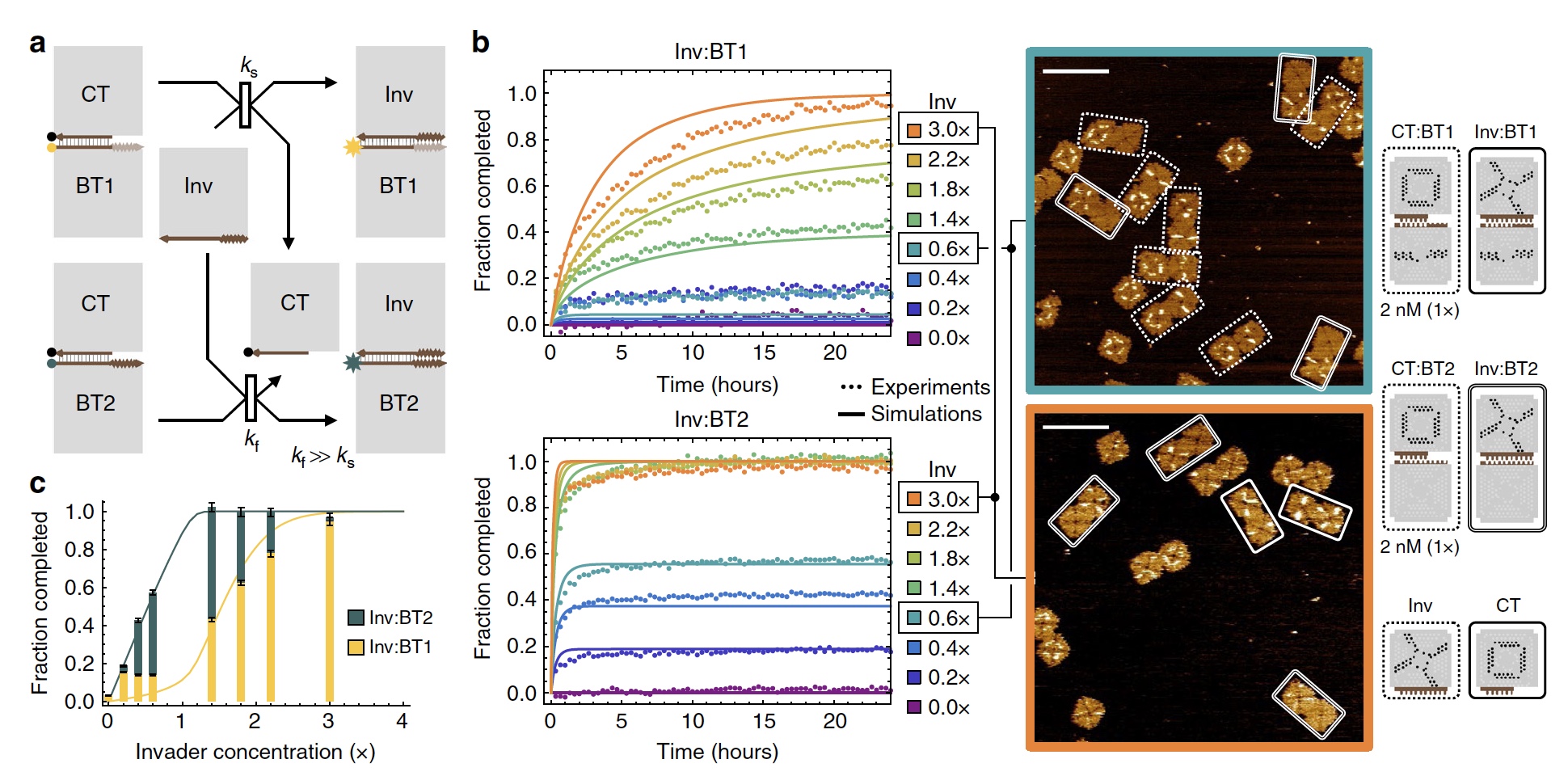 Imagen No. 2
Imagen No. 2Con el fin de demostrar el "trabajo" de la función sigmoidea, los científicos crearon 2 reacciones de desplazamiento de fichas que se activan por la misma ficha subordinada. La diferencia en su velocidad (
2a ).
En los azulejos accesorios, el punto de conexión son cuatro grapas de 2 nucleótidos. Se espera que se conecte con un punto de enlace adecuado y con cuatro grapas de 1 nucleótido. La velocidad de reacción de los azulejos accesorios difiere en 18 veces. Cuando la concentración del azulejo accesorio fue inferior a 2 nM, se desencadenó una reacción más rápida (
2b ). Si la concentración fue superior a 2 nM, se inició una reacción más lenta.
Después de 24 horas, se descubrió que el resultado de una reacción más rápida aumentaba linealmente, y el resultado de una reacción más lenta mostraba una función sigmoidea (
2c ).
Reconfiguración secuencialSeguir la reconfiguración competitiva fue consistente. Este tipo es mucho más complicado, pero brinda más control sobre el proceso, a menos que, por supuesto, todo funcione.
Entonces, los científicos crearon una matriz de 2x2 en la que la primera subplaca desplaza a otra baldosa, liberando el punto de conexión utilizado anteriormente. A continuación, la segunda estructura subordinada de 2 fichas desplaza 2 fichas de la matriz. Una especie de desplazamiento en cascada (
3a ).
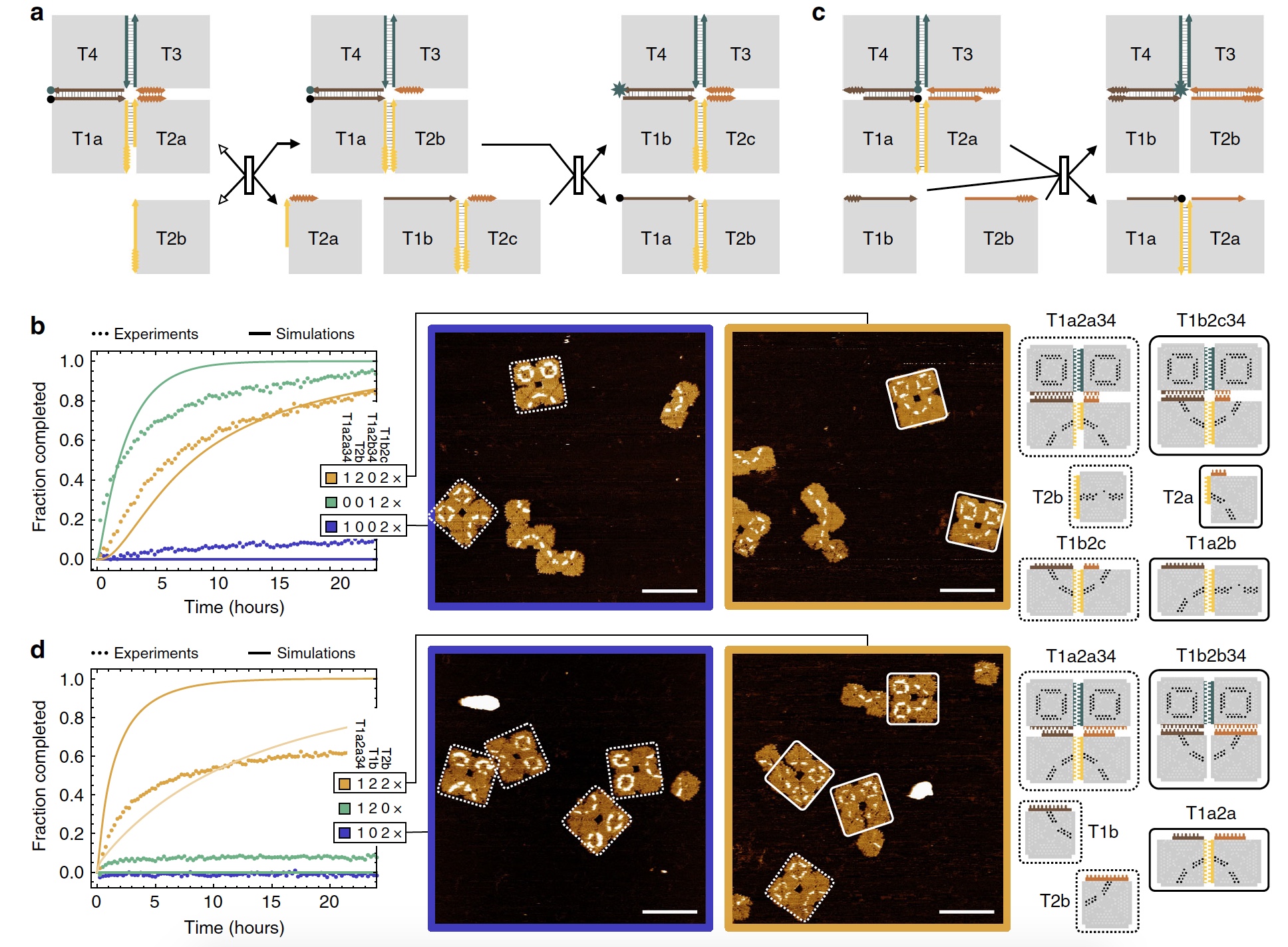 Imagen No. 3
Imagen No. 3Los científicos han notado que el desplazamiento en las esquinas de las baldosas es lento, aproximadamente 100 veces más lento que en el resto del borde de las baldosas. Por lo tanto, tales estados intermedios de desplazamiento deben tenerse en cuenta para procedimientos de reconfiguración más complejos.
En la imagen
3b podemos ver resultados bastante exitosos del experimento con la reconfiguración secuencial (curva naranja).
Y ahora hay un poco más de creatividad por parte de los investigadores. Presta atención a las imágenes de arriba (
3b ): matrices que reaccionaron a la primera ficha subordinada, pero no a la segunda, al final se ven como una "cara triste". Pero las matrices, en las cuales la conexión con ambas fichas subordinadas fue exitosa, parecen una "cara sonriente". Y usted dice que los científicos no tienen sentido del humor.
Según los científicos, cuanto más compleja es la estructura de los participantes de la reacción, más complicado es el proceso de reconfiguración en sí, pero más interesante es su resultado. El uso de dos tipos de participantes (matriz de 2x2 y estructura de dos mosaicos) mostró los siguientes resultados sobre la eficiencia del desplazamiento después de 48 horas de observación: 83.3 ± 9.8% y 90.5 ± 6.1%. En otras palabras, las reacciones de desplazamiento son excelentes. Hay algunos errores, pero son insignificantes.
Los experimentos con la reconfiguración secuencial permitieron no solo comprender los principios básicos de la secuencia de puntos de comunicación y la "cascada" de la reacción, sino que también demostraron nuevas características. El sistema es capaz de responder a más de 1 señal que indica qué instrucciones requieren ejecución y qué hay disponible para esto. Esta señal se presenta en el experimento en forma de dos tipos de estructuras subordinadas. Incluso si la primera estructura subordinada entra en contacto con la base antes que la segunda, la estructura aún muestra la reconfiguración esperada.
Estos procesos en su mayor parte proceden por sí mismos. Los científicos solo los controlan ligeramente, pero no intervienen en su totalidad. El siguiente paso en los experimentos es aumentar el control sobre el proceso de reconfiguración y estudiar la posibilidad de programar este proceso.
Reconfiguración cooperativaEn este experimento, hubo 2 mosaicos accesorios que se conectaron a los dos bordes de la matriz de 2x2 (
3s ). Si solo hay un mosaico presente, entonces debería moverse al centro de la matriz. Además, la ausencia de un segundo mosaico le permite revertir el proceso, desconectando repetidamente el mosaico accesorio de la matriz. En presencia de ambas teselas subordinadas, surgen dos procesos de migración de ramas inmediatamente, que convergen en el centro de la matriz, y como resultado obtenemos un desplazamiento cooperativo.
La experiencia con fluorescencia demostró que si solo hay un mosaico subordinado, la señal de fluorescencia es muy débil, pero se amplifica si ambos mosaicos están presentes (
3d : curva azul y verde - señal débil, amarillo - señal fuerte).
Como en el experimento anterior, las matrices que interactúan solo con una ficha subordinada se marcaron como "cara triste", y las matrices y ambas fichas fueron "cara sonriente". Además, este último se formó en 68.0 ± 7.7% de los casos.
Tic Tac ToeY finalmente, el experimento "tic-tac-toe". En este experimento, se usó una matriz de 3x3, que permite 9 reacciones únicas de desplazamiento de la loseta en cualquier orden (
4a ).
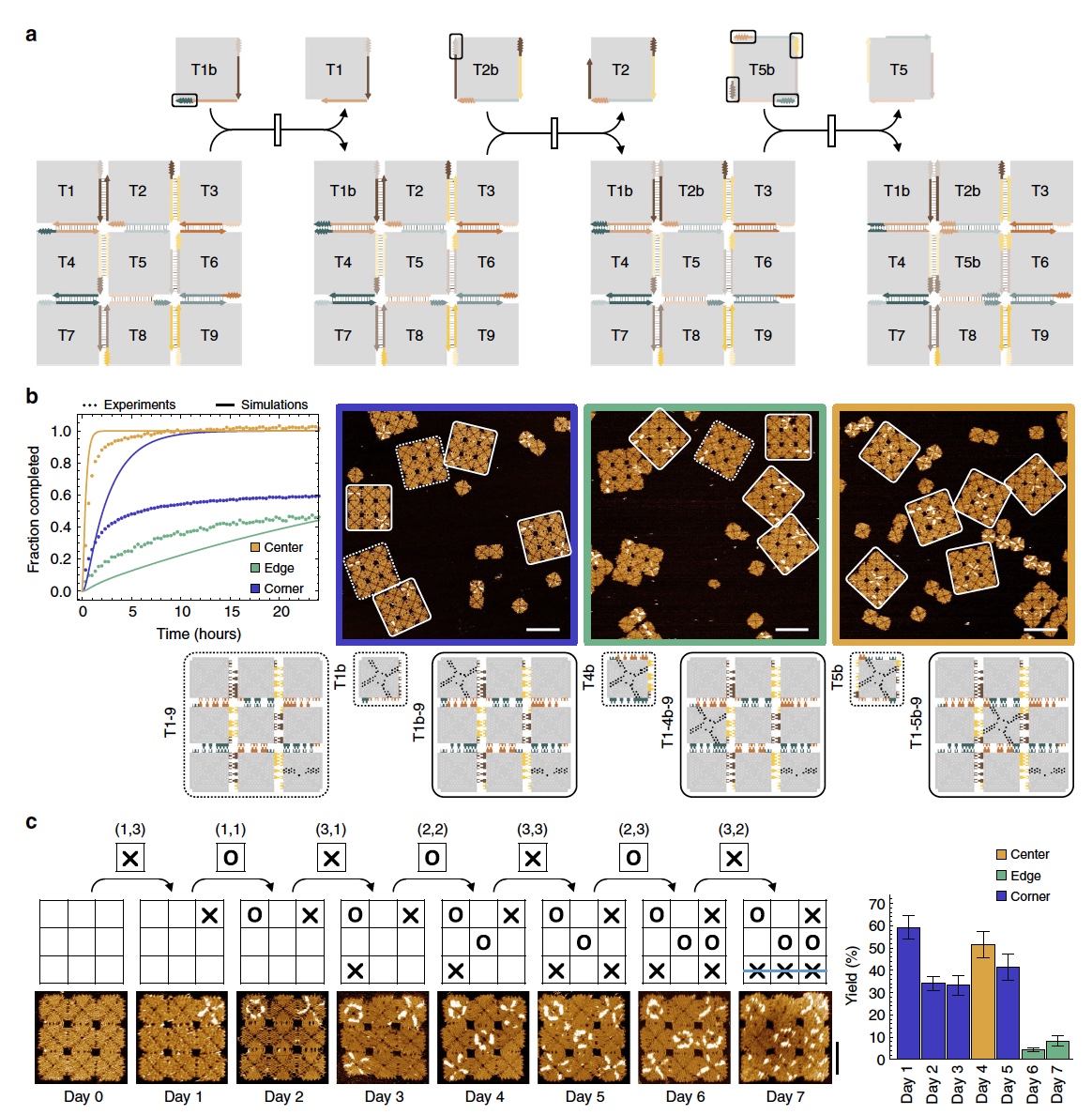 Imagen No. 4
Imagen No. 4Este experimento es una combinación de varios tipos de reacciones a la vez: el desplazamiento de la loseta de esquina, el desplazamiento de la loseta de borde y el desplazamiento de la central. A lo largo del perímetro de la matriz entre cada mosaico adyacente había un punto de conexión, un total de 8 de esos puntos. 4 de ellos participaron en el desplazamiento de la loseta de esquina, cuando la loseta accesoria con el punto de conexión correspondiente está en contacto con la loseta de borde cerca de la esquina. En este momento, la rama migra dentro del borde de la loseta y a través de un ángulo de 90 °, y la loseta de esquina previamente conectada a la matriz se libera e integra en la matriz misma (dentro de la matriz cuando estaba previamente en el borde). Los otros 4 puntos de amarre se usaron para activar el desplazamiento de las baldosas de borde.
Lo más difícil en este experimento fue iniciar un desplazamiento de la baldosa central, debido al número limitado de puntos de comunicación disponibles. Por lo tanto, se agregaron 4 puntos adicionales entre la loseta central y todas las losetas vecinas (
4a ).
Las observaciones mostraron excelentes resultados. Todos los sub-mosaicos fueron incrustados en la matriz, que fue reconstruida como los científicos habían planeado. El porcentaje de conversión dentro de la matriz fue el siguiente: 78.4 ± 6.0% para el desplazamiento de las losetas de esquina, 52.8 ± 6.0% para el desplazamiento de las losetas de borde y 100% de las centrales.
Y ahora pasamos directamente al juego en sí, que combina todo tipo de reacciones en forma de cascada. El tablero de juego era una matriz de 3x3, todas las fichas estaban "limpias", es decir, no estaban marcadas de ninguna manera.
Cada uno de los jugadores, de los cuales había 2 naturalmente, recibió a su disposición 9 fichas subordinadas marcadas con "X" y "O". Hacer un movimiento en este juego es agregar este mosaico a la cámara de prueba con un intervalo de 24 horas (un lote largo de tres en raya, ¿no es así?).
Video de demostración de jugar al tic-tac-toe con fichas de ADN.Las observaciones (
4b y
4c ) mostraron que el campo de juego reaccionó a todos los movimientos, es decir, la reconfiguración se realizó de acuerdo con las instrucciones de los jugadores (agregando fichas marcadas).
Todo el problema era que al final del juego en el campo había muchas fichas accesorias, respectivamente, aumentaba la probabilidad de su desplazamiento espontáneo. Por lo tanto, la precisión de la reconfiguración también se redujo. Al final del juego, este indicador era de solo 8.3 ± 2.3%. Sin embargo, el resultado aún se logró, como podemos ver en las imágenes de arriba.
Para un conocimiento más detallado de los matices de la investigación y los experimentos, le recomiendo que consulte el
informe de los científicos y los
materiales adicionales .
EpílogoEl juego de fichas de ADN de tres en raya no se llevó a cabo por diversión, sino como un método para demostrar un estudio muy serio, que revela la capacidad de manipular los procesos de desplazamiento de elementos en nanoestructuras construidas a partir de ADN.
Por supuesto, queda mucho por pulir: el número de mosaicos accesorios que afectan la tasa final de reconfiguración, la tasa de reacciones, la precisión del desplazamiento, la expansión de las posibles opciones de desplazamiento y, por supuesto, el aumento de las dimensiones de la estructura de ADN reconfigurable.
, . , , , — , , .
, google «-», . :) ( , )
Gracias por quedarte con nosotros. ¿Te gustan nuestros artículos? ¿Quieres ver más materiales interesantes?
Apóyenos haciendo un pedido o recomendándolo a sus amigos, un
descuento del 30% para los usuarios de Habr en un análogo único de servidores de nivel de entrada que inventamos para usted: toda la verdad sobre VPS (KVM) E5-2650 v4 (6 núcleos) 10GB DDR4 240GB SSD 1Gbps de $ 20 o cómo dividir el servidor? (las opciones están disponibles con RAID1 y RAID10, hasta 24 núcleos y hasta 40GB DDR4).
VPS (KVM) E5-2650 v4 (6 núcleos) 10GB DDR4 240GB SSD 1Gbps hasta la primavera sin cargo al pagar durante medio año, puede ordenar
aquí .
Dell R730xd 2 veces más barato?Solo tenemos 2 x Intel Dodeca-Core Xeon E5-2650v4 128GB DDR4 6x480GB SSD 1Gbps 100 TV desde $ 249 en los Países Bajos y los EE. UU. Lea sobre
Cómo construir un edificio de infraestructura. clase utilizando servidores Dell R730xd E5-2650 v4 que cuestan 9,000 euros por un centavo?