¿A quién no le gustaba coleccionar constructores en la infancia? Todavía recuerdo esta caja roja con un montón de piezas metálicas, herramientas y un mar de posibles resultados, si solo hubiera fantasía, tiempo y deseo. LEGO tampoco debe ser olvidado, aunque aquí todo fue un poco más simple y mucho más colorido. Pero lo que es más complicado de ambos son los diseñadores de nanoestructuras basadas en origami de ADN. Hasta ahora, todos los "detalles" de tales estructuras se modelaron manualmente, lo que tomó mucho tiempo y esfuerzo. Imagine que necesita crear todas las piezas LEGO usted mismo antes de ensamblarlas en un robot gigante con láser, motores a reacción y una ametralladora en su hombro. Pero algo que los recuerdos de la infancia nos llevaron a la estepa equivocada.
Chistes a un lado. Hoy conoceremos el algoritmo que le permite crear automáticamente origami de ADN de una forma bastante diversa. Anteriormente, el cambio de la forma de las hebras de ADN a lo necesario se realizaba manualmente, lo que limitaba enormemente las posibilidades de dicho procedimiento. El mismo algoritmo le permite crear partes de ADN de diseño automáticamente, lo que le permite usarlas para la formación de nanoestructuras bidimensionales y tridimensionales. Además, este algoritmo está disponible para todos. Un informe del equipo de investigación nos ayudará a descubrir qué funciona y cómo. Vamos
Base de estudioEl uso del ADN como material de construcción en nanoestructuras de una amplia gama de aplicaciones en los últimos años se ha convertido en el objetivo de muchos estudios y experimentos. Muchos de ellos han logrado excelentes resultados, pero como sabemos, no hay límite para la perfección. Como ejemplo, los investigadores citan estructuras de ADN monodispersas en una escala de megadalt con un alto coeficiente de coagulación, que se realizó utilizando marcos de ADNss largos (ADN monocatenario): ADN monocatenario / monocatenario. Sin embargo, en este caso, la longitud de las cadenas de ADN es la principal limitación en el tamaño de un solo ADN de origami. Esta limitación se puede eludir mediante la formación de una estructura que consta de varios origami a la vez.
También ya sabemos (
Tic-tac-toe: una demostración del proceso controlado de reconfiguración de estructuras de ADN ) que algunas estructuras de ADN se pueden crear utilizando mosaicos de ADN, lo que le permite formar una geometría compleja de esta estructura.
Pero de nuevo, todo se reduce al hecho de que el proceso se realiza manualmente, y esto es largo y triste. ¿Qué les gusta a los científicos? Así es, la automatización, o al menos la automatización parcial. Herramientas similares ya están disponibles (un ejemplo de científicos - caDNAno o Tiamat), sin embargo, estos programas tienen sus inconvenientes. En primer lugar, la creación manual del marco. En segundo lugar, las limitaciones de los algoritmos anteriores en el tema de la sistematización del diseño de la geometría de la estructura.
En pocas palabras, en este documento, los investigadores se propusieron el objetivo de crear un algoritmo que haga casi todo por sí mismo. Por lo tanto, cualquier persona puede establecer solo los parámetros de la capa externa de la estructura deseada, y el programa llenará la parte interna de forma independiente con todos los hilos de ADN, grapas, secuencias y más necesarios. También puede llevar a cabo el proceso en el orden inverso: establezca todos los "interiores", por así decirlo, la cuadrícula de estructura y el algoritmo creará el marco correcto y más óptimo a su alrededor.
Este programa de código abierto (PERDIX) está disponible para absolutamente todos, por lo que gracias a los investigadores. Por lo tanto, cualquiera no solo puede "jugar" con el programa en sí, sino también, posiblemente, hacer algunos cambios en su código. Dónde descargarlo y cómo iniciarlo lo consideraremos al final del artículo.
Y ahora veamos ejemplos de la aplicación de este programa que el equipo de investigación nos proporcionó.
Resultados de validación PERDIX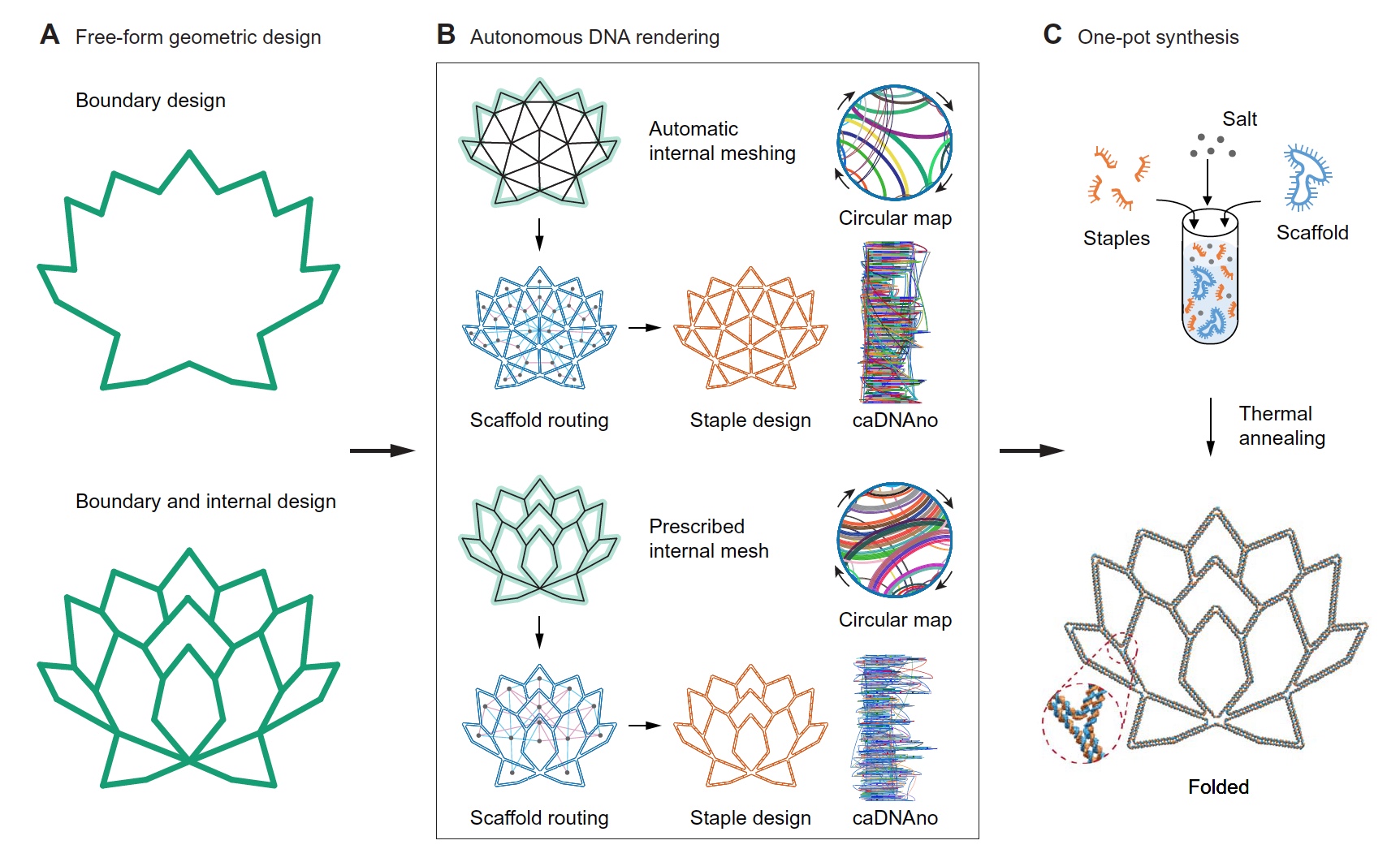 Imagen No. 1
Imagen No. 1Como datos de entrada, se utiliza la imagen del borde de la estructura deseada (
1A , arriba) o una imagen con detalles geométricos más precisos (
1A , abajo). Además, el programa en sí mismo llena el espacio interno.
Cuando solo se especifica el "contorno", la geometría interna de la cuadrícula de la estructura está formada por triángulos que utilizan el programa de código abierto DistMesh, que requiere la introducción de solo la densidad de la cuadrícula como parámetro necesario.
Cuando se especifican la forma y las líneas geométricas (
1A , abajo), se usa Shapely, un paquete de Python para manipular y analizar objetos geométricos planos. Shapely genera una malla poligonal, en la que sus líneas y sus puntos de intersección forman la geometría necesaria del marco.
Además, después de determinar la forma deseada, fue necesario establecer la longitud nominal de la costilla, que debería ser ≥ 38 pb (bases emparejadas) o 12,58 nm. Por lo tanto, al menos dos cruces de emparejamiento por borde definitivamente estarán presentes en la estructura requerida.
Luego, todas las líneas de destino de la estructura futura se transforman en bordes DX, en los que todos los vértices son puntos de conexión de cuadrícula múltiple (
1B ). Siguiendo los principios de la teoría de grafos, la estructura esquelética óptima se forma automáticamente, después de lo cual se completa el proceso de asignación de una secuencia de grapas de ADN adicionales. El proceso de edición manual de los datos de salida (resultados) se lleva a cabo mediante el archivo caDNAno generado.
Una característica muy importante de su sistema, los científicos llaman al hecho de que las costillas no tienen que ser iguales a un número entero de hélices dobles de ADN B (10.5 pb). Y esto le permite obtener más libertad en el diseño de la geometría de la estructura.
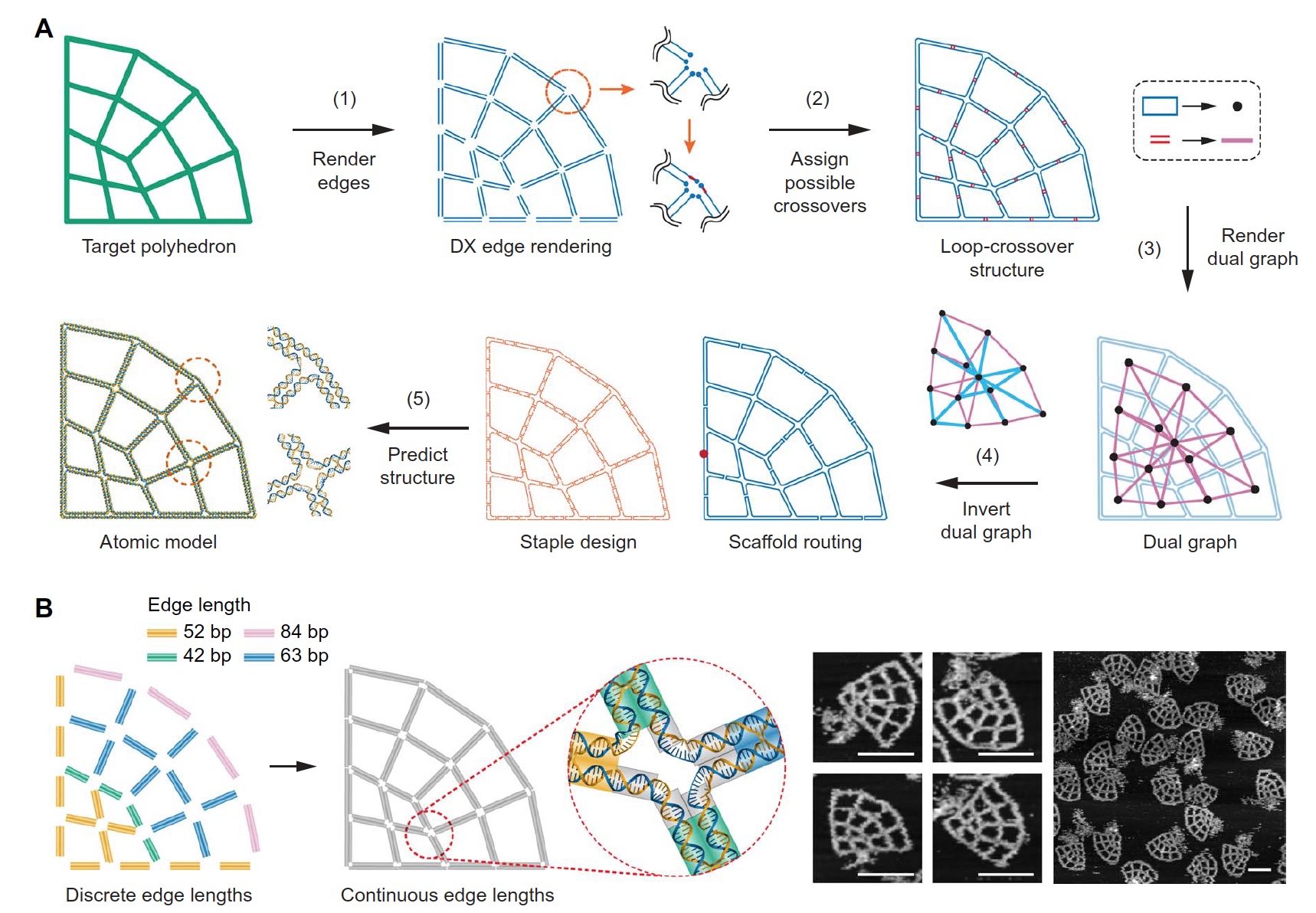 Imagen No. 2
Imagen No. 2Para realizar de forma rápida y precisa la parte automática del proceso de formación de la geometría de la estructura, primero se deben convertir todos los bordes en motivos DX renderizando cada borde de la geometría objetivo utilizando dos líneas de marco antiparalelas (
2A ). Luego se combinan en un solo vértice, lo que hace que cada borde forme parte de un bucle. Y esto a su vez conduce a la formación de un solo bucle externo grande, que consiste en pequeños bucles. El siguiente paso es encontrar todas las opciones posibles de cruce entre bucles adyacentes, es decir, la formación de una estructura de intersección de bucles.
El tercer paso en este proceso es representar todas las estructuras resultantes como un conjunto de nodos, y todos los cruces como bordes de la estructura futura.
A continuación, se calcula el árbol de expansión del gráfico doble, después de lo cual se convierte en un marco de ssDNA. Este proceso es completamente automático. Y finalmente, se genera un modelo estructural tridimensional a nivel atómico.
La Figura
2B muestra dos opciones para la formación de la geometría requerida de la estructura: con longitud de borde discreta o con longitud de borde continua. La segunda opción le permite crear una estructura con una longitud de borde arbitraria, que a su vez permite obtener la mayor coagulación. Estas variaciones de la estructuración de la geometría se verificaron utilizando un microscopio de fuerza atómica de barrido (imágenes a la derecha en la imagen
2B ).
 Imagen No. 3
Imagen No. 3Los investigadores también se propusieron el objetivo de examinar con más detalle aspectos de la geometría del objetivo como la rigidez mecánica, la precisión de la forma, el tamaño de la estructura, el tamaño de la malla interna y los tipos de sus elementos. También fue necesario comprender la importancia de las ramas N, es decir, el número de conexiones entre los bordes y, como resultado, la complejidad general de la estructura. El análisis de los datos de AFM mostró que las estructuras bidimensionales con una gran cantidad de ramas y una longitud de borde más larga (debido a nucleótidos no apareados) se obtienen bastante bien formadas, y la heterogeneidad de partículas en su estructura es mínima.
La cuadrícula interna de la estructura tenía tres opciones: triángulos con la misma dirección, cuadrados y triángulos con diferentes direcciones (
3C ). Fue el modelo con triángulos el que mostró la mayor precisión en la formación de la geometría necesaria, en contraste con el modelo con cuadrados. La presencia de direcciones mixtas en el modelo triangular, por así decirlo, permitió obtener ramas N más simétricas y una forma más precisa (menos distorsionada).
Los científicos señalan que el análisis de la malla interna de la estructura simulada es extremadamente importante para comprender la rigidez mecánica de esta estructura. Y es precisamente el modelo descrito anteriormente que es ideal para crear estructuras suficientemente fuertes, precisas (en términos de cumplir con los parámetros especificados) y flexibles.
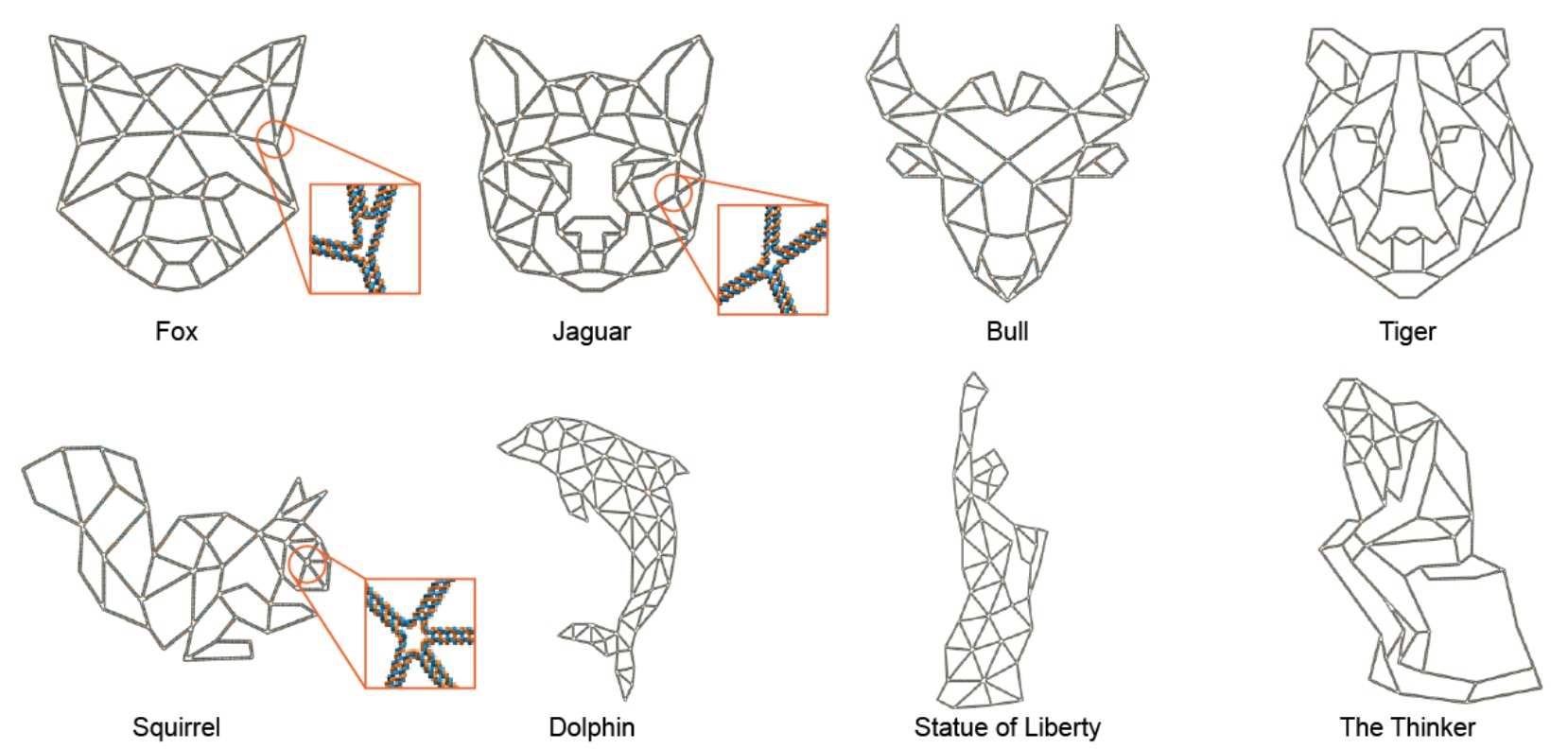
 Imagen No. 4
Imagen No. 4Por supuesto, los científicos no están exentos de belleza, por así decirlo. Por lo tanto, decidieron demostrar las posibilidades de su desarrollo mediante el diseño de estructuras de 15 formas diferentes, más precisamente con diferentes rejillas internas y marco. Podemos ver las opciones en la imagen de arriba: aquí están los cuadrados "habituales", y un cuarto de círculo, e incluso un loto.
Software de código abiertoComo mencioné anteriormente, el trabajo de los investigadores está abierto a todos en el enlace (), donde pueden encontrar los elementos necesarios del programa (MATLAB, Python 2.7 y Shapely 1.6.4).
En cuanto al algoritmo para la formación de la geometría de las estructuras de ADN-origami, las instrucciones para descargarlo y ejecutarlo se presentan a continuación en formato de video. Ocultaré todos los videos debajo del spoiler para no estirar el artículo.
Video # 1: lanzamiento de PERDIXEl enlace para descargar los archivos necesarios que estaba en el video no funciona ahora. Aquí hay una alternativa:
enlace de descarga .
Video No. 2: diseño del marco (perímetro) de una estructura en PERDIX
Video # 3: diseño del marco y la estructura interna en PERDIX
Video No. 4: modelos atómicos (diferentes cuadrículas)
Video No. 5: modelos atómicos (ramas N)
Video No. 6: modelos atómicos (modelo L)
Video No. 7: modelos atómicos ("rama" curva)
Para conocer más detalladamente este estudio y sus resultados, le recomiendo que consulte el
informe del grupo de investigación y los
materiales adicionales .
EpílogoEste estudio tuvo como objetivo simplificar un proceso que consume bastante tiempo y tiempo. Y funcionó. El algoritmo PERDIX le permite crear estructuras con geometrías muy diferentes, mientras establece los parámetros mínimos necesarios. El ADN ha sido durante mucho tiempo un objeto de estudio para los científicos, no solo como parte de toda la vida y el portador de información, sino también como una posible opción para la fundación de tecnologías futuras. Tales trabajos permiten comprender con más detalle (y claramente) cuán amplias son las posibilidades de las nanoestructuras creadas a partir del origami de ADN.
El segundo momento agradable en este estudio, al menos para mí, es que el algoritmo está disponible para todos. Cualquiera puede usarlo, cualquiera puede mejorarlo. Al dar acceso completo tanto a su informe como al software en sí, los científicos no solo contribuyen a la popularización de su campo científico, sino también a la ciencia como tal.
Conocí artículos en los que los científicos eran bastante críticos sobre el sistema de acceso pagado a los informes, argumentando que esto pone una barrera adicional entre el conocimiento y la gente común. La situación en este asunto es muy ambigua y controvertida, porque todos deberían ganar dinero (publicaciones científicas, grupos de investigación y sitios científicos), pero el alto costo de acceso a algunos estudios los hace inaccesibles para los amantes de la ciencia ordinaria, los estudiantes e incluso algunos profesores y Figuras científicas. Como dije, la pregunta es ambigua, por lo tanto, no la tocaremos mucho. En cualquier caso, me alegra que nuestros héroes de hoy nos hayan dado acceso total a su trabajo, que merece no solo atención, sino también una sincera admiración.
Gracias por su atención, sigan curiosos y tengan una buena semana laboral, muchachos.
Gracias por quedarte con nosotros. ¿Te gustan nuestros artículos? ¿Quieres ver más materiales interesantes?
Apóyenos haciendo un pedido o recomendándolo a sus amigos, un
descuento del 30% para los usuarios de Habr en un análogo único de servidores de nivel de entrada que inventamos para usted: toda la verdad sobre VPS (KVM) E5-2650 v4 (6 núcleos) 10GB DDR4 240GB SSD 1Gbps de $ 20 o cómo dividir el servidor? (las opciones están disponibles con RAID1 y RAID10, hasta 24 núcleos y hasta 40GB DDR4).
VPS (KVM) E5-2650 v4 (6 núcleos) 10GB DDR4 240GB SSD 1Gbps hasta la primavera sin cargo al pagar durante medio año, puede ordenar
aquí .
Dell R730xd 2 veces más barato? ¡Solo tenemos
2 x Intel Dodeca-Core Xeon E5-2650v4 128GB DDR4 6x480GB SSD 1Gbps 100 TV desde $ 249 en los Países Bajos y los Estados Unidos! Lea sobre
Cómo construir un edificio de infraestructura. clase utilizando servidores Dell R730xd E5-2650 v4 que cuestan 9,000 euros por un centavo?