
Lors de la rédaction d'un article sur le développement d'un détecteur d'anomalies, j'ai implémenté l'un des algorithmes appelé Incremental Growing Neural Gas.
Dans Littérature soviétique le segment russe de l'Internet, ce sujet est assez mal couvert, et il n'y avait qu'un seul article , et même alors avec l'application de cet algorithme.
Alors, qu'est-ce qu'un algorithme incrémentiel de croissance des gaz neuronaux?
Présentation
IGNG, comme GNG , est un algorithme de clustering adaptatif.
L'algorithme lui-même est décrit dans un article de Prudent et Ennadji pour 2005 .
Comme GNG, il existe de nombreux vecteurs de données ou générer une fonction , qui fournit des vecteurs à partir de données distribuées aléatoirement (paramètre - heure ou numéro d'échantillon dans l'échantillon).
L'algorithme n'impose pas de restrictions supplémentaires sur ces données.
Mais l'intérieur est très différent du GNG.
Cet algorithme est également intéressant en ce qu'il est légèrement plus précis que la neurogenèse des modèles GNG.
Description de l'algorithme
L'algorithme décompose un grand nombre de données en grappes.
Comparé au GNG, son avantage est un taux de convergence plus élevé.
Idées sur lesquelles l'algorithme est basé:
- Théorie de la résonance adaptative : Premièrement, le neurone le plus proche est recherché, et si la différence ne dépasse pas le seuil (le «paramètre de vigilance»), les poids sont ajustés ou, sinon, les coordonnées des neurones dans l'espace de données sont modifiées. Si le seuil n'a pas été dépassé, de nouveaux neurones sont créés qui se rapprochent mieux de la valeur de l'échantillon de données.
- Les connexions et les neurones ont un paramètre d' âge (GNG n'a que des connexions), qui est d'abord nul, mais augmente à mesure que vous apprenez.
- Un neurone n'apparaît pas immédiatement: un embryon (ou neurone germinal) apparaît d'abord, dont l'âge augmente à chaque itération, jusqu'à ce qu'il mûrisse. Après l'entraînement, seuls les neurones matures participent à la classification .
Cycle principal
Le travail commence par un graphique vide. Paramètre initialisé par l' écart-type de l'échantillon d'apprentissage:
Où: - la moyenne entre les coordonnées de l'échantillon.
La boucle principale à chaque étape diminue la valeur , qui est le seuil de proximité, et calcule la différence entre le niveau de qualité de clustering précédent et le niveau obtenu après le clustering par la procédure IGNG .
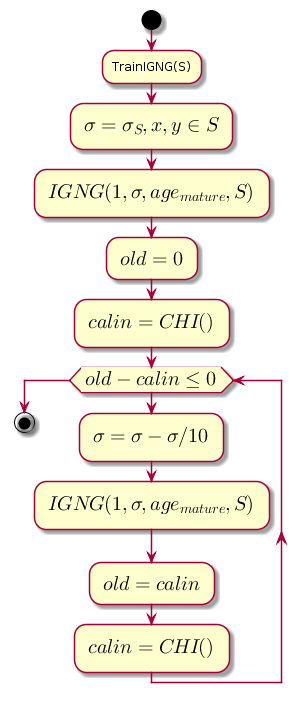
Code graphique.@startuml start :TrainIGNG(S); :<latex>\sigma = \sigma_S,x,y \in S</latex>; :<latex>IGNG(1, \sigma, age_{mature}, S)</latex>; :<latex>old = 0</latex>; :<latex>calin = CHI()</latex>; while (<latex>old - calin \leq 0</latex>) :<latex>\sigma=\sigma - \sigma / 10</latex>; :<latex>IGNG(1, \sigma, age_{mature}, S)</latex>; :<latex>old = calin</latex>; :<latex>calin = CHI()</latex>; endwhile stop @enduml
CHI est l'indice de Kalinsky-Kharabaz, montrant la qualité du clustering:
Où:
- - le nombre d'échantillons de données.
- - le nombre de clusters (dans ce cas, le nombre de neurones).
- - matrice de dispersion interne (la somme des distances au carré entre les coordonnées des neurones et la moyenne de toutes les données).
- - matrice de dispersion externe (somme des distances au carré entre toutes les données et le neurone le plus proche).
Plus la valeur de l'indice est élevée, mieux c'est, car si la différence entre les indices après clustering et avant qu'il soit négatif, il est possible de supposer que l'indice est devenu positif et a dépassé le précédent, c'est-à-dire le clustering s'est terminé avec succès.
Procédure IGNG
Il s'agit de la procédure de base de l'algorithme.
Il est divisé en trois étapes mutuellement exclusives:
- Aucun neurone trouvé.
- Un neurone satisfaisant a été trouvé.
- Trouvé deux qui satisfont les conditions du neurone.
Si l'une des conditions est remplie, les autres étapes ne sont pas exécutées.
Tout d'abord, un neurone est recherché pour le meilleur échantillon de données d'approximation:
Ici - fonction de calcul de distance, qui est généralement une métrique euclidienne .
Si le neurone n'est pas trouvé ou trop éloigné des données, c'est-à-dire ne satisfait pas au critère de proximité , un nouveau neurone embryonnaire est créé avec des coordonnées égales aux coordonnées de l'échantillon dans l'espace de données.

Si la vérification de proximité a réussi, un deuxième neurone est recherché de la même manière et vérifié la proximité de l'échantillon de données.
Si le deuxième neurone n'est pas trouvé, il est créé.

Si deux neurones ont été trouvés qui satisfont à la condition de proximité de l'échantillon de données, leurs coordonnées sont corrigées selon la formule suivante:
Où:
- - étape d'adaptation.
- Est le numéro du neurone.
- - Fonction de voisinage neuronal avec le neurone gagnant (dans ce cas, il renverra 1 pour les voisins directs, 0 sinon, car l'étape d'adaptation, pour le calcul sera différent de zéro uniquement pour les voisins directs).
En d'autres termes, la coordonnée (poids) du neurone gagnant est changée en , et tous ses voisins directs (ceux qui lui sont connectés par un bord du graphique) sur où - les coordonnées du neurone correspondant avant le changement.
Ensuite, une connexion est créée entre les deux neurones gagnants, et si elle a déjà été créée, son âge est réinitialisé.
L'âge de toutes les autres relations augmente.
Toutes les communications dont l'âge a dépassé la constante sont supprimés.
Après cela, tous les neurones matures isolés (ceux qui n'ont aucun lien avec les autres) sont supprimés.
L'âge des neurones immédiats voisins du neurone gagnant augmente.
Si l'âge de l'un des neurones de la lignée germinale dépasse Il devient un neurone mature.
Le graphique final ne contient que des neurones matures.
Une condition pour terminer la procédure IGNG ci-dessous peut être considérée comme un nombre fixe de cycles.
L'algorithme est illustré ci-dessous (l'image est cliquable):
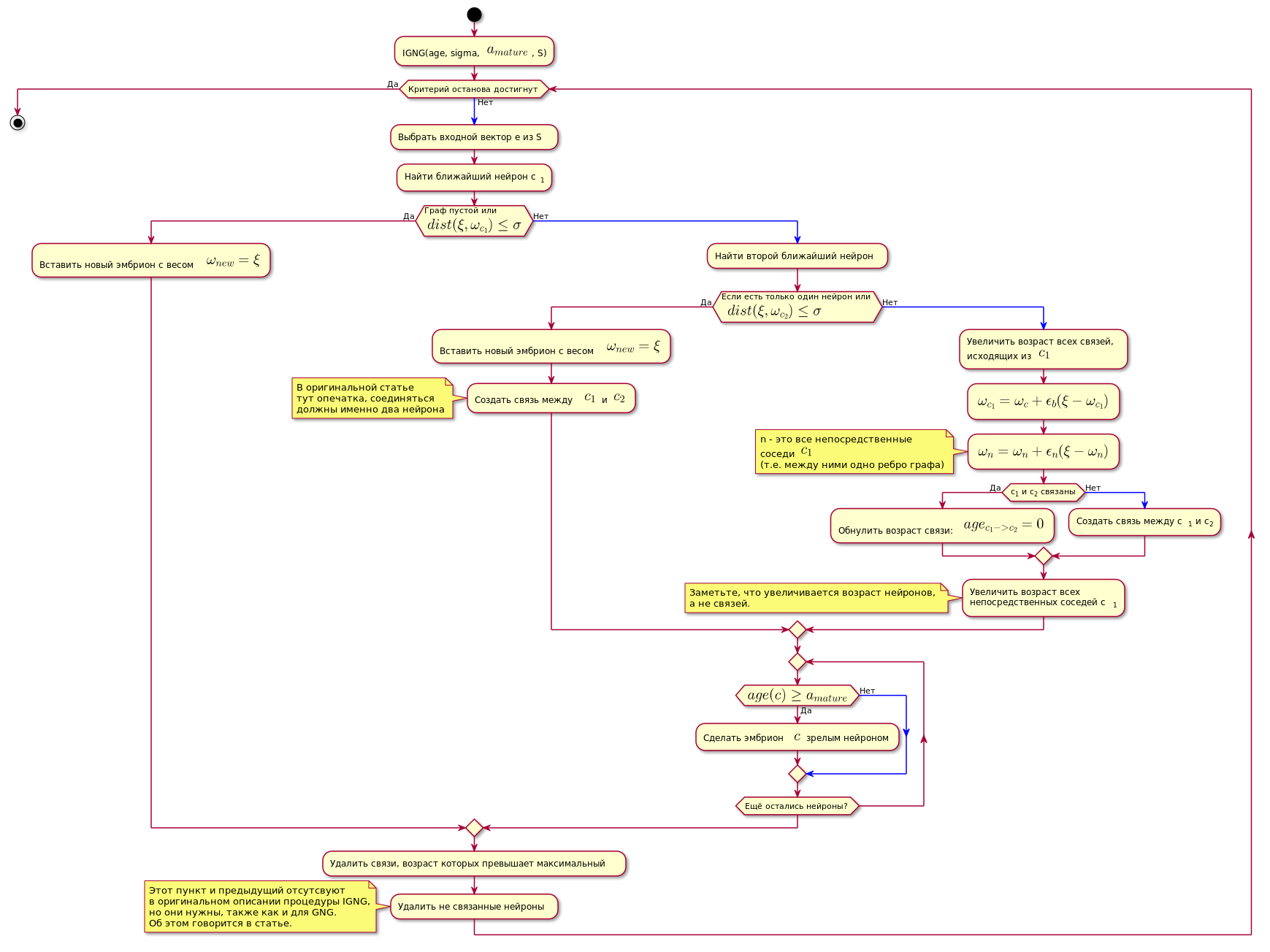
Code graphique. @startuml skinparam nodesep 10 skinparam ranksep 20 start :IGNG(age, sigma, <latex>a_{mature}</latex>, S); while ( ) is () -[#blue]-> : e S; : c<sub>1</sub>; if ( \n<latex>dist(\xi, \omega_{c_1}) \leq \sigma</latex>) then () : <latex>\omega_{new} = \xi</latex>; else () -[#blue]-> : ; if ( \n <latex>dist(\xi, \omega_{c_2}) \leq \sigma</latex>) then () : <latex>\omega_{new} = \xi</latex>; : <latex>c_1</latex> <latex>c_2</latex>; note , end note else () -[#blue]-> : ,\n <latex>c_1</latex>; :<latex>\omega_{c_1} = \omega_c + \epsilon_b(\xi - \omega_{c_1})</latex>; :<latex>\omega_n = \omega_n + \epsilon_n(\xi - \omega_n)</latex>; note n - <latex>c_1</latex> (.. ) end note if (c<sub>1</sub> c<sub>2</sub> ) then () : : <latex>age_{c_1 -> c_2} = 0</latex>; else () -[#blue]-> : c<sub>1</sub> c<sub>2</sub>; endif : \n c<sub>1</sub>; note , , . end note endif repeat if (<latex>age(c) \geq a_{mature}</latex>) then () : $$ ; else () -[#blue]-> endif repeat while ( ?) endif : , ; : ; note IGNG, , GNG. . endnote endwhile () stop @enduml
Implémentation
Le réseau a été implémenté en Python à l'aide de la bibliothèque de graphes NetworkX . La coupe du code du prototype dans l' article précédent est donnée ci-dessous. Il y a aussi de brèves explications pour le code.
Si quelqu'un est intéressé par le code complet, voici un lien vers le référentiel .
Un exemple de l'algorithme:

La majeure partie du code class NeuralGas(): __metaclass__ = ABCMeta def __init__(self, data, surface_graph=None, output_images_dir='images'): self._graph = nx.Graph() self._data = data self._surface_graph = surface_graph