Bonjour à tous!
Parlons, comme vous l'avez peut-être deviné, des réseaux de neurones et de l'apprentissage automatique. D'après le nom, il est clair ce qui sera dit sur les réseaux de densité de mélange, puis juste MDN, je ne veux pas traduire le nom et le laisser tel quel. Oui, oui, oui ... il y aura un peu de mathématiques ennuyeuses et de théorie des probabilités, mais sans cela, malheureusement ou heureusement, c'est à vous de décider s'il est difficile d'imaginer le monde de l'apprentissage automatique. Mais je m'empresse de vous rassurer, ce sera relativement petit et ce ne sera pas très difficile. Quoi qu'il en soit, vous pouvez l'ignorer, mais regardez juste une petite quantité de code en Python et PyTorch, c'est vrai, nous écrirons le réseau en utilisant PyTorch, ainsi que divers graphiques avec les résultats. Mais le plus important est qu'il y aura une opportunité de comprendre un peu et de comprendre ce que sont les réseaux MD.
Eh bien, commençons!
Régression
Pour commencer, rafraîchissons un peu nos connaissances et rappelons brièvement ce qu'est
la régression linéaire .
Nous avons un vecteur
X = \ {x_1, x_2, ..., x_n \}X = \ {x_1, x_2, ..., x_n \} nous devons prédire la valeur
Y , qui dépend en quelque sorte de
X en utilisant un modèle linéaire:
hatY=XT hat beta
Comme fonction d'erreur, nous utiliserons l'erreur quadratique:
SE( beta)= sumni=1(yi− hatyi)2= sumNi=1(yi−xTi hat beta)2
Ce problème peut être résolu directement en prenant la dérivée de SE et en mettant sa valeur à zéro:
frac deltaSE( beta) delta beta=2XT( mathbfy−X beta)=0
Ainsi, nous trouvons simplement son minimum, et SE est une fonction quadratique, ce qui signifie que le minimum existera toujours. Après cela, vous pouvez déjà facilement trouver
beta :
hat beta=(XTX)−1XT mathbfy
C'est tout, le problème est résolu. C'est là que nous finissons de rappeler ce qu'est la régression linéaire.
Bien sûr, la dépendance inhérente à la nature de la génération de données peut être différente et alors une certaine non-linéarité doit déjà être ajoutée à notre modèle. Résoudre le problème de régression directement pour des données volumineuses et réelles est également une mauvaise idée, car il existe une matrice
XTX les dimensions
n foisn , et il faut encore trouver sa matrice inverse, et il arrive souvent qu'une telle matrice n'existe tout simplement pas. Dans ce cas, différentes méthodes basées sur la descente de gradient viennent à notre secours. La non-linéarité des modèles peut être implémentée de différentes manières, notamment en utilisant des réseaux de neurones.
Mais maintenant, ne parlons pas de cela, mais des fonctions d'erreur. Quelle est la différence entre SE et log-vraisemblance lorsque les données peuvent avoir une relation non linéaire?
Nous traitons avec le zoo, à savoir: OLS, LS, SE, MSE, RSSTout cela est une seule et même essence, RSS - somme résiduelle des carrés, OLS - moindres carrés ordinaires, LS - moindres carrés, MSE - erreur quadratique moyenne, SE - erreur quadratique moyenne. Dans différentes sources, vous pouvez trouver des noms différents. L'essence n'en est qu'une:
la déviation quadratique . Vous pouvez vous embrouiller bien sûr, mais vous vous y habituez rapidement.
Il convient de noter que MSE est l'écart-type, une certaine valeur moyenne de l'erreur pour l'ensemble des données d'entraînement. En pratique, MSE est généralement utilisé. La formule n'est pas particulièrement différente:
MSE( beta)= frac1N sumni=1(yi− hatyi)2
N - la taille de l'ensemble de données,
chapeauyi - prédiction du modèle pour
yi .
Arrête ça! Probabilité? C'est quelque chose de la théorie des probabilités. C'est vrai - c'est une pure théorie des probabilités. Mais comment relier l'écart quadratique à la fonction de vraisemblance? Et comment ça se passe. Elle est liée à la recherche du maximum de vraisemblance (Maximum de vraisemblance) et à une distribution normale, pour être plus précis, à sa moyenne
mu .
Afin de réaliser qu'il en est ainsi, regardons à nouveau la fonction de déviation carrée:
RSS( beta)= sumni=1(yi− hatyi)2 qquad qquad(1)
Supposons maintenant que la fonction de vraisemblance ait une forme normale, c'est-à-dire une distribution gaussienne ou normale:
L(X)=p(X| theta)= prodX mathcalN(xi; mu, sigma2)
En général, quelle est la fonction de vraisemblance et quelle est sa signification, je ne le dirai pas, vous pouvez en lire ailleurs, vous devez également vous familiariser avec le concept de probabilité conditionnelle, le théorème de Bayes, et bien plus encore, pour une compréhension plus approfondie. Tout cela entre dans la pure théorie des probabilités, étudiée à la fois à l'école et à l'université.
Maintenant, en se rappelant la formule de distribution normale, nous obtenons:
L(X; mu, sigma2)= prodX frac1 sqrt2 pi sigma2e− frac(xi− mu)22 sigma2 qquad qquad(2)
Et si on mettait l'écart type
sigma2=1 et supprimez toutes les constantes de la formule (2), supprimez simplement, ne réduisez pas, car trouver le minimum de la fonction ne dépend pas d'eux. Ensuite, nous verrons ceci:
L(X; mu, sigma2) sim prodXe−(xi− mu)2
Toujours rien de tel? Non? Et si on prend le logarithme de la fonction? Du logarithme, en général, il y a quelques avantages: la multiplication se transformera en somme, un degré en multiplication, et
loge=1 - pour cette propriété, il convient de préciser que nous parlons du logarithme naturel et, à proprement parler
lne=1 . Et en général, le logarithme d'une fonction ne change pas son maximum, et c'est la caractéristique la plus importante pour nous. La connexion avec Log-Lik vraisemblance et Probabilité et pourquoi cela sera utile sera décrite ci-dessous dans une petite digression. Et donc ce que nous avons fait: supprimé toutes les constantes et pris le logarithme de la fonction de vraisemblance. Ils ont également supprimé le signe moins, transformant ainsi Log-Likelihood en Log-Likelihood négatif (NLL), la connexion entre eux sera également décrite comme un bonus. En conséquence, nous avons obtenu la fonction NLL:
logL(X; mu,I2) sim sum(X− mu)2
Jetez un autre coup d'œil à la fonction RSS (1). Oui, ce sont les mêmes! Exactement! On voit également que
mu= haty .
Si vous utilisez la fonction d'écart type MSE, nous obtenons de ceci:
operatornameargminMSE( beta) sim operatornameargmax mathbbEX simPdata logPmodèle(x; beta)
où
mathbbE - attente mathématique
beta - paramètres du modèle, nous les désignerons à l'avenir comme:
theta .
Conclusion: Si nous utilisons la famille LS comme fonctions d'erreur dans la question de régression, alors nous résolvons essentiellement le problème de trouver la fonction de vraisemblance maximale dans le cas où la distribution est gaussienne. Et la valeur prédite
chapeauy égale à la moyenne dans la distribution normale. Et maintenant, nous savons comment tout cela est connecté, comment la théorie des probabilités (avec sa fonction de vraisemblance et sa distribution normale) et les méthodes d'écart type ou OLS sont connectées. Plus de détails à ce sujet peuvent être trouvés dans [2].
Et voici le bonus promis. Puisque nous parlons des relations entre les différentes fonctions d'erreur, nous considérerons (pas nécessairement à lire):
La relation entre entropie croisée, vraisemblance, log-vraisemblance et log-vraisemblance négativeSupposons que nous ayons des données
X = \ {x_1, x_2, x_3, x_4, ... \} , chaque point appartient à une classe spécifique, par exemple
\ {x_1 \ rightarrow1, x_2 \ rightarrow2, x_3 \ rightarrow n, ... \} . Total là-bas
n classes, tandis que la classe 1 se produit
c1 fois, classe 2 -
c2 horaires et classe
n -
cn fois. Sur ces données, nous avons formé un modèle
theta . La fonction de vraisemblance (vraisemblance) pour elle ressemblera à ceci:
P(data| theta)=P(0,1,...,n| theta)=P(0| theta)P(1| theta)...P(n| theta)
P(1| theta)P(2| theta)...P(n| theta)= prodc1 haty1 prodc2 haty2... prodcn hatyn= hatyc11 hatyc22... hatycnn
où
P(n| theta)= chapeauyn - probabilité prédite pour la classe
n .
Nous prenons le logarithme de la fonction de vraisemblance et obtenons Log-Lik vraisemblance:
logP(data| theta)= log( hatyc11... hatycnn)=c1 log haty1+...+cn log hatyn= sumnici log hatyi
Probabilité
haty in[0,1] se situe dans la plage de 0 à 1, sur la base de la définition de la probabilité. Par conséquent, le logarithme aura une valeur négative. Et si nous multiplions Log-Likelihood par -1, nous obtenons la fonction Negative Log-Likelihood (NLL):
NLL=− logP(data| theta)=− sumnici log hatyi
Si nous divisons le NLL par le nombre de points
X ,
N=c1+c2+...+cn on obtient alors:
- \ frac {1} {N} \ log {P (data | \ theta)} = - \ sum_i ^ n {\ frac {c_i} {N} \ log {\ hat {y_i}}}}
on peut noter que la probabilité réelle pour la classe
n est égal à:
yn= fraccnN . De là, nous obtenons:
NLL=− sumniyi log hatyi
maintenant, si vous regardez la définition de l'entropie croisée
H(p,q)=− sump logq on obtient alors:
NLL=H(yi, hatyi)
Dans le cas où nous n'avons que deux classes
n=2 (classification binaire) nous obtenons la formule d'entropie croisée binaire (vous pouvez également rencontrer le nom bien connu Log-Loss):
H(y, haty)=−(y log haty+(1−y) log(1− haty))
De tout cela, on peut comprendre que dans certains cas, la minimisation de l'entropie croisée équivaut à la minimisation des NLL ou à la recherche du maximum de la fonction de vraisemblance (vraisemblance) ou log-vraisemblance.
Un exemple. Considérons une classification binaire. Nous avons des valeurs de classe:
y = np.array([0, 1, 1, 1, 1, 0, 1, 1]).astype(np.float32)
Probabilité réelle
y pour la classe 0 est égal

, pour la classe 1 est égal

. Supposons que nous ayons un classificateur binaire qui prédit la probabilité de classe 0
chapeauy pour chaque exemple, respectivement, pour la classe 1, la probabilité est
(1− chapeauy) . Tracons les valeurs de la fonction Log-Loss pour différentes prédictions
chapeauy :
Sur le graphique, vous pouvez voir que le minimum de la fonction Log-Loss correspond au point 0,75, c'est-à-dire si notre modèle a complètement «appris» la distribution des données sources, chapeauy=y . Régression du réseau neuronal
Nous sommes donc arrivés à une pratique plus intéressante. Voyons comment vous pouvez résoudre le problème de la régression en utilisant des réseaux de neurones (réseaux de neurones). Nous allons implémenter tout dans le langage de programmation Python, pour créer un réseau que nous utilisons la bibliothèque d'apprentissage en profondeur PyTorch.
Génération de données source
Entrer les données
mathbfX in mathbbRN générer en utilisant une distribution uniforme, prendre l'intervalle de -15 à 15,
mathbfX enU[−15,15] . Des points
mathbfY on obtient en utilisant l'équation:
mathbfY=0,5 mathbfX+8 sin(0,3 mathbfX)+bruit qquad qquad(3)
où
bruit Est un vecteur de bruit de dimension
N obtenu en utilisant la distribution normale avec des paramètres:
mu=0, sigma2=1 .
Le graphique des données reçues.Création de réseaux
Créez un réseau de neurones à rétroaction régulière ou FFNN.
Bâtiment FFNN class Net(nn.Module): def __init__(self, input_dim=IN_DIM, out_dim=OUT_DIM, layer_size=40): super(Net, self).__init__() self.fc = nn.Linear(input_dim, layer_size) self.logit = nn.Linear(layer_size, out_dim) def forward(self, x): x = F.tanh(self.fc(x))
Notre réseau est constitué d'une couche cachée d'une dimension de 40 neurones et d'une fonction d'activation - tangente hyperbolique:
tanhx= fracex−e−xex+e−x qquad qquad(4)
La couche de sortie est une transformation linéaire normale sans fonction d'activation.
Apprendre et obtenir des résultats
En tant qu'optimiseur, nous utiliserons AdamOptimizer. Le nombre d'époques d'étude = 2000, le taux d'apprentissage (taux d'apprentissage ou lr) = 0,1.
Formation FFNN def train(net, x_train, y_train, x_test, y_test, epoches=2000, lr=0.1): criterion = nn.MSELoss() optimizer = optim.Adam(net.parameters(), lr=lr) N_EPOCHES = epoches BS = 1500 n_batches = int(np.ceil(x_train.shape[0] / BS)) train_losses = [] test_losses = [] for i in range(N_EPOCHES): for bi in range(n_batches): x_batch, y_batch = fetch_batch(x_train, y_train, bi, BS) x_train_var = Variable(torch.from_numpy(x_batch)) y_train_var = Variable(torch.from_numpy(y_batch)) optimizer.zero_grad() outputs = net(x_train_var) loss = criterion(outputs, y_train_var) loss.backward() optimizer.step() with torch.no_grad(): x_test_var = Variable(torch.from_numpy(x_test)) y_test_var = Variable(torch.from_numpy(y_test)) outputs = net(x_test_var) test_loss = criterion(outputs, y_test_var) test_losses.append(test_loss.item()) train_losses.append(loss.item()) if i%100 == 0: sys.stdout.write('\r Iter: %d, test loss: %.5f, train loss: %.5f' %(i, test_loss.item(), loss.item())) sys.stdout.flush() return train_losses, test_losses net = Net() train_losses, test_losses = train(net, x_train, y_train, x_test, y_test)
Examinons maintenant les résultats d'apprentissage.
Graphique des valeurs de la fonction MSE en fonction de l'itération de la formation; graphique des valeurs des données de formation et des données de test.Résultats réels et prévus sur les données de test.Données inversées
Nous compliquons la tâche et inversons les données.
Inversion de données x_train_inv = y_train y_train_inv = x_train x_test_inv = y_train y_test_inv = x_train
Graphique de données inversé.Pour la prédiction
mathbf hatY utilisons le réseau de distribution direct de la section précédente et voyons comment il gère cela.
inv_train_losses, inv_test_losses = train(net, x_train_inv, y_train_inv, x_test_inv, y_test_inv)
Graphique des valeurs de la fonction MSE en fonction de l'itération de la formation; graphique des valeurs des données de formation et des données de test.Résultats réels et prévus sur les données de test.Comme vous pouvez le voir sur les graphiques ci-dessus, notre réseau
n'a pas du tout géré ces données, il n'est tout simplement pas en mesure de les prédire. Et tout cela est arrivé parce que dans un tel problème inversé pour un point
x peut correspondre à plusieurs points
y . Vous demandez, qu'en est-il du bruit? Il a également créé une situation dans laquelle, pour une
x pourrait obtenir quelques valeurs
y . Oui, c'est vrai. Mais le fait est que, malgré le bruit, tout était une distribution définie. Et puisque notre modèle prédit essentiellement
p(y|x) , et dans le cas de MSE, c'était la valeur moyenne de la distribution normale (pourquoi elle est décrite dans la première partie de l'article), puis elle a bien fait face à la tâche «directe». Sinon, nous obtenons plusieurs distributions différentes pour une
x et en conséquence, nous ne pouvons pas obtenir un bon résultat avec une seule distribution normale.
Réseau de densité de mélange
Le plaisir commence! Qu'est-ce que le réseau de densité de mélange (ci-après réseau MDN ou MD)? En général, il s'agit d'un certain modèle capable de simuler plusieurs distributions à la fois:
p( mathbfy| mathbfx; theta)= sumKk pik( mathbfx) mathcalN( mathbfy; muk( mathbfx), sigma2( mathbfx)) qquad qquad(5)
Quelle étrange formule, dites-vous. Voyons cela. Notre réseau MD apprend à modéliser la moyenne
mu et variance
sigma2 pour
plusieurs distributions. Dans la formule (5)
pik( mathbfx) - les soi-disant facteurs de signification d'une distribution distincte pour chaque point
xi in mathbfx , un certain facteur de mélange ou la contribution de chacune des distributions à un certain point. Total là-bas
K distributions.
Encore quelques mots sur
pik( mathbfx) - en fait, c'est aussi une distribution et représente la probabilité que pour un point
xi in mathbfx sera une condition
k .
Fuh, encore une fois, ce calcul, écrivons déjà quelque chose. Et donc, nous allons commencer à réaliser un réseau. Pour notre réseau nous prenons
K=30 .
self.fc = nn.Linear(input_dim, layer_size) self.fc2 = nn.Linear(layer_size, 50) self.pi = nn.Linear(layer_size, coefs) self.mu = nn.Linear(layer_size, out_dim*coefs)
Définissez les couches de sortie pour notre réseau:
x = F.relu(self.fc(x)) x = F.relu(self.fc2(x)) pi = F.softmax(self.pi(x), dim=1) sigma_sq = torch.exp(self.sigma_sq(x)) mu = self.mu(x)
Nous écrivons la fonction d'erreur ou la fonction de perte, formule (5):
def gaussian_pdf(x, mu, sigma_sq): return (1/torch.sqrt(2*np.pi*sigma_sq)) * torch.exp((-1/(2*sigma_sq)) * torch.norm((x-mu), 2, 1)**2) losses = Variable(torch.zeros(y.shape[0]))
Code de construction MDN complet COEFS = 30 class MDN(nn.Module): def __init__(self, input_dim=IN_DIM, out_dim=OUT_DIM, layer_size=50, coefs=COEFS): super(MDN, self).__init__() self.fc = nn.Linear(input_dim, layer_size) self.fc2 = nn.Linear(layer_size, 50) self.pi = nn.Linear(layer_size, coefs) self.mu = nn.Linear(layer_size, out_dim*coefs)
Notre réseau MD est prêt à fonctionner. Presque prêt. Reste à la former et à regarder les résultats.
Formation MDN def train_mdn(net, x_train, y_train, x_test, y_test, epoches=1000): optimizer = optim.Adam(net.parameters(), lr=0.01) N_EPOCHES = epoches BS = 1500 n_batches = int(np.ceil(x_train.shape[0] / BS)) train_losses = [] test_losses = [] for i in range(N_EPOCHES): for bi in range(n_batches): x_batch, y_batch = fetch_batch(x_train, y_train, bi, BS) x_train_var = Variable(torch.from_numpy(x_batch)) y_train_var = Variable(torch.from_numpy(y_batch)) optimizer.zero_grad() pi, mu, sigma_sq = net(x_train_var) loss = loss_fn(y_train_var, pi, mu, sigma_sq) loss.backward() optimizer.step() with torch.no_grad(): if i%10 == 0: x_test_var = Variable(torch.from_numpy(x_test)) y_test_var = Variable(torch.from_numpy(y_test)) pi, mu, sigma_sq = net(x_test_var) test_loss = loss_fn(y_test_var, pi, mu, sigma_sq) train_losses.append(loss.item()) test_losses.append(test_loss.item()) sys.stdout.write('\r Iter: %d, test loss: %.5f, train loss: %.5f' %(i, test_loss.item(), loss.item())) sys.stdout.flush() return train_losses, test_losses mdn_net = MDN() mdn_train_losses, mdn_test_losses = train_mdn(mdn_net, x_train_inv, y_train_inv, x_test_inv, y_test_inv)
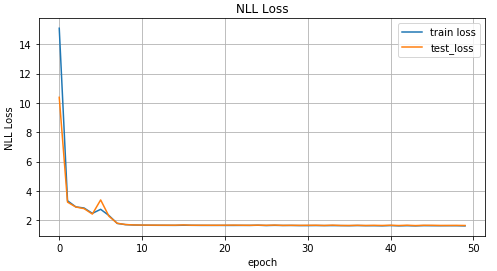
Puisque notre réseau a appris les valeurs moyennes de plusieurs distributions, regardons ceci:
pi, mu, sigma_sq = mdn_net(Variable(torch.from_numpy(x_test_inv)))
Graphique pour les deux valeurs moyennes les plus probables pour chaque point (à gauche). Graphique pour les 4 valeurs moyennes les plus probables pour chaque point (à droite).Graphique pour toutes les valeurs moyennes pour chaque point.Pour prédire les données, nous sélectionnerons aléatoirement plusieurs valeurs
mu et
sigma2 basé sur la valeur
pik( mathbfx) . Et puis en fonction d'eux pour générer des données cibles
chapeauy en utilisant une distribution normale.
Prédiction du résultat def rand_n_sample_cumulative(pi, mu, sigmasq, samples=10): n = pi.shape[0] out = Variable(torch.zeros(n, samples, OUT_DIM)) for i in range(n): for j in range(samples): u = np.random.uniform() prob_sum = 0 for k in range(COEFS): prob_sum += pi.data[i, k] if u < prob_sum: for od in range(OUT_DIM): sample = np.random.normal(mu.data[i, k*OUT_DIM+od], np.sqrt(sigmasq.data[i, k])) out[i, j, od] = sample break return out pi, mu, sigma_sq = mdn_net(Variable(torch.from_numpy(x_test_inv))) preds = rand_n_sample_cumulative(pi, mu, sigma_sq, samples=10)
Données prédites pour 10 valeurs sélectionnées au hasard mu et sigma2 (à gauche) et pour deux (à droite).Les chiffres montrent que MDN a fait un excellent travail avec la tâche «inverse».
Utiliser des données plus complexes
Voyons comment notre réseau MD gère les données plus complexes, telles que les données en spirale. L'équation de la spirale hyperbolique en coordonnées cartésiennes:
x= rho cos phi qquad qquad qquad qquad qquad qquad(6)y= rho sin phi
Génération de données en spirale N = 2000 x_train_compl = [] y_train_compl = [] x_test_compl = [] y_test_compl = [] noise_train = np.random.uniform(-1, 1, (N, IN_DIM)).astype(np.float32) noise_test = np.random.uniform(-1, 1, (N, IN_DIM)).astype(np.float32) for i, theta in enumerate(np.linspace(0, 5*np.pi, N).astype(np.float32)):
Graphique des données en spirale.Pour le plaisir, voyons comment un réseau Feed-Forward ordinaire fera face à une telle tâche.
Comme prévu, le réseau Feed-Forward n'est pas en mesure de résoudre le problème de régression pour ces données.Nous utilisons le réseau MD décrit et créé précédemment pour la formation sur les données en spirale.
Mixture Density Network a fait un excellent travail dans cette situation.Conclusion
Au début de cet article, nous avons rappelé les bases de la régression linéaire. Nous avons vu cela en commun entre trouver la moyenne pour la distribution normale et MSE. Démonté comment connecté NLL et entropie croisée. Et surtout, nous avons trouvé le modèle MDN, qui est capable d'apprendre des données obtenues à partir d'une distribution mixte. J'espère que l'article est compréhensible et intéressant, malgré le fait qu'il y ait eu un peu de maths.
Le code complet peut être consulté sur
GitHub .
Littérature
- Réseaux de densité de mélange (Christopher M. Bishop, Neural Computing Research Group, Dept. of Computer Science and Applied Mathematics, Aston University, Birmingham) - l'article décrit en détail la théorie des réseaux MD.
- Moindres carrés et maximum de vraisemblance (MROsborne)