Mi-2018, les
travaux sur l'électrophysiologie du cerveau des rats ont été
publiés , avec lesquels un
ensemble de données unique a été rendu public. L'ensemble de données est unique en ce qu'il contient des enregistrements simultanés du potentiel de champ local à l'aide d'une nouvelle électrode
Neuropixels haute densité (sonde ou sonde) et d'une électrode patch provenant d'une cellule située près de l'échantillon. L'intérêt pour de tels enregistrements est non seulement fondamental, mais également appliqué, car il permet de valider des modèles d'analyse de l'activité neuronale enregistrés par des échantillons modernes. Et cela, à son tour, est directement lié au développement de nouvelles neuro-prothèses. Quelle est la nouveauté fondamentale et pourquoi cet ensemble de données est si important - je vais vous dire sous la coupe.
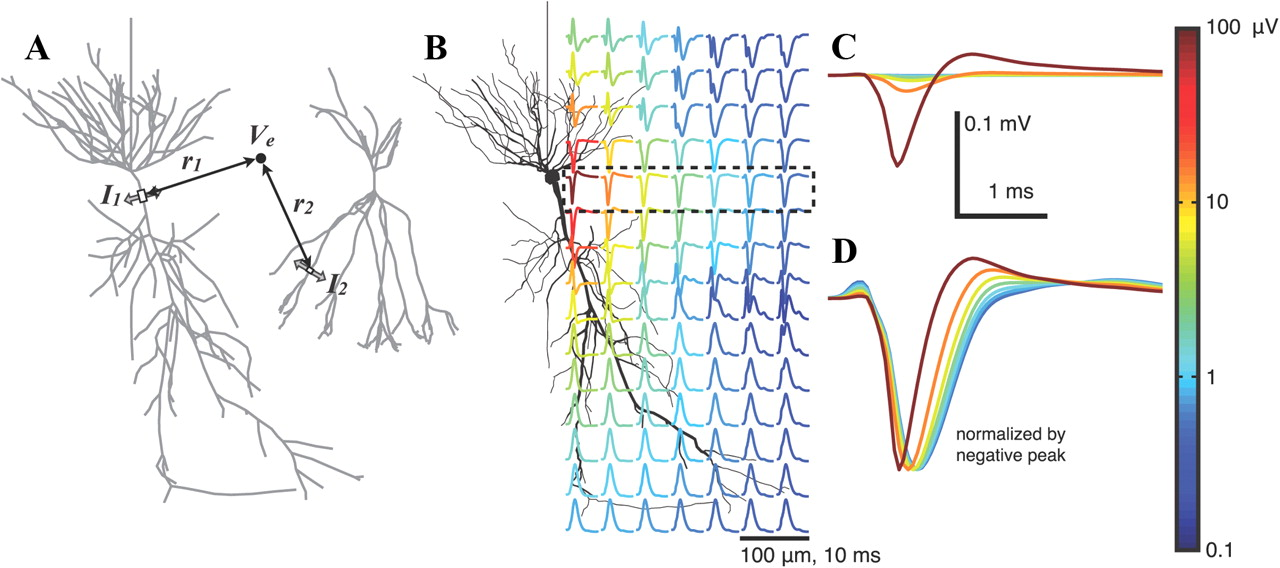 KDPV: résultat de la modélisation du potentiel extracellulaire à proximité d'un neurone lors de la génération du potentiel d'action ( source ). La couleur indique l'amplitude du potentiel. Cette illustration sera importante pour une meilleure compréhension.Les méthodes électrophysiologiques pour étudier le
KDPV: résultat de la modélisation du potentiel extracellulaire à proximité d'un neurone lors de la génération du potentiel d'action ( source ). La couleur indique l'amplitude du potentiel. Cette illustration sera importante pour une meilleure compréhension.Les méthodes électrophysiologiques pour étudier le cerveau sont basées sur l'enregistrement du potentiel électrique du cerveau. Ils peuvent être divisés en non invasifs - principalement l'électroencéphalographie (EEG) - et invasifs, par exemple, l'électrocorticographie (ECoG), le patch-clamp (patch-clamp) ou l'enregistrement du potentiel de champ local (LPP = potentiels de champ local, LFP) . Pour ce dernier, une petite électrode de 10 à 100 microns est injectée directement dans le cerveau et son potentiel est enregistré. Afin d'étudier l'activité du cerveau de mammifère au niveau cellulaire, c'est-à-dire de mesurer l'activité de cellules individuelles, les méthodes non invasives disponibles ne peuvent pas être appliquées, car le potentiel d'une cellule se désintègre très rapidement dans l'espace, littéralement pour 100 μm (voir KDPV). Par conséquent, dans tout modèle animal, ainsi que chez l'homme, les méthodes non invasives fournissent des informations uniquement sur l'activité collective des neurones et travaillent, très probablement, au niveau des tissus, mais pas sur les neurones individuels.
Mais avec les méthodes invasives, ce n'est pas si simple. Pour enregistrer l'activité d'un neurone, il est nécessaire de rapprocher l'électrode très près du neurone, de la placer idéalement à l'intérieur de la cellule, comme cela se fait dans le patch clamp, ou d'utiliser
des électrodes pointues , ce qui est difficile en pratique, très difficile. D'un autre côté, toute électrode extracellulaire de ~ 10 μm enregistrera les potentiels d'action de 5 à 10 cellules autour en raison de la haute densité de neurones et de la conductivité ionique élevée de la solution extracellulaire. Par conséquent, la tâche d'enregistrement des cellules individuelles est techniquement résolue en augmentant la densité des électrodes situées à proximité de la cellule. À cet égard, l'électrophysiologie moderne s'oriente vers une augmentation de la densité des électrodes, une augmentation de leur nombre et une diminution de leur taille. Parmi les exigences, il est nécessaire d'amplifier le signal plus près du site d'enregistrement afin de réduire le bruit et de placer un multiplexeur pour réduire la taille. Ainsi, en 2016, il a été annoncé dans une préimpression, et en 2017, il a été publié dans Nature, et en 2018, il est déjà apparu
sur le marché , un nouvel échantillon de Neuropixels haute densité fabriqué par la technologie CMOS, sur 960 électrodes, dont 384 sont disponibles pour un enregistrement simultané. La taille d'un site d'enregistrement est de 12 microns. L'épaisseur de l'échantillon est de 24 microns. De plus, avec des électrodes haute densité, ainsi qu'avec une amplification active, les gens ont commencé à travailler pendant longtemps, mais Neuropixels a été le premier à réaliser la production et la vente, donc dans un avenir proche, ce test particulier dans les articles sera de plus en plus fréquent.
 Fig. Schéma des neuropixels. Sur un substrat de silicium monolithique, il y a 960 sites, ainsi qu'un multiplexeur complet et une interface AD pour 384 canaux.
Fig. Schéma des neuropixels. Sur un substrat de silicium monolithique, il y a 960 sites, ainsi qu'un multiplexeur complet et une interface AD pour 384 canaux.Structure des données
En plus des
rythmes d'activité classiques (alpha, bêta, gamma, etc.) qui sont responsables de la synchronisation de groupe, les données obtenues en utilisant de tels échantillons contiennent également les potentiels d'action des cellules individuelles (AP = potentiels d'action, AP, pointes, adhérences) , qui sur l'enregistrement ressemblent à de courts pics d'une durée de ~ 1 ms.
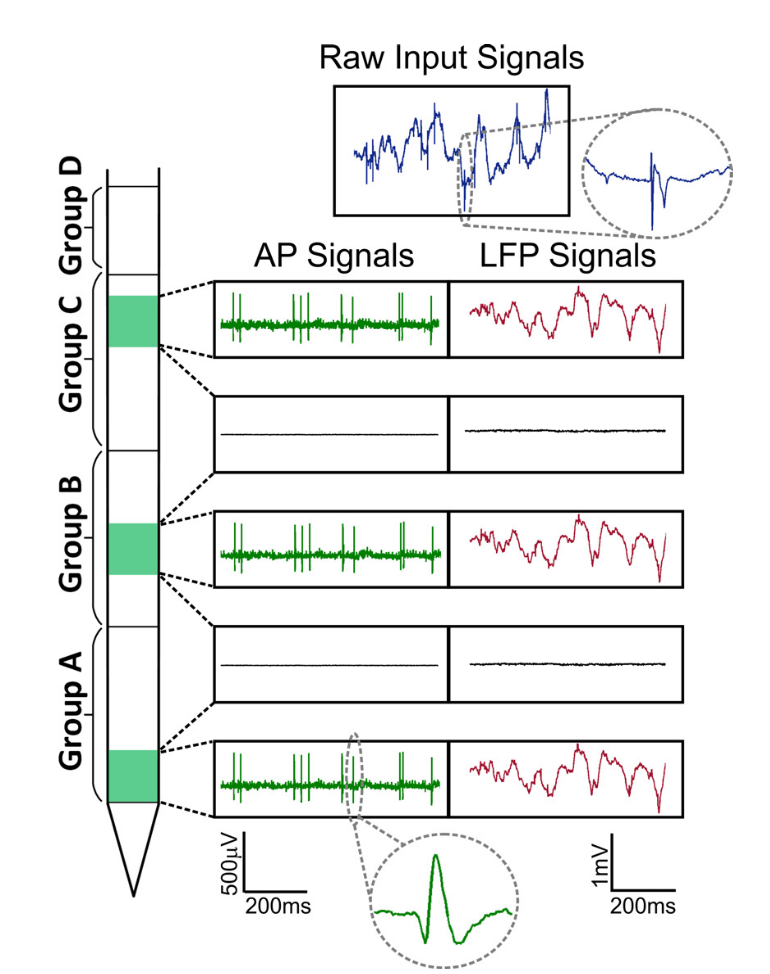 Fig. Signaux Neuropixels. On distingue deux parties du signal: le potentiel de champ local (LFP, jusqu'à ~ 300 Hz) et l'activité cellulaire (AP, à partir de 300 Hz).
Fig. Signaux Neuropixels. On distingue deux parties du signal: le potentiel de champ local (LFP, jusqu'à ~ 300 Hz) et l'activité cellulaire (AP, à partir de 300 Hz).Dans le même temps, si le potentiel de champ local basse fréquence est généralement analysé dans le cadre d'oscillations et qu'une analyse spectrale ou ondelette est utilisée comme dans l'EEG, alors l'activité cellulaire contient les potentiels d'action des cellules individuelles, elle est constituée d'événements discrets sur fond de bruit. La tâche d'isoler l'activité des cellules individuelles se réduit formellement à la tâche d'un problème de cocktail, lorsqu'un orateur séparé doit être distingué de l'ensemble des orateurs. Les mégadonnées apparaissent lorsque nous évaluons le flux de données d'un tel échantillon. Pour l'analyse des pointes, l'échantillonnage est effectué à 30–40 kHz avec numérisation à partir de 16 bits par point (uint16), ainsi, l'enregistrement de 100 électrodes déjà en 1 seconde pèsera à partir de 8 Mo. Dans le même temps, les expériences durent généralement des heures, ce qui équivaut à des centaines de gigaoctets sur une seule journée de travail, et pour une étude complète, disons, à partir de 10 de ces enregistrements. Par conséquent, le potentiel de cet échantillon dépend également fortement des algorithmes d'apprentissage automatique, qui sont utilisés pour l'analyse des données.
Apprentissage automatique et activité cellulaire
En règle générale, une ligne d'analyse d'analyse de l'activité cellulaire consiste en un prétraitement, une segmentation des pointes et un regroupement. Cette partie de la recherche est communément appelée analyse des grappes ou tri des pointes. En tant que prétraitement, un filtrage passe-bas (> 300 Hz) est généralement utilisé, car on pense qu'il n'y a pas d'autres rythmes physiologiques au-dessus de 300 Hz, et il ne reste que des informations sur l'activité cellulaire individuelle. De plus, lors du prétraitement dans des échantillons denses, il est possible de réduire le bruit corrélé, par exemple, des micros à 50 Hz. La segmentation est le plus souvent considérée comme un simple seuil, par exemple, tout élément supérieur à 5 écarts de bruit standard peut être considéré comme un événement. Il arrive que la segmentation à deux seuils soit utilisée, avec un seuil doux et dur, pour distinguer les événements liés dans l'espace et le temps, comme dans l'algorithme de
segmentation des bassins versants, uniquement dans le clustering de cluster, la propagation des marqueurs prend en compte la topologie de l'échantillon. Après la segmentation, une fenêtre d'une durée de 1 à 2 ms est prise près du centre de chaque événement, et le signal dans cette fenêtre, collecté sur tous les canaux, devient un échantillon pour un regroupement ultérieur. Cet échantillon est appelé la forme d'onde de pointe. Différentes cellules et leur distance différente du site d'enregistrement conduisent au fait que leurs formes d'onde varieront (voir KDPV). L'algorithme de clustering de formes d'onde utilise lui-même EM, la recherche de correspondance de modèles, l'apprentissage en profondeur et de nombreuses variantes (
sujet sur github ). La seule exigence est une formation sans professeur. Mais il y a un problème. Personne ne sait avec certitude quels paramètres vous devez prendre pour votre pipeline afin que l'analyse soit la plus efficace. Habituellement, après le clustering, l'analyste examine manuellement les résultats et apporte des modifications à sa discrétion. Ainsi, les résultats de l'analyse peuvent inclure à la fois des erreurs d'algorithme et des erreurs humaines. Et ils peuvent ne pas être, par conséquent, la question de la validation objective reste ouverte.
Il existe plusieurs façons de valider un pipeline. Tout d'abord, changer les conditions externes de l'objet d'étude. Par exemple, pendant une expérience, si vous étudiez les parties visuelles du cortex, vous pouvez modifier la texture, la couleur et la luminosité de l'image. Si dans l'analyse il y a une cellule qui change son activité en fonction du stimulus, alors vous avez de la chance. Deuxièmement, vous pouvez augmenter ou diminuer pharmacologiquement l'activité d'un type particulier de cellule, par exemple en utilisant certains bloqueurs de canaux. Ensuite, l'activité de votre cellule augmentera / diminuera et vous verrez la différence de regroupement. Cependant, une telle modulation d'activité entraînera également des changements dans les formes d'onde, car le profil du potentiel d'action dans le temps est complètement déterminé par la cinétique des canaux ioniques. Troisièmement, vous pouvez mesurer ou induire l'activité de certaines cellules optogénétiquement ou en utilisant une pipette de patch, comme dans cet ensemble de données. En raison du grand rapport signal / bruit et de la stabilité de l'électrode patch, vous serez totalement confiant dans l'activité d'une seule cellule. Conceptuellement, la publication était dédiée à l'assemblage d'un ensemble de données de validation à l'aide d'un patch clamp.
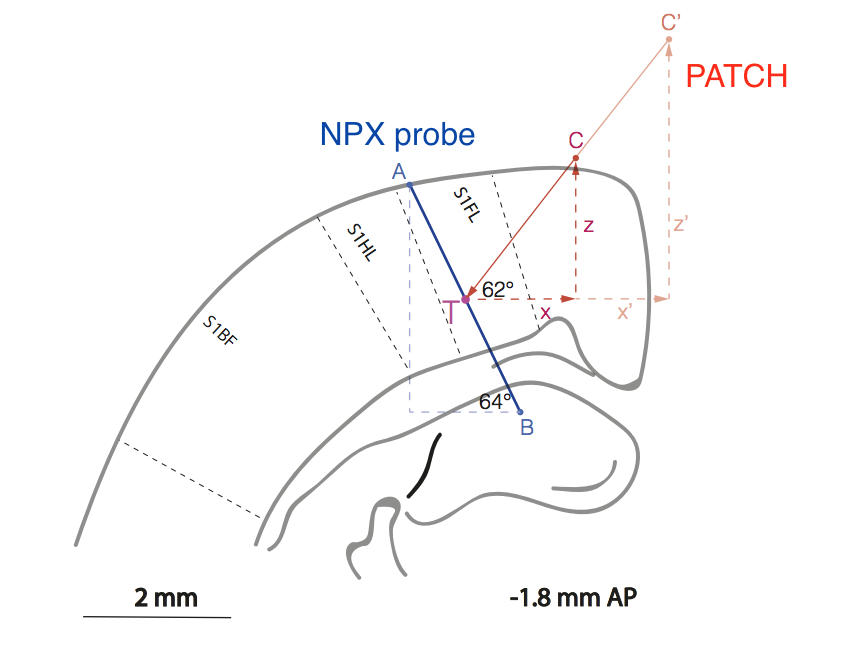 Fig. Représentation schématique de la position relative de l'échantillon (ligne AB) et de la pipette de patch (ligne C'CT) dans la région du cortex du rat, qui est responsable du traitement des informations sensorielles de la patte avant (S1FL = membre antérieur du cortex sensoriel 1).
Fig. Représentation schématique de la position relative de l'échantillon (ligne AB) et de la pipette de patch (ligne C'CT) dans la région du cortex du rat, qui est responsable du traitement des informations sensorielles de la patte avant (S1FL = membre antérieur du cortex sensoriel 1).Inutile de dire que le travail méthodologique est extrêmement difficile, car les expérimentateurs ont dû développer une méthode pour l'arrangement mutuel de deux électrodes dans le cortex cérébral sans contrôle visuel avec une précision de ~ 10 μm.
L'effet de la densité des électrodes sur le regroupement des pointes
Pourquoi est-il si important d'augmenter la densité des sites d'inscription? Pour une analogie, nous prenons le fait connu parmi les chercheurs de l'EEG que, à partir d'un certain seuil, une augmentation du nombre d'électrodes dans le capuchon n'entraîne pas une augmentation notable des informations reçues, c'est-à-dire que le signal de l'électrode diffère légèrement de l'interpolation linéaire des signaux des électrodes voisines. Quelqu'un dit que ce seuil est déjà atteint à 30, quelqu'un à 50, quelqu'un à 100 électrodes. Ceux qui travaillent avec EEG en détail peuvent être corrigés. Mais dans le cas de l'activité cellulaire, le seuil de densité des sites d'enregistrement sur un échantillon n'est pas encore connu, donc la course des échantillons à haute densité se poursuit. Pour cela, l'équipe du Kampff Lab continue de travailler avec un échantillon avec un site de 5x5 μm
2 , et
des données préliminaires ont été publiées pour cela. Les spécialistes travaillant avec des électrodes denses partagent leur expérience selon laquelle, de manière inattendue, le nombre spécifique de cellules individuelles qui peuvent être isolées d'échantillons de la même zone est plus élevé là où la densité des sites d'enregistrement est plus élevée. Cet effet est bien illustré dans
une autre étude des mêmes co-auteurs, qui n'ont sélectionné artificiellement qu'une partie des sites à partir d'un échantillon dense et évalué visuellement la qualité des grappes résultantes après la conversion du tSNE en valeurs PCA à partir de pointes de forme d'onde. Ce n'est pas un canon pour le clustering, mais c'est bon pour illustrer les dépendances. À titre de test, Neuroseeker a agi sur 128 canaux d'une taille totale de 700x70 μm
2 avec un site de 20x20 μm
2 .
 Fig. TSNE sur des diagrammes PCA sur des formes d'ondes brutes tout en réduisant artificiellement la densité de sites sur l'échantillon. Les chantiers sont représentés schématiquement en haut de chaque diagramme. Il est clairement montré comment le nombre de grappes ségrégées augmente avec l'augmentation de la densité du site, A est le meilleur, F est le pire.
Fig. TSNE sur des diagrammes PCA sur des formes d'ondes brutes tout en réduisant artificiellement la densité de sites sur l'échantillon. Les chantiers sont représentés schématiquement en haut de chaque diagramme. Il est clairement montré comment le nombre de grappes ségrégées augmente avec l'augmentation de la densité du site, A est le meilleur, F est le pire.Quelle est l'essence du travail
Dans les données de Marques-Smith et al. il y a enregistrement simultané du patch clamp et des échantillons. En utilisant des données de patchclamp, les scientifiques ont trouvé des moments de potentiels d'action et ont utilisé ces moments pour segmenter et faire la moyenne des formes d'onde déjà présentes sur l'échantillon. En conséquence, ils ont pu construire des distributions de très haute qualité du potentiel d'action dans le temps et l'espace sur toute la zone d'échantillonnage.
 Fig. À gauche, des traces d'activité cellulaire sont affichées simultanément dans le clip de patch (noir) et sur le canal Neuropixels le plus proche (bleu). Au milieu - 500 échantillons individuels et leur moyenne. À droite, la répartition du potentiel d'action dans l'espace sur la zone d'échantillonnage et dans le temps.
Fig. À gauche, des traces d'activité cellulaire sont affichées simultanément dans le clip de patch (noir) et sur le canal Neuropixels le plus proche (bleu). Au milieu - 500 échantillons individuels et leur moyenne. À droite, la répartition du potentiel d'action dans l'espace sur la zone d'échantillonnage et dans le temps.Ensuite, la question se pose de la variation de la forme d'onde extracellulaire de pic à pic - oui, elle est palpable et doit être prise en compte. Ils montrent ensuite qu'il est fondamentalement possible de suivre la distribution du potentiel d'action le long de la membrane cellulaire à l'aide de leurs électrodes denses, mais cela a déjà été montré plus tôt dans les travaux d'autres groupes. En conclusion, ils offrent aux collaborateurs potentiels des questions fondamentales de la neurophysiologie, auxquelles vous pouvez essayer de répondre à l'aide de leur jeu de données, et proposent également d'utiliser le jeu de données pour valider les pipelines pour regrouper l'activité cellulaire. Ce dernier sonne comme un défi audacieux, car il existe actuellement de nombreux algorithmes de clustering et la concurrence entre les méthodes est très grande. Tout d'abord, toutes les méthodes ne fonctionnent pas avec un si grand nombre de canaux et, d'autre part, tout le monde ne peut pas offrir un clustering de haute qualité objective.
Et ensuite
Premièrement, une nouvelle version de
Neuroseeker sur 1300 canaux utilisant également les technologies CMOS approche, des données préliminaires sont
déjà disponibles .
Deuxièmement, nous attendons un autre ensemble de données, déjà du Allen Institute for Brain Science, qui a été annoncé lors de la conférence FENS en 2018. Il utilisera simultanément 4 (!) Échantillons de Neuropixels pour étudier les sections visuelles du cortex de la souris avec divers stimuli visuels. Ils ont promis de publier fin 2018
ici , à côté des données biphotoniques (également un ensemble de données très puissant), mais jusqu'à présent rien.
Troisièmement, la tâche de regrouper les cellules de l'enregistrement du potentiel extracellulaire me semble esthétiquement belle. Il fait converger les méthodes de la microélectronique, de la neurophysiologie et du machine learning. De plus, il a une grande valeur fondamentale et appliquée. Je suppose que le public du habr sera intéressé à en apprendre davantage sur la cuisine technique de l'électrophysiologie, à savoir sur les algorithmes de clustering, car dans ce domaine a déjà développé son propre zoo. À mon tour, j'ai accumulé plusieurs questions pour ces algorithmes, et un tel ensemble de données ne peut pas être ignoré. Par conséquent, dans la partie suivante, nous passerons à l'analyse de certains algorithmes, en commençant par le canonique Klustakwik, en poursuivant avec les méthodes de modèle Kilosort ou Spyking Circus, puis YASS, qui
déclare très fortement à son
sujet qu'il fonctionne plus rapidement et mieux que tout le monde, parce que DL et parce qu'il le peut. Sujet sur github avec une liste de quelques clusters
ici . Anticipant certaines questions, je ne vois pas l'intérêt de développer mon propre algorithme, car la concurrence est déjà très grande, et beaucoup d'idées ont déjà été mises en œuvre et testées par d'autres. Mais s'il y a des casse-cou, je contribuerai avec plaisir.
Les suggestions et souhaits sont acceptés. Merci de votre attention!