Qui n'a pas aimé collectionner les constructeurs dans l'enfance? Je me souviens encore de cette boîte rouge avec un tas de pièces métalliques, d'outils et une mer de résultats possibles, si seulement il y avait de la fantaisie, du temps et du désir. LEGO ne doit pas non plus être oublié, bien qu'ici tout était un peu plus simple et beaucoup plus coloré. Mais ce qui est plus compliqué des deux, ce sont les concepteurs de nanostructures basées sur l'ADN origami. Jusqu'à présent, tous les "détails" de ces structures étaient modélisés manuellement, ce qui prenait beaucoup de temps et d'efforts. Imaginez que vous devez créer toutes les pièces LEGO vous-même avant de les assembler en un robot géant avec des lasers, des moteurs à réaction et une mitrailleuse sur votre épaule. Mais quelque chose de souvenirs d'enfance nous a conduits dans la mauvaise steppe.
Blagues à part. Aujourd'hui, nous allons découvrir l'algorithme qui vous permet de créer automatiquement un origami d'ADN d'une forme assez diversifiée. Auparavant, le changement de la forme des brins d'ADN au besoin était effectué manuellement, ce qui limitait considérablement les possibilités d'une telle procédure. Le même algorithme vous permet de créer automatiquement des pièces d'ADN concepteur, ce qui vous permet de les utiliser pour la formation de nanostructures bidimensionnelles et tridimensionnelles. De plus, cet algorithme est accessible à tous. Un rapport de l'équipe de recherche nous aidera à déterminer ce qui fonctionne et comment. Allons-y.
Base d'étudeL'utilisation de l'ADN comme matériau de construction dans les nanostructures d'un large éventail d'applications est devenue ces dernières années l'objectif de nombreuses études et expériences. Beaucoup d'entre eux ont obtenu d'excellents résultats, mais comme nous le savons, il n'y a pas de limite à la perfection. À titre d'exemple, les chercheurs citent des structures d'ADN monodispersées à une échelle mégadaltique avec un coefficient de coagulation élevé, qui a été réalisé en utilisant de longues trames d'ADNsb (ADN simple brin) - ADN simple brin / simple brin. Cependant, dans ce cas, c'est la longueur des brins d'ADN qui est la principale limitation de la taille d'un seul ADN origami. Cette limitation peut être contournée par la formation d'une structure composée de plusieurs origamis à la fois.
Nous savons également déjà (
Tic-tac-toe: une démonstration du processus contrôlé de reconfiguration des structures d'ADN ) que certaines structures d'ADN peuvent être créées à l'aide de tuiles d'ADN, ce qui vous permet de former une géométrie complexe de cette structure même.
Mais encore une fois, tout se résume au fait que le processus est effectué manuellement, ce qui est long et morne. Qu'est-ce que les scientifiques aiment? C'est vrai, l'automatisation, ou au moins une automatisation partielle. Des outils similaires sont déjà disponibles (un exemple de scientifiques - caDNAno ou Tiamat), mais ces programmes ont leurs inconvénients. Tout d'abord, la création manuelle du cadre. Deuxièmement, les limites des algorithmes ci-dessus dans la question de la systématisation de la conception de la géométrie de la structure.
En un mot, dans cet article, les chercheurs se sont fixé pour objectif de créer un algorithme qui fera presque tout seul. Ainsi, toute personne ne peut définir que les paramètres de l'enveloppe extérieure de la structure souhaitée, et le programme remplira la partie intérieure indépendamment avec tous les fils d'ADN nécessaires, les agrafes, les séquences et plus encore. Vous pouvez également exécuter le processus dans l'ordre inverse - définissez tous les "intérieurs", pour ainsi dire, la grille de structure, et l'algorithme construira le cadre correct et le plus optimal autour de lui.
Ce programme open source (PERDIX) est à la disposition de tout le monde, grâce aux chercheurs. Ainsi, n'importe qui peut non seulement "jouer" avec le programme lui-même, mais aussi, éventuellement, apporter des modifications améliorées à son code. Où le télécharger et comment le démarrer, nous considérerons à la fin de l'article.
Et maintenant, regardons des exemples d'application de ce programme que l'équipe de recherche nous a fournis.
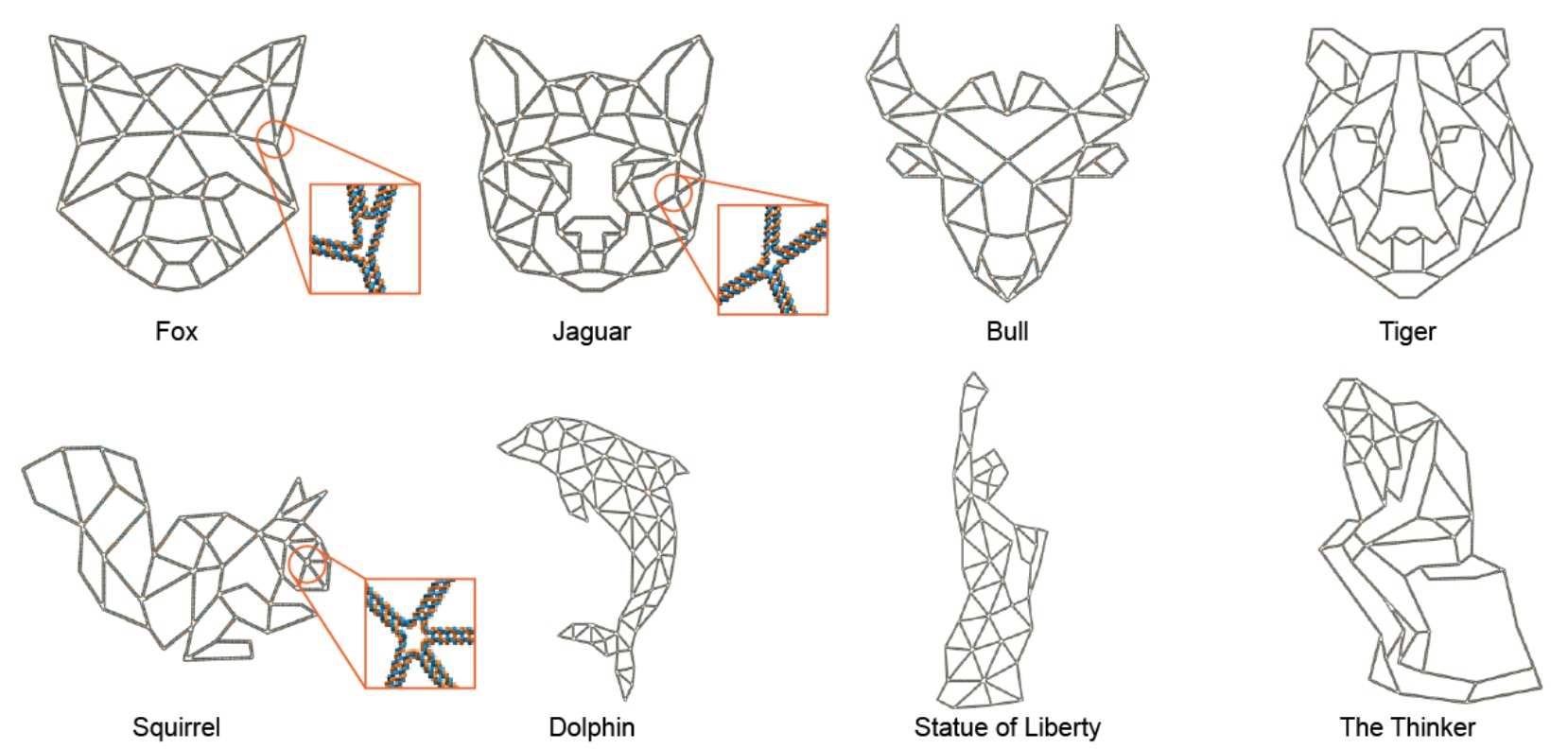
Résultats de la validation PERDIX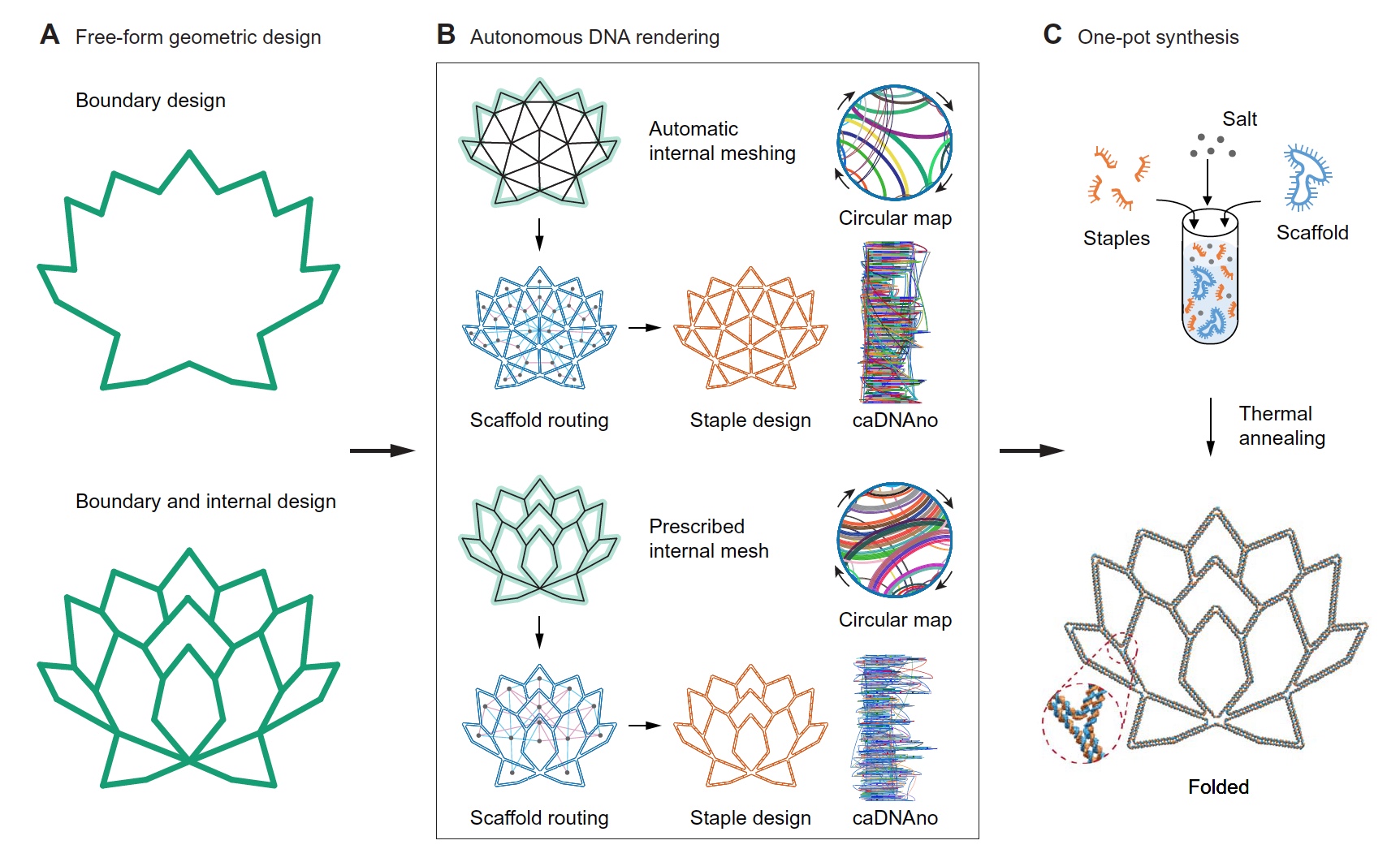 Image n ° 1
Image n ° 1En tant que données d'entrée, soit l'image de la bordure de la structure souhaitée (
1A , ci-dessus) ou une image avec des détails géométriques plus précis (
1A , ci-dessous) est utilisée. De plus, le programme lui-même remplit l'espace interne.
Lorsque seul le «contour» est spécifié, la géométrie interne de la grille de la structure est formée de triangles utilisant le programme open-source DistMesh, qui nécessite l'introduction de la densité de grille uniquement comme paramètre nécessaire.
Lorsque la forme et les lignes géométriques sont spécifiées (
1A , en bas), Shapely est utilisé - un package Python pour manipuler et analyser des objets géométriques plats. Shapely génère un maillage polygonal, dans lequel ses lignes et leurs points d'intersection forment la géométrie nécessaire du cadre.
En outre, après avoir déterminé la forme souhaitée, il était nécessaire de définir la longueur nominale de la nervure, qui devrait être ≥ 38 pb (bases appariées) ou 12,58 nm. Ainsi, au moins deux croisements d'appariement par bord seront certainement présents dans la structure requise.
Ensuite, toutes les lignes cibles de la future structure sont transformées en arêtes DX, dans lesquelles tous les sommets sont des points de connexion multi-grille (
1B ). Suivant les principes de la théorie des graphes, la structure squelettique optimale est automatiquement formée, après quoi le processus d'attribution d'une séquence d'agrafes d'ADN supplémentaires est terminé. Le processus d'édition manuelle des données de sortie (résultats) est effectué au moyen du fichier caDNAno généré.
Une caractéristique très importante de leur système, les scientifiques appellent le fait que les côtes n'ont pas à égaler un nombre entier d'hélices à double ADN-B (10,5 pb). Et cela vous permet d'obtenir plus de liberté dans la conception de la géométrie de la structure.
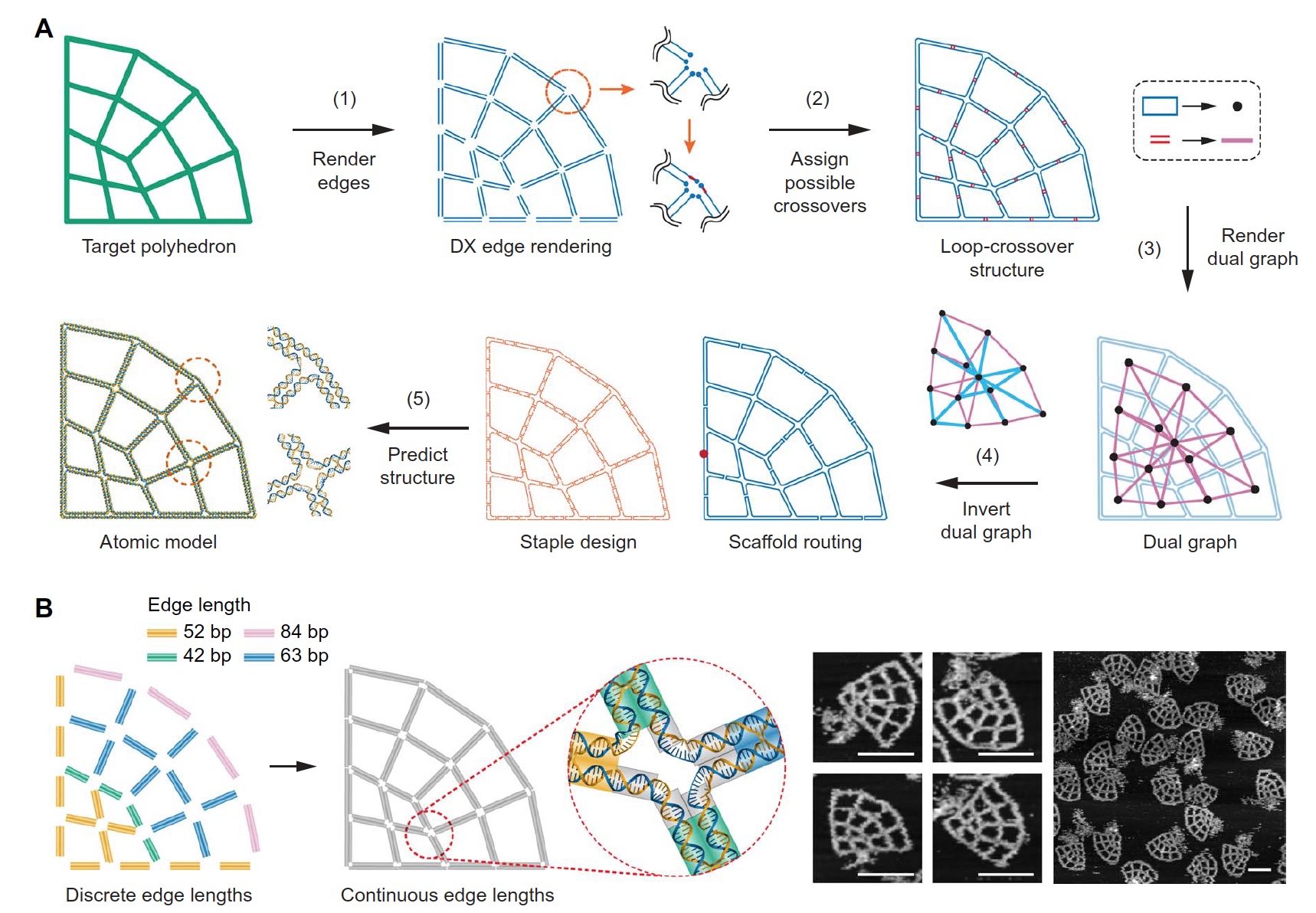 Image n ° 2
Image n ° 2Afin d'effectuer rapidement et avec précision la partie automatique du processus de formation de la géométrie de la structure, tous les bords doivent d'abord être convertis en motifs DX en rendant chaque bord de la géométrie cible à l'aide de deux lignes de cadre antiparallèles (
2A ). Ensuite, ils sont combinés en un seul sommet, ce qui fait que chaque bord fait partie d'une boucle. Et cela conduit à son tour à la formation d'une seule grande boucle externe, constituée de petites boucles. L'étape suivante consiste à trouver toutes les options de croisement possibles entre les boucles adjacentes, c'est-à-dire la formation d'une structure d'intersection de boucles.
La troisième étape de ce processus consiste à représenter toutes les structures résultantes comme un ensemble de nœuds et tous les croisements comme des bords de la future structure.
Ensuite, l'arbre couvrant le double graphe est calculé, après quoi il est converti en un framework ssDNA. Ce processus est entièrement automatique. Et enfin, un modèle structurel tridimensionnel est généré au niveau atomique.
La figure
2B montre deux options pour la formation de la géométrie requise de la structure: avec une longueur d'arête discrète ou avec une longueur d'arête continue. La deuxième option vous permet de créer une structure avec une longueur d'arête arbitraire, ce qui permet à son tour d'obtenir la plus grande coagulation. Ces variations de la structuration géométrique ont été vérifiées à l'aide d'un microscope à force atomique à balayage (images à droite sur l'image
2B ).
 Image n ° 3
Image n ° 3Les chercheurs se sont également fixé pour objectif d'examiner plus en détail des aspects de la géométrie cible tels que la rigidité mécanique, la précision de la forme, la taille de la structure, la taille du maillage interne et les types de ses éléments. Il était également nécessaire de comprendre l'importance des N-branches, c'est-à-dire le nombre de connexions entre les bords et, par conséquent, la complexité globale de la structure. L'analyse des données AFM a montré que les structures bidimensionnelles avec un grand nombre de branches et une longueur de bord plus longue (en raison de nucléotides non appariés) sont obtenues assez bien formées, et l'hétérogénéité des particules dans leur structure est minime.
La grille interne de la structure avait trois options: triangles avec la même direction, carrés et triangles avec des directions différentes (
3C ). C'est le modèle à triangles qui a montré la plus grande précision dans la formation de la géométrie nécessaire, contrairement au modèle à carrés. La présence de directions mixtes dans le modèle triangulaire, pour ainsi dire, a permis d'obtenir des branches N plus symétriques et une forme plus précise (moins déformée).
Les scientifiques notent que l'analyse du maillage interne de la structure simulée est extrêmement importante pour comprendre la rigidité mécanique de cette structure. Et c'est précisément le modèle décrit ci-dessus qui est idéal pour créer des structures suffisamment solides, précises (en termes de respect des paramètres spécifiés) et flexibles.
 Image n ° 4
Image n ° 4Bien sûr, les scientifiques ne sont pas sans sens de la beauté, pour ainsi dire. Par conséquent, ils ont décidé de démontrer les possibilités de leur développement en concevant des structures de 15 formes différentes, plus précisément avec différentes grilles et cadres internes. Nous pouvons voir les options dans l'image ci-dessus: voici les carrés «habituels», et un quart de cercle, et même un lotus.
Logiciels libresComme je l'ai mentionné précédemment, le travail des chercheurs est ouvert à tous sur le lien (), où vous pouvez trouver les éléments de programme nécessaires (MATLAB, Python 2.7 et Shapely 1.6.4).
Quant à l'algorithme de formation de la géométrie des structures ADN-origami, les instructions de téléchargement et d'exécution sont présentées ci-dessous au format vidéo. Je cacherai toutes les vidéos sous le spoiler afin de ne pas étirer l'article.
Vidéo # 1: lancement de PERDIXLe lien pour télécharger les fichiers nécessaires qui était dans la vidéo ne fonctionne pas maintenant. Voici une alternative -
lien de téléchargement .
Vidéo n ° 2: conception du cadre (périmètre) d'une structure dans PERDIX
Vidéo # 3: conception du cadre et de la structure interne dans PERDIX
Vidéo n ° 4: modèles atomiques (différentes grilles)
Vidéo n ° 5: modèles atomiques (branches N)
Vidéo n ° 6: modèles atomiques (modèle L)
Vidéo n ° 7: modèles atomiques ("branche" courbe)
Pour une connaissance plus détaillée de cette étude et de ses résultats, je vous recommande fortement de consulter le
rapport du groupe de recherche et
les documents complémentaires .
ÉpilogueCette étude visait à simplifier un processus plutôt long et long. Et ça a marché. L'algorithme PERDIX vous permet de créer des structures avec des géométries très différentes, tout en fixant les paramètres minimum nécessaires. L'ADN a longtemps été un objet d'étude pour les scientifiques, non seulement en tant que partie intégrante de toute vie et vecteur d'informations, mais également en tant qu'option possible pour la fondation des technologies futures. De tels travaux permettent de comprendre plus en détail (et clairement) l'étendue des possibilités des nanostructures créées à partir de l'origami ADN.
Le deuxième moment agréable de cette étude, du moins pour moi, est que l'algorithme est accessible à tous. Tout le monde peut l'utiliser, tout le monde peut l'améliorer. En donnant un accès complet à la fois à leur rapport et au logiciel lui-même, les scientifiques contribuent non seulement à la vulgarisation de leur domaine scientifique, mais aussi à la science en tant que telle.
J'ai rencontré des articles dans lesquels les scientifiques critiquaient assez le système d'accès payant aux rapports, arguant que cela crée une barrière supplémentaire entre les connaissances et les gens ordinaires. La situation dans ce domaine est très ambiguë et controversée, car tout le monde devrait gagner de l'argent (publications scientifiques, groupes de recherche et sites scientifiques), mais le coût élevé d'accès à certaines études les rend inaccessibles aux amateurs de sciences ordinaires, aux étudiants et même à certains professeurs et figures scientifiques. Comme je l'ai dit, la question est ambiguë, nous n'y reviendrons donc pas beaucoup. En tout cas, je suis heureux que nos héros d'aujourd'hui nous aient donné un accès complet à leur travail, qui mérite non seulement une attention, mais aussi une sincère admiration.
Merci de votre attention, restez curieux et bonne semaine de travail, les gars.
Merci de rester avec nous. Aimez-vous nos articles? Vous voulez voir des matériaux plus intéressants? Soutenez-nous en passant une commande ou en le recommandant à vos amis, une
réduction de 30% pour les utilisateurs Habr sur un analogue unique de serveurs d'entrée de gamme que nous avons inventés pour vous: Toute la vérité sur VPS (KVM) E5-2650 v4 (6 cœurs) 10 Go DDR4 240 Go SSD 1 Gbps à partir de 20 $ ou comment diviser le serveur? (les options sont disponibles avec RAID1 et RAID10, jusqu'à 24 cœurs et jusqu'à 40 Go de DDR4).
VPS (KVM) E5-2650 v4 (6 cœurs) 10 Go DDR4 240 Go SSD 1 Gbit / s jusqu'au printemps gratuitement lors du paiement pendant
six mois, vous pouvez commander
ici .
Dell R730xd 2 fois moins cher? Nous avons seulement
2 x Intel Dodeca-Core Xeon E5-2650v4 128 Go DDR4 6x480 Go SSD 1 Gbps 100 TV à partir de 249 $ aux Pays-Bas et aux États-Unis! Pour en savoir plus sur la
création d'un bâtiment d'infrastructure. classe utilisant des serveurs Dell R730xd E5-2650 v4 coûtant 9 000 euros pour un sou?