Équations différentielles ordinaires neuronales
Une proportion importante de processus est décrite par des équations différentielles, cela peut être l’évolution du système physique dans le temps, l’état de santé du patient, les caractéristiques fondamentales de la bourse, etc. Les données sur ces processus sont de nature cohérente et continue, en ce sens que les observations ne sont que des manifestations d'une sorte d'état en constante évolution.
Il existe également un autre type de données série, ce sont les données discrètes, par exemple les données de tâche PNL. L'état de ces données varie discrètement: d'un caractère ou d'un mot à l'autre.
Désormais, les deux types de ces données série sont généralement traités par des réseaux récursifs, bien qu'ils soient de nature différente et semblent nécessiter des approches différentes.
Un article très intéressant a été présenté lors de la dernière
conférence NIPS , qui peut aider à résoudre ce problème. Les auteurs proposent une approche qu'ils ont appelée
Neural ODEs .
Ici, j'ai essayé de reproduire et de résumer les résultats de cet article afin de faciliter un peu la connaissance de son idée. Il me semble que cette nouvelle architecture pourrait bien trouver sa place dans les outils standards d'un data scientist aux côtés de réseaux convolutionnels et récurrents.
Figure 1: La rétropropagation à gradient continu nécessite de résoudre l'équation différentielle augmentée dans le temps.
Les flèches représentent l'ajustement des gradients propagés vers l'arrière par les gradients à partir des observations.
Illustration de l'article original.
Énoncé du problème
Qu'il y ait un processus qui obéit à une ODE inconnue et qu'il y ait plusieurs observations (bruyantes) le long de la trajectoire du processus
Comment trouver une approximation

fonctions de haut-parleur

?
Considérons d'abord une tâche plus simple: il n'y a que 2 observations, au début et à la fin de la trajectoire,

.
L'évolution du système commence à partir de l'état

à l'heure

avec une fonction de dynamique paramétrée utilisant n'importe quelle méthode d'évolution des systèmes ODE. Une fois que le système est dans un nouvel état

, il est comparé à l'état

et la différence entre eux est minimisée en faisant varier les paramètres

fonctions dynamiques.
Ou, plus formellement, envisagez de minimiser la fonction de perte

:
Pour minimiser

, vous devez calculer les gradients pour tous ses paramètres:

. Pour ce faire, vous devez d'abord déterminer comment

dépend de l'état à chaque instant

:

est appelé état
adjoint , sa dynamique est donnée par une autre équation différentielle, qui peut être considérée comme un analogue continu de la différenciation d'une fonction complexe (
règle de chaîne ):
Le résultat de cette formule se trouve en annexe de l'article d'origine.
Les vecteurs de cet article doivent être considérés comme des vecteurs en minuscules, bien que l'article d'origine utilise à la fois une représentation en ligne et en colonne.En résolvant diffur (4) dans le temps, on obtient une dépendance de l'état initial

:
Pour calculer le gradient par rapport à

et

, vous pouvez simplement les considérer comme faisant partie de l'État. Cette condition est appelée
augmentée . La dynamique de cet état est obtenue trivialement à partir de la dynamique d'origine:
Puis l'état conjugué à cet état augmenté:
Dynamique augmentée par gradient:
L'équation différentielle de l'état augmenté conjugué de la formule (4) alors:
Résoudre cette ODE dans le temps donne:
Avec quoi
donne des dégradés dans tous les paramètres d'entrée au
solveur ODESolve ODE.
Tous les gradients (10), (11), (12), (13) peuvent être calculés ensemble dans un appel
ODESolve avec la dynamique de l'état augmenté conjugué (9).
 Illustration de l'article original.
Illustration de l'article original.L'algorithme ci-dessus décrit la propagation inverse du gradient de la solution ODE pour des observations successives.
Dans le cas de plusieurs observations sur une même trajectoire, tout est calculé de la même manière, mais aux moments des observations, l'inverse du gradient propagé doit être ajusté avec des gradients de l'observation courante, comme le montre la
figure 1 .
Implémentation
Le code ci-dessous est mon implémentation des
ODE neuronaux . Je l'ai fait uniquement pour mieux comprendre ce qui se passe. Cependant, il est très proche de ce qui est implémenté dans le
référentiel des auteurs de l'article. Il contient tout le code que vous devez comprendre en un seul endroit, il est également légèrement plus commenté. Pour des applications et expérimentations réelles, il est toujours préférable d'utiliser l'implémentation des auteurs de l'article original.
import math import numpy as np from IPython.display import clear_output from tqdm import tqdm_notebook as tqdm import matplotlib as mpl import matplotlib.pyplot as plt %matplotlib inline import seaborn as sns sns.color_palette("bright") import matplotlib as mpl import matplotlib.cm as cm import torch from torch import Tensor from torch import nn from torch.nn import functional as F from torch.autograd import Variable use_cuda = torch.cuda.is_available()
Vous devez d'abord implémenter n'importe quelle méthode pour l'évolution des systèmes ODE. Par souci de simplicité, la méthode Euler est implémentée ici, bien que toute méthode explicite ou implicite convienne.
def ode_solve(z0, t0, t1, f): """ - """ h_max = 0.05 n_steps = math.ceil((abs(t1 - t0)/h_max).max().item()) h = (t1 - t0)/n_steps t = t0 z = z0 for i_step in range(n_steps): z = z + h * f(z, t) t = t + h return z
Il décrit également la superclasse d'une fonction de dynamique paramétrée avec quelques méthodes utiles.
Premièrement: vous devez renvoyer tous les paramètres dont dépend la fonction sous la forme d'un vecteur.
Deuxièmement: il est nécessaire de calculer la dynamique augmentée. Cette dynamique dépend du gradient de la fonction paramétrée en termes de paramètres et de données d'entrée. Afin de ne pas avoir à enregistrer le gradient avec chaque main pour chaque nouvelle architecture, nous utiliserons la méthode
torch.autograd.grad .
class ODEF(nn.Module): def forward_with_grad(self, z, t, grad_outputs): """Compute f and a df/dz, a df/dp, a df/dt""" batch_size = z.shape[0] out = self.forward(z, t) a = grad_outputs adfdz, adfdt, *adfdp = torch.autograd.grad( (out,), (z, t) + tuple(self.parameters()), grad_outputs=(a), allow_unused=True, retain_graph=True )
Le code ci-dessous décrit la propagation avant et arrière pour
les ODE neuronaux . Il est nécessaire de séparer ce code du
module torch.nn.Module principal sous la forme de la fonction
torch.autograd.Function car dans ce dernier, vous pouvez implémenter une méthode de
rétropropagation arbitraire, par opposition à un module. Ce n'est donc qu'une béquille.
Cette fonctionnalité sous-tend toute l'approche
Neural ODE .
class ODEAdjoint(torch.autograd.Function): @staticmethod def forward(ctx, z0, t, flat_parameters, func): assert isinstance(func, ODEF) bs, *z_shape = z0.size() time_len = t.size(0) with torch.no_grad(): z = torch.zeros(time_len, bs, *z_shape).to(z0) z[0] = z0 for i_t in range(time_len - 1): z0 = ode_solve(z0, t[i_t], t[i_t+1], func) z[i_t+1] = z0 ctx.func = func ctx.save_for_backward(t, z.clone(), flat_parameters) return z @staticmethod def backward(ctx, dLdz): """ dLdz shape: time_len, batch_size, *z_shape """ func = ctx.func t, z, flat_parameters = ctx.saved_tensors time_len, bs, *z_shape = z.size() n_dim = np.prod(z_shape) n_params = flat_parameters.size(0)
Maintenant, pour plus de commodité,
encapsulez cette fonction dans
nn.Module .
class NeuralODE(nn.Module): def __init__(self, func): super(NeuralODE, self).__init__() assert isinstance(func, ODEF) self.func = func def forward(self, z0, t=Tensor([0., 1.]), return_whole_sequence=False): t = t.to(z0) z = ODEAdjoint.apply(z0, t, self.func.flatten_parameters(), self.func) if return_whole_sequence: return z else: return z[-1]
Candidature
Récupération de la fonction dynamique réelle (vérification d'approche)
À titre de test de base, vérifions maintenant si
Neural ODE peut restaurer la véritable fonction de la dynamique à l'aide de données d'observation.
Pour ce faire, nous déterminons d'abord la fonction dynamique de l'ODE, faisons évoluer la trajectoire en fonction de celle-ci, puis essayons de la restaurer à partir de la fonction dynamique paramétrée au hasard.
Tout d'abord, examinons le cas le plus simple d'une ODE linéaire. La fonction dynamique n'est que l'action d'une matrice.
La fonction entraînée est paramétrée par une matrice aléatoire.
De plus, une dynamique un peu plus sophistiquée (sans gif, car le processus d'apprentissage n'est pas si beau :))
La fonction d'apprentissage ici est un réseau entièrement connecté avec une couche cachée.
Code class LinearODEF(ODEF): def __init__(self, W): super(LinearODEF, self).__init__() self.lin = nn.Linear(2, 2, bias=False) self.lin.weight = nn.Parameter(W) def forward(self, x, t): return self.lin(x)
La fonction dynamique n'est qu'une matrice
class SpiralFunctionExample(LinearODEF): def __init__(self): matrix = Tensor([[-0.1, -1.], [1., -0.1]]) super(SpiralFunctionExample, self).__init__(matrix)
Matrice paramétrée au hasard
class RandomLinearODEF(LinearODEF): def __init__(self): super(RandomLinearODEF, self).__init__(torch.randn(2, 2)/2.)
Dynamique pour des trajectoires plus sophistiquées
class TestODEF(ODEF): def __init__(self, A, B, x0): super(TestODEF, self).__init__() self.A = nn.Linear(2, 2, bias=False) self.A.weight = nn.Parameter(A) self.B = nn.Linear(2, 2, bias=False) self.B.weight = nn.Parameter(B) self.x0 = nn.Parameter(x0) def forward(self, x, t): xTx0 = torch.sum(x*self.x0, dim=1) dxdt = torch.sigmoid(xTx0) * self.A(x - self.x0) + torch.sigmoid(-xTx0) * self.B(x + self.x0) return dxdt
Dynamique d'apprentissage sous la forme d'un réseau entièrement connecté
class NNODEF(ODEF): def __init__(self, in_dim, hid_dim, time_invariant=False): super(NNODEF, self).__init__() self.time_invariant = time_invariant if time_invariant: self.lin1 = nn.Linear(in_dim, hid_dim) else: self.lin1 = nn.Linear(in_dim+1, hid_dim) self.lin2 = nn.Linear(hid_dim, hid_dim) self.lin3 = nn.Linear(hid_dim, in_dim) self.elu = nn.ELU(inplace=True) def forward(self, x, t): if not self.time_invariant: x = torch.cat((x, t), dim=-1) h = self.elu(self.lin1(x)) h = self.elu(self.lin2(h)) out = self.lin3(h) return out def to_np(x): return x.detach().cpu().numpy() def plot_trajectories(obs=None, times=None, trajs=None, save=None, figsize=(16, 8)): plt.figure(figsize=figsize) if obs is not None: if times is None: times = [None] * len(obs) for o, t in zip(obs, times): o, t = to_np(o), to_np(t) for b_i in range(o.shape[1]): plt.scatter(o[:, b_i, 0], o[:, b_i, 1], c=t[:, b_i, 0], cmap=cm.plasma) if trajs is not None: for z in trajs: z = to_np(z) plt.plot(z[:, 0, 0], z[:, 0, 1], lw=1.5) if save is not None: plt.savefig(save) plt.show() def conduct_experiment(ode_true, ode_trained, n_steps, name, plot_freq=10):
Comme vous pouvez le voir,
Neural ODE fait un très bon travail de restauration de la dynamique. Autrement dit, le concept dans son ensemble fonctionne.
Maintenant, vérifiez un problème légèrement plus compliqué (MNIST, haha).
Neural ODE inspiré par ResNets
Dans ResNet'ax, l'état latent change selon la formule
où

Est le numéro de bloc et

c'est une fonction apprise par les couches à l'intérieur du bloc.
À la limite, si nous prenons un nombre infini de blocs avec des pas toujours plus petits, nous obtenons la dynamique continue de la couche cachée sous la forme d'un ODE, tout comme ce qui était ci-dessus.
À partir de la couche d'entrée

nous pouvons définir la couche de sortie

comme solution à cette ODE au temps T.
Maintenant on peut compter

en tant que paramètres distribués (
partagés ) entre tous les blocs infinitésimaux.
Validation de l'architecture Neural ODE sur MNIST
Dans cette partie, nous allons tester la capacité de
Neural ODE à être utilisé comme composants dans des architectures plus familières.
En particulier, nous remplacerons les blocs résiduels par
Neural ODE dans le classificateur MNIST.
Code def norm(dim): return nn.BatchNorm2d(dim) def conv3x3(in_feats, out_feats, stride=1): return nn.Conv2d(in_feats, out_feats, kernel_size=3, stride=stride, padding=1, bias=False) def add_time(in_tensor, t): bs, c, w, h = in_tensor.shape return torch.cat((in_tensor, t.expand(bs, 1, w, h)), dim=1) class ConvODEF(ODEF): def __init__(self, dim): super(ConvODEF, self).__init__() self.conv1 = conv3x3(dim + 1, dim) self.norm1 = norm(dim) self.conv2 = conv3x3(dim + 1, dim) self.norm2 = norm(dim) def forward(self, x, t): xt = add_time(x, t) h = self.norm1(torch.relu(self.conv1(xt))) ht = add_time(h, t) dxdt = self.norm2(torch.relu(self.conv2(ht))) return dxdt class ContinuousNeuralMNISTClassifier(nn.Module): def __init__(self, ode): super(ContinuousNeuralMNISTClassifier, self).__init__() self.downsampling = nn.Sequential( nn.Conv2d(1, 64, 3, 1), norm(64), nn.ReLU(inplace=True), nn.Conv2d(64, 64, 4, 2, 1), norm(64), nn.ReLU(inplace=True), nn.Conv2d(64, 64, 4, 2, 1), ) self.feature = ode self.norm = norm(64) self.avg_pool = nn.AdaptiveAvgPool2d((1, 1)) self.fc = nn.Linear(64, 10) def forward(self, x): x = self.downsampling(x) x = self.feature(x) x = self.norm(x) x = self.avg_pool(x) shape = torch.prod(torch.tensor(x.shape[1:])).item() x = x.view(-1, shape) out = self.fc(x) return out func = ConvODEF(64) ode = NeuralODE(func) model = ContinuousNeuralMNISTClassifier(ode) if use_cuda: model = model.cuda() import torchvision img_std = 0.3081 img_mean = 0.1307 batch_size = 32 train_loader = torch.utils.data.DataLoader( torchvision.datasets.MNIST("data/mnist", train=True, download=True, transform=torchvision.transforms.Compose([ torchvision.transforms.ToTensor(), torchvision.transforms.Normalize((img_mean,), (img_std,)) ]) ), batch_size=batch_size, shuffle=True ) test_loader = torch.utils.data.DataLoader( torchvision.datasets.MNIST("data/mnist", train=False, download=True, transform=torchvision.transforms.Compose([ torchvision.transforms.ToTensor(), torchvision.transforms.Normalize((img_mean,), (img_std,)) ]) ), batch_size=128, shuffle=True ) optimizer = torch.optim.Adam(model.parameters()) def train(epoch): num_items = 0 train_losses = [] model.train() criterion = nn.CrossEntropyLoss() print(f"Training Epoch {epoch}...") for batch_idx, (data, target) in tqdm(enumerate(train_loader), total=len(train_loader)): if use_cuda: data = data.cuda() target = target.cuda() optimizer.zero_grad() output = model(data) loss = criterion(output, target) loss.backward() optimizer.step() train_losses += [loss.item()] num_items += data.shape[0] print('Train loss: {:.5f}'.format(np.mean(train_losses))) return train_losses def test(): accuracy = 0.0 num_items = 0 model.eval() criterion = nn.CrossEntropyLoss() print(f"Testing...") with torch.no_grad(): for batch_idx, (data, target) in tqdm(enumerate(test_loader), total=len(test_loader)): if use_cuda: data = data.cuda() target = target.cuda() output = model(data) accuracy += torch.sum(torch.argmax(output, dim=1) == target).item() num_items += data.shape[0] accuracy = accuracy * 100 / num_items print("Test Accuracy: {:.3f}%".format(accuracy)) n_epochs = 5 test() train_losses = [] for epoch in range(1, n_epochs + 1): train_losses += train(epoch) test() import pandas as pd plt.figure(figsize=(9, 5)) history = pd.DataFrame({"loss": train_losses}) history["cum_data"] = history.index * batch_size history["smooth_loss"] = history.loss.ewm(halflife=10).mean() history.plot(x="cum_data", y="smooth_loss", figsize=(12, 5), title="train error")
Testing... 100% 79/79 [00:01<00:00, 45.69it/s] Test Accuracy: 9.740% Training Epoch 1... 100% 1875/1875 [01:15<00:00, 24.69it/s] Train loss: 0.20137 Testing... 100% 79/79 [00:01<00:00, 46.64it/s] Test Accuracy: 98.680% Training Epoch 2... 100% 1875/1875 [01:17<00:00, 24.32it/s] Train loss: 0.05059 Testing... 100% 79/79 [00:01<00:00, 46.11it/s] Test Accuracy: 97.760% Training Epoch 3... 100% 1875/1875 [01:16<00:00, 24.63it/s] Train loss: 0.03808 Testing... 100% 79/79 [00:01<00:00, 45.65it/s] Test Accuracy: 99.000% Training Epoch 4... 100% 1875/1875 [01:17<00:00, 24.28it/s] Train loss: 0.02894 Testing... 100% 79/79 [00:01<00:00, 45.42it/s] Test Accuracy: 99.130% Training Epoch 5... 100% 1875/1875 [01:16<00:00, 24.67it/s] Train loss: 0.02424 Testing... 100% 79/79 [00:01<00:00, 45.89it/s] Test Accuracy: 99.170%
Après un entraînement très rude en seulement 5 époques et 6 minutes d'entraînement, le modèle a déjà atteint une erreur de test de moins de 1%. Nous pouvons dire que
les ODE neuronaux s'intègrent bien en
tant que composant dans des réseaux plus traditionnels.
Dans leur article, les auteurs comparent également ce classificateur (ODE-Net) avec un réseau régulier entièrement connecté, avec ResNet avec une architecture similaire, et avec exactement la même architecture, dans laquelle le gradient se propage directement via les opérations dans
ODESolve (sans la méthode du gradient conjugué) ( RK-Net).
Illustration de l'article original.Selon eux, un réseau à une couche entièrement connecté avec environ le même nombre de paramètres que
Neural ODE a une erreur beaucoup plus élevée sur le test, ResNet avec à peu près la même erreur a beaucoup plus de paramètres et RK-Net sans la méthode du gradient conjugué a une erreur légèrement plus élevée et avec une consommation de mémoire augmentant linéairement (plus l'erreur tolérée est petite, plus
ODESolve doit faire de pas, ce qui augmente linéairement la consommation de mémoire avec le nombre de pas).
Les auteurs utilisent la méthode implicite Runge-Kutta avec une taille de pas adaptative dans leur implémentation, contrairement à la méthode Euler plus simple ici. Ils étudient également certaines propriétés de la nouvelle architecture.
Fonction ODE-Net (NFE Forward - le nombre de calculs de fonction dans un passage direct)
Illustration de l'article original.- (a) La modification du niveau acceptable d'erreur numérique modifie le nombre d'étapes de la distribution directe.
- (b) Le temps consacré à la distribution directe est proportionnel au nombre de calculs de la fonction.
- (c) Le nombre de calculs de la fonction de rétropropagation représente environ la moitié de la propagation directe, ce qui indique que la méthode du gradient conjugué peut être plus efficace sur le plan du calcul que la propagation du gradient directement via ODESolve .
- (d) Comme ODE-Net devient de plus en plus entraîné, il nécessite de plus en plus de calculs d'une fonction (une étape toujours plus petite), s'adaptant éventuellement à la complexité croissante du modèle.
Fonction générative cachée pour la modélisation de séries chronologiques
Neural ODE convient au traitement de données en série continues même lorsque le chemin se trouve dans un espace caché inconnu.
Dans cette section, nous allons expérimenter
et modifier la génération de séquences continues à l'aide de
Neural ODE , et jeter un œil à l'espace caché appris.
Les auteurs comparent également cela avec des séquences similaires générées par des réseaux récurrents.
L'expérience ici est légèrement différente de l'exemple correspondant dans le référentiel des auteurs, ici il y a un ensemble plus diversifié de chemins.
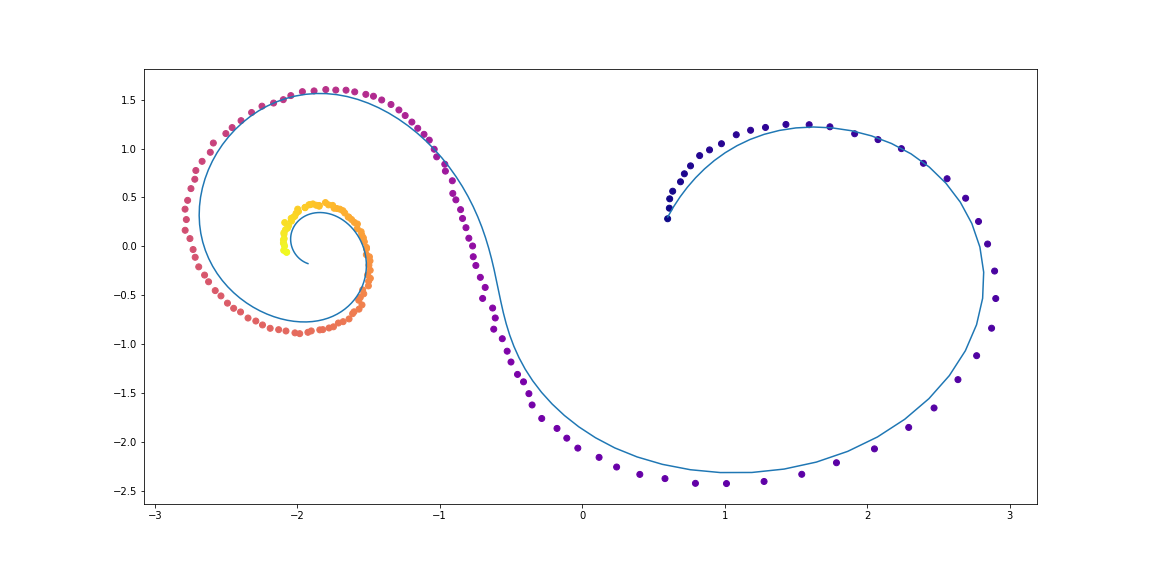
Les données
Les données d'entraînement sont constituées de spirales aléatoires, dont la moitié dans le sens horaire et la seconde dans le sens antihoraire. Des sous-séquences aléatoires sont ensuite échantillonnées à partir de ces spirales, traitées par le modèle récursif de codage dans la direction opposée, générant un état caché de départ, qui évolue ensuite, créant une trajectoire dans l'espace caché. Ce chemin latent est ensuite mappé à l'espace de données et comparé à la sous-séquence échantillonnée. Ainsi, le modèle apprend à générer des trajectoires similaires à un ensemble de données.
Exemples de spirales d'ensembles de donnéesLa VAE comme modèle génératif
Modèle génératif par une procédure d'échantillonnage:
Qui peut être formé en utilisant l'approche auto-encodeur variationnel.
- Parcourez un encodeur récurrent à travers une séquence temporelle dans le temps pour obtenir les paramètres
 ,
,  distribution variationnelle postérieure, puis en échantillonner:
distribution variationnelle postérieure, puis en échantillonner:
- Obtenez une trajectoire cachée:
- Mappez un chemin caché sur un chemin dans les données à l'aide d'un autre réseau de neurones:

- Maximisez l'évaluation de la limite inférieure de validité (ELBO) pour le chemin échantillonné:
Et dans le cas d'une distribution postérieure gaussienne

et niveau de bruit connu

:
Le graphique de calcul d'un modèle ODE masqué peut être représenté comme suit
Illustration de l'article original.Ce modèle peut ensuite être testé pour savoir comment il interpole le chemin en utilisant uniquement les observations initiales.
CodeDéfinir des modèles
class RNNEncoder(nn.Module): def __init__(self, input_dim, hidden_dim, latent_dim): super(RNNEncoder, self).__init__() self.input_dim = input_dim self.hidden_dim = hidden_dim self.latent_dim = latent_dim self.rnn = nn.GRU(input_dim+1, hidden_dim) self.hid2lat = nn.Linear(hidden_dim, 2*latent_dim) def forward(self, x, t):
Génération de jeux de données
t_max = 6.29*5 n_points = 200 noise_std = 0.02 num_spirals = 1000 index_np = np.arange(0, n_points, 1, dtype=np.int) index_np = np.hstack([index_np[:, None]]) times_np = np.linspace(0, t_max, num=n_points) times_np = np.hstack([times_np[:, None]] * num_spirals) times = torch.from_numpy(times_np[:, :, None]).to(torch.float32)
La formation
vae = ODEVAE(2, 64, 6) vae = vae.cuda() if use_cuda: vae = vae.cuda() optim = torch.optim.Adam(vae.parameters(), betas=(0.9, 0.999), lr=0.001) preload = False n_epochs = 20000 batch_size = 100 plot_traj_idx = 1 plot_traj = orig_trajs[:, plot_traj_idx:plot_traj_idx+1] plot_obs = samp_trajs[:, plot_traj_idx:plot_traj_idx+1] plot_ts = samp_ts[:, plot_traj_idx:plot_traj_idx+1] if use_cuda: plot_traj = plot_traj.cuda() plot_obs = plot_obs.cuda() plot_ts = plot_ts.cuda() if preload: vae.load_state_dict(torch.load("models/vae_spirals.sd")) for epoch_idx in range(n_epochs): losses = [] train_iter = gen_batch(batch_size) for x, t in train_iter: optim.zero_grad() if use_cuda: x, t = x.cuda(), t.cuda() max_len = np.random.choice([30, 50, 100]) permutation = np.random.permutation(t.shape[0]) np.random.shuffle(permutation) permutation = np.sort(permutation[:max_len]) x, t = x[permutation], t[permutation] x_p, z, z_mean, z_log_var = vae(x, t) z_var = torch.exp(z_log_var) kl_loss = -0.5 * torch.sum(1 + z_log_var - z_mean**2 - z_var, -1) loss = 0.5 * ((x-x_p)**2).sum(-1).sum(0) / noise_std**2 + kl_loss loss = torch.mean(loss) loss /= max_len loss.backward() optim.step() losses.append(loss.item()) print(f"Epoch {epoch_idx}") frm, to, to_seed = 0, 200, 50 seed_trajs = samp_trajs[frm:to_seed] ts = samp_ts[frm:to] if use_cuda: seed_trajs = seed_trajs.cuda() ts = ts.cuda() samp_trajs_p = to_np(vae.generate_with_seed(seed_trajs, ts)) fig, axes = plt.subplots(nrows=3, ncols=3, figsize=(15, 9)) axes = axes.flatten() for i, ax in enumerate(axes): ax.scatter(to_np(seed_trajs[:, i, 0]), to_np(seed_trajs[:, i, 1]), c=to_np(ts[frm:to_seed, i, 0]), cmap=cm.plasma) ax.plot(to_np(orig_trajs[frm:to, i, 0]), to_np(orig_trajs[frm:to, i, 1])) ax.plot(samp_trajs_p[:, i, 0], samp_trajs_p[:, i, 1]) plt.show() print(np.mean(losses), np.median(losses)) clear_output(wait=True) spiral_0_idx = 3 spiral_1_idx = 6 homotopy_p = Tensor(np.linspace(0., 1., 10)[:, None]) vae = vae if use_cuda: homotopy_p = homotopy_p.cuda() vae = vae.cuda() spiral_0 = orig_trajs[:, spiral_0_idx:spiral_0_idx+1, :] spiral_1 = orig_trajs[:, spiral_1_idx:spiral_1_idx+1, :] ts_0 = samp_ts[:, spiral_0_idx:spiral_0_idx+1, :] ts_1 = samp_ts[:, spiral_1_idx:spiral_1_idx+1, :] if use_cuda: spiral_0, ts_0 = spiral_0.cuda(), ts_0.cuda() spiral_1, ts_1 = spiral_1.cuda(), ts_1.cuda() z_cw, _ = vae.encoder(spiral_0, ts_0) z_cc, _ = vae.encoder(spiral_1, ts_1) homotopy_z = z_cw * (1 - homotopy_p) + z_cc * homotopy_p t = torch.from_numpy(np.linspace(0, 6*np.pi, 200)) t = t[:, None].expand(200, 10)[:, :, None].cuda() t = t.cuda() if use_cuda else t hom_gen_trajs = vae.decoder(homotopy_z, t) fig, axes = plt.subplots(nrows=2, ncols=5, figsize=(15, 5)) axes = axes.flatten() for i, ax in enumerate(axes): ax.plot(to_np(hom_gen_trajs[:, i, 0]), to_np(hom_gen_trajs[:, i, 1])) plt.show() torch.save(vae.state_dict(), "models/vae_spirals.sd")
Voilà ce qui se passe après une nuit d'entraînementLes points sont des observations bruyantes de la trajectoire d'origine (bleu), le
jaune sont des trajectoires reconstruites et interpolées, en utilisant des points comme entrées.
La couleur du point indique le temps.Les reconstitutions de certains exemples ne semblent pas trop bonnes. Peut-être que le modèle n'est pas assez complexe ou pas étudié assez longtemps. En tout cas, la reconstruction semble très raisonnable.Voyons maintenant ce qui se passe si nous interpolons une variable cachée dans le sens horaire vers un chemin anti-horloge.Les auteurs comparent également les reconstructions et les interpolations de chemin entre Neural ODE et un simple réseau récursif.Illustration de l'article original.Flux de normalisation continus
L'article original apporte également beaucoup au sujet de la normalisation des flux. Les flux de normalisation sont utilisés lorsque vous devez échantillonner à partir d'une distribution complexe qui apparaît à travers le changement de variables à partir d'une distribution simple (gaussienne, par exemple), et que vous connaissez toujours la densité de probabilité au point de chaque échantillon.Les auteurs montrent que l'utilisation de la substitution de variable continue est beaucoup plus efficace sur le plan informatique et interprétable que les méthodes précédentes.Les flux de normalisation sont très utiles dans des modèles tels que les codeurs automatiques de variation , les réseaux neuronaux bayésiens et d'autres de l'approche bayésienne.Ce sujet, cependant, sort du cadre de cettearticles, et ceux qui sont intéressés devraient lire l'article scientifique original.Pour les semences:Visualisation de la transformation du bruit (distribution simple) en données (distribution complexe) pour deux jeux de données;
L'axe des X montre la transformation de la densité et des échantillons au cours du «temps» (pour NS) et de la «profondeur» (pour NS).
Illustration de l'article originalMerci à bekemax pour son aide dans l'édition de la version anglaise du texte et pour ses commentaires physiques intéressants.Ceci conclut ma petite recherche sur les ODE neuronales . Merci de votre attention!Liens utiles