
Bonjour, Habr!
L'automne dernier, à Kaggle, un concours a été organisé pour le classement des images de reconnaissance de dessin rapide dessinées à la main, auquel a notamment participé une équipe de R-schiks composée d'
Artem Klevtsov ,
Philip Upravitelev et
Andrey Ogurtsov . Nous ne décrirons pas le concours en détail, cela a déjà été fait dans une
publication récente .
Cette fois, il n'y a pas eu de médailles pour les produits pharmaceutiques agricoles, mais beaucoup d'expérience précieuse a été acquise, alors je voudrais parler à la communauté d'un certain nombre de choses les plus intéressantes et utiles sur Kagl et dans le travail quotidien. Parmi les sujets traités: la vie dure sans
OpenCV , l'analyse des JSON (ces exemples
montrent l' intégration du code C ++ dans des scripts ou des packages dans R à l'aide de
Rcpp ), le paramétrage des scripts et la dockerisation de la solution finale. Tout le code du message sous une forme adaptée au lancement est disponible dans le
référentiel .
Contenu:
- Chargement efficace des données du CSV vers la base de données MonetDB
- Préparation des lots
- Itérateurs pour décharger des lots de la base de données
- Sélection de l'architecture du modèle
- Paramétrage du script
- Scripts d'ancrage
- Utilisation de plusieurs GPU dans Google Cloud
- Au lieu d'une conclusion
1. Chargement efficace des données du CSV vers la base de données MonetDB
Les données de ce concours ne sont pas fournies sous forme d'images prêtes à l'emploi, mais sous la forme de 340 fichiers CSV (un fichier pour chaque classe) contenant des JSON avec des coordonnées ponctuelles. En connectant ces points avec des lignes, nous obtenons l'image finale mesurant 256x256 pixels. De plus, pour chaque enregistrement, une étiquette indique si l'image a été correctement reconnue par le classificateur utilisé au moment de la collecte de l'ensemble de données, le code à deux lettres du pays de résidence de l'auteur, un identifiant unique, un horodatage et un nom de classe correspondant au nom du fichier. Une version simplifiée des données source pèse 7,4 Go dans l'archive et environ 20 Go après le déballage, les données complètes après le déballage prennent 240 Go. Les organisateurs ont garanti que les deux versions reproduisent les mêmes dessins, c'est-à-dire que la version complète est redondante. Dans tous les cas, le stockage de 50 millions d'images dans des fichiers graphiques ou dans des tableaux a été immédiatement considéré comme non rentable, et nous avons décidé de fusionner tous les fichiers CSV à partir de l'archive train_simplified.zip dans une base de données avec la génération ultérieure d'images de la bonne taille à la volée pour chaque lot .
Le MonetDB bien établi a été choisi comme SGBD, à savoir l'implémentation de R sous la forme du package MonetDBLite . Le package comprend une version intégrée du serveur de base de données et vous permet de soulever le serveur directement à partir de la session R et d'y travailler. La création d'une base de données et la connexion à celle-ci sont effectuées par une seule commande:
con <- DBI::dbConnect(drv = MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR"))
Nous devrons créer deux tableaux: un pour toutes les données, l'autre pour les informations de surcharge sur les fichiers téléchargés (utile en cas de problème et le processus devra être repris après le chargement de plusieurs fichiers):
Créer des tableaux if (!DBI::dbExistsTable(con, "doodles")) { DBI::dbCreateTable( con = con, name = "doodles", fields = c( "countrycode" = "char(2)", "drawing" = "text", "key_id" = "bigint", "recognized" = "bool", "timestamp" = "timestamp", "word" = "text" ) ) } if (!DBI::dbExistsTable(con, "upload_log")) { DBI::dbCreateTable( con = con, name = "upload_log", fields = c( "id" = "serial", "file_name" = "text UNIQUE", "uploaded" = "bool DEFAULT false" ) ) }
Le moyen le plus rapide de charger des données dans la base de données était de copier directement les fichiers CSV à l'aide de SQL - la commande COPY OFFSET 2 INTO tablename FROM path USING DELIMITERS ',','\\n','\"' NULL AS '' BEST EFFORT , où tablename est le nom de la table et du path est le chemin d'accès au fichier. Plus tard , une autre méthode a été trouvée pour augmenter la vitesse: remplacez simplement BEST EFFORT par LOCKED BEST EFFORT . Lorsque vous travaillez avec l'archive, il s'est avéré que l'implémentation de unzip intégrée dans R ne fonctionne pas correctement avec un certain nombre de fichiers de l'archive, nous avons donc utilisé unzip système (en utilisant le getOption("unzip") ).
Fonction d'écriture dans la base de données #' @title #' #' @description #' CSV- ZIP- #' #' @param con ( `MonetDBEmbeddedConnection`). #' @param tablename . #' @oaram zipfile ZIP-. #' @oaram filename ZIP-. #' @param preprocess , . #' `data` ( `data.table`). #' #' @return `TRUE`. #' upload_file <- function(con, tablename, zipfile, filename, preprocess = NULL) { # checkmate::assert_class(con, "MonetDBEmbeddedConnection") checkmate::assert_string(tablename) checkmate::assert_string(filename) checkmate::assert_true(DBI::dbExistsTable(con, tablename)) checkmate::assert_file_exists(zipfile, access = "r", extension = "zip") checkmate::assert_function(preprocess, args = c("data"), null.ok = TRUE) # path <- file.path(tempdir(), filename) unzip(zipfile, files = filename, exdir = tempdir(), junkpaths = TRUE, unzip = getOption("unzip")) on.exit(unlink(file.path(path))) # if (!is.null(preprocess)) { .data <- data.table::fread(file = path) .data <- preprocess(data = .data) data.table::fwrite(x = .data, file = path, append = FALSE) rm(.data) } # CSV sql <- sprintf( "COPY OFFSET 2 INTO %s FROM '%s' USING DELIMITERS ',','\\n','\"' NULL AS '' BEST EFFORT", tablename, path ) # DBI::dbExecute(con, sql) # DBI::dbExecute(con, sprintf("INSERT INTO upload_log(file_name, uploaded) VALUES('%s', true)", filename)) return(invisible(TRUE)) }
Si vous devez convertir la table avant d'écrire dans la base de données, il suffit de passer la fonction qui convertira les données en argument de preprocess .
Code pour le chargement séquentiel des données dans la base de données:
Écriture de données dans la base de données # files <- unzip(zipfile, list = TRUE)$Name # , to_skip <- DBI::dbGetQuery(con, "SELECT file_name FROM upload_log")[[1L]] files <- setdiff(files, to_skip) if (length(files) > 0L) { # tictoc::tic() # pb <- txtProgressBar(min = 0L, max = length(files), style = 3) for (i in seq_along(files)) { upload_file(con = con, tablename = "doodles", zipfile = zipfile, filename = files[i]) setTxtProgressBar(pb, i) } close(pb) # tictoc::toc() } # 526.141 sec elapsed - SSD->SSD # 558.879 sec elapsed - USB->SSD
Le temps de chargement des données peut varier en fonction des caractéristiques de vitesse du lecteur utilisé. Dans notre cas, la lecture et l'écriture sur le même SSD ou depuis une clé USB (fichier source) vers un SSD (base de données) prend moins de 10 minutes.
Il faut quelques secondes de plus pour créer une colonne avec une étiquette de classe entière et une colonne d'index ( ORDERED INDEX ) avec des numéros de ligne, qui seront utilisées pour sélectionner les cas lors de la création de lots:
Créer des colonnes et un index supplémentaires message("Generate lables") invisible(DBI::dbExecute(con, "ALTER TABLE doodles ADD label_int int")) invisible(DBI::dbExecute(con, "UPDATE doodles SET label_int = dense_rank() OVER (ORDER BY word) - 1")) message("Generate row numbers") invisible(DBI::dbExecute(con, "ALTER TABLE doodles ADD id serial")) invisible(DBI::dbExecute(con, "CREATE ORDERED INDEX doodles_id_ord_idx ON doodles(id)"))
Pour résoudre le problème de la création d'un lot «à la volée», nous devions atteindre la vitesse maximale d'extraction de chaînes aléatoires de la table de doodles . Pour cela, nous avons utilisé 3 astuces. La première consistait à réduire la dimension du type dans lequel l'ID d'observation est stocké. Dans le jeu de données d'origine, le type bigint est requis pour stocker l'ID, mais le nombre d'observations permet d'ajuster leurs identifiants égaux au numéro de série dans le type int . La recherche est beaucoup plus rapide. La deuxième astuce consistait à utiliser ORDERED INDEX - cette décision a été prise empiriquement, en triant toutes les options disponibles. La troisième consistait à utiliser des requêtes paramétrées. L'essence de la méthode est d'exécuter la commande PREPARE une fois, puis d'utiliser l'expression préparée pour créer un tas du même type de requêtes, mais en réalité le gain par rapport au simple SELECT dans le domaine des erreurs statistiques.
Le processus de remplissage des données ne consomme pas plus de 450 Mo de RAM. C'est-à-dire que l'approche décrite vous permet de faire pivoter des ensembles de données pesant des dizaines de gigaoctets sur presque n'importe quel matériel budgétaire, y compris certains ordinateurs à carte unique, ce qui est plutôt cool.
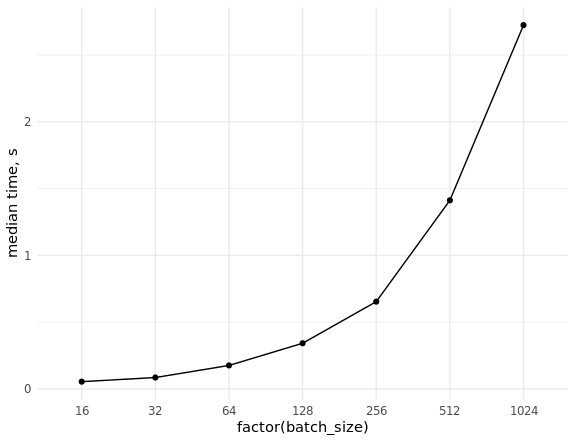
Il reste à prendre des mesures du taux d'extraction des données (aléatoires) et à évaluer la mise à l'échelle lors de l'échantillonnage de lots de tailles différentes:
Base de données de référence library(ggplot2) set.seed(0) # con <- DBI::dbConnect(MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR")) # prep_sql <- function(batch_size) { sql <- sprintf("PREPARE SELECT id FROM doodles WHERE id IN (%s)", paste(rep("?", batch_size), collapse = ",")) res <- DBI::dbSendQuery(con, sql) return(res) } # fetch_data <- function(rs, batch_size) { ids <- sample(seq_len(n), batch_size) res <- DBI::dbFetch(DBI::dbBind(rs, as.list(ids))) return(res) } # res_bench <- bench::press( batch_size = 2^(4:10), { rs <- prep_sql(batch_size) bench::mark( fetch_data(rs, batch_size), min_iterations = 50L ) } ) # cols <- c("batch_size", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # batch_size min median max `itr/sec` total_time n_itr # <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 16 23.6ms 54.02ms 93.43ms 18.8 2.6s 49 # 2 32 38ms 84.83ms 151.55ms 11.4 4.29s 49 # 3 64 63.3ms 175.54ms 248.94ms 5.85 8.54s 50 # 4 128 83.2ms 341.52ms 496.24ms 3.00 16.69s 50 # 5 256 232.8ms 653.21ms 847.44ms 1.58 31.66s 50 # 6 512 784.6ms 1.41s 1.98s 0.740 1.1m 49 # 7 1024 681.7ms 2.72s 4.06s 0.377 2.16m 49 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = 1)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() DBI::dbDisconnect(con, shutdown = TRUE)
2. Préparation des lots
L'ensemble du processus de préparation des lots comprend les étapes suivantes:
- Analyser plusieurs JSON contenant des vecteurs de ligne avec des coordonnées de point.
- Dessiner des lignes colorées par les coordonnées des points dans l'image de la taille souhaitée (par exemple, 256x256 ou 128x128).
- Convertissez les images résultantes en un tenseur.
Dans le cadre de la concurrence entre les noyaux en Python, le problème a été résolu principalement au moyen d' OpenCV . L'un des analogues les plus simples et les plus évidents sur R ressemblera à ceci:
Implémenter la conversion JSON en tensor sur R r_process_json_str <- function(json, line.width = 3, color = TRUE, scale = 1) { # JSON coords <- jsonlite::fromJSON(json, simplifyMatrix = FALSE) tmp <- tempfile() # on.exit(unlink(tmp)) png(filename = tmp, width = 256 * scale, height = 256 * scale, pointsize = 1) # plot.new() # plot.window(xlim = c(256 * scale, 0), ylim = c(256 * scale, 0)) # cols <- if (color) rainbow(length(coords)) else "#000000" for (i in seq_along(coords)) { lines(x = coords[[i]][[1]] * scale, y = coords[[i]][[2]] * scale, col = cols[i], lwd = line.width) } dev.off() # 3- res <- png::readPNG(tmp) return(res) } r_process_json_vector <- function(x, ...) { res <- lapply(x, r_process_json_str, ...) # 3- 4- res <- do.call(abind::abind, c(res, along = 0)) return(res) }
Le dessin est effectué à l'aide d'outils R standard et enregistré dans un PNG temporaire stocké dans la RAM (sous Linux, les répertoires R temporaires sont situés dans le /tmp monté dans la RAM). Ensuite, ce fichier est lu sous la forme d'un tableau tridimensionnel avec des nombres compris entre 0 et 1. Ceci est important, car le BMP le plus courant serait lu dans un tableau brut avec des codes de couleur hexadécimaux.
Testez le résultat:
zip_file <- file.path("data", "train_simplified.zip") csv_file <- "cat.csv" unzip(zip_file, files = csv_file, exdir = tempdir(), junkpaths = TRUE, unzip = getOption("unzip")) tmp_data <- data.table::fread(file.path(tempdir(), csv_file), sep = ",", select = "drawing", nrows = 10000) arr <- r_process_json_str(tmp_data[4, drawing]) dim(arr) # [1] 256 256 3 plot(magick::image_read(arr))

Le lot lui-même sera formé comme suit:
res <- r_process_json_vector(tmp_data[1:4, drawing], scale = 0.5) str(res) # num [1:4, 1:128, 1:128, 1:3] 1 1 1 1 1 1 1 1 1 1 ... # - attr(*, "dimnames")=List of 4 # ..$ : NULL # ..$ : NULL # ..$ : NULL # ..$ : NULL
Cette implémentation ne nous a pas semblé optimale, car la formation de gros lots prend indécemment beaucoup de temps, et nous avons décidé d'utiliser l'expérience de nos collègues utilisant la puissante bibliothèque OpenCV . À ce moment-là, il n'y avait pas de package prêt à l'emploi pour R (il n'y en a même pas maintenant), donc une implémentation minimale des fonctionnalités requises en C ++ a été écrite avec intégration dans le code R à l'aide de Rcpp .
Pour résoudre le problème, les packages et bibliothèques suivants ont été utilisés:
- OpenCV pour l'imagerie et le dessin au trait. Nous avons utilisé des bibliothèques système et des fichiers d'en-tête préinstallés, ainsi qu'une liaison dynamique.
- xtensor pour travailler avec des tableaux et tenseurs multidimensionnels. Nous avons utilisé des fichiers d'en-tête inclus dans le R-package du même nom. La bibliothèque vous permet de travailler avec des tableaux multidimensionnels, à la fois dans l'ordre des lignes principales et des colonnes.
- ndjson pour analyser JSON. Cette bibliothèque est utilisée dans xtensor automatiquement lorsqu'elle est disponible dans le projet.
- RcppThread pour organiser le traitement multi-thread d'un vecteur de JSON. Utilisé les fichiers d'en-tête fournis par ce package. Le package diffère du RcppParallel plus populaire entre autres par son mécanisme d'interruption intégré.
Il convient de noter que xtensor s'est avéré être une trouvaille: en plus d'avoir des fonctionnalités étendues et de hautes performances, ses développeurs se sont révélés assez réactifs et ont répondu rapidement et en détail aux questions qui se posaient. Avec leur aide, il a été possible de mettre en œuvre la transformation des matrices OpenCV en tenseurs xtensor, ainsi qu'un moyen de combiner des tenseurs d'images en 3 dimensions en un tenseur en 4 dimensions de la dimension correcte (en fait le lot).
Matériel d'étude pour Rcpp, xtensor et RcppThread Pour compiler des fichiers en utilisant des fichiers système et une liaison dynamique avec des bibliothèques installées dans le système, nous avons utilisé le mécanisme de plug-in implémenté dans le package Rcpp . Pour trouver automatiquement les chemins et les indicateurs, nous avons utilisé le populaire utilitaire linux pkg-config .
Implémentation d'un plugin Rcpp pour l'utilisation de la bibliothèque OpenCV Rcpp::registerPlugin("opencv", function() { # pkg_config_name <- c("opencv", "opencv4") # pkg-config pkg_config_bin <- Sys.which("pkg-config") # checkmate::assert_file_exists(pkg_config_bin, access = "x") # OpenCV pkg-config check <- sapply(pkg_config_name, function(pkg) system(paste(pkg_config_bin, pkg))) if (all(check != 0)) { stop("OpenCV config for the pkg-config not found", call. = FALSE) } pkg_config_name <- pkg_config_name[check == 0] list(env = list( PKG_CXXFLAGS = system(paste(pkg_config_bin, "--cflags", pkg_config_name), intern = TRUE), PKG_LIBS = system(paste(pkg_config_bin, "--libs", pkg_config_name), intern = TRUE) )) })
À la suite du plugin, les valeurs suivantes seront remplacées lors de la compilation:
Rcpp:::.plugins$opencv()$env # $PKG_CXXFLAGS # [1] "-I/usr/include/opencv" # # $PKG_LIBS # [1] "-lopencv_shape -lopencv_stitching -lopencv_superres -lopencv_videostab -lopencv_aruco -lopencv_bgsegm -lopencv_bioinspired -lopencv_ccalib -lopencv_datasets -lopencv_dpm -lopencv_face -lopencv_freetype -lopencv_fuzzy -lopencv_hdf -lopencv_line_descriptor -lopencv_optflow -lopencv_video -lopencv_plot -lopencv_reg -lopencv_saliency -lopencv_stereo -lopencv_structured_light -lopencv_phase_unwrapping -lopencv_rgbd -lopencv_viz -lopencv_surface_matching -lopencv_text -lopencv_ximgproc -lopencv_calib3d -lopencv_features2d -lopencv_flann -lopencv_xobjdetect -lopencv_objdetect -lopencv_ml -lopencv_xphoto -lopencv_highgui -lopencv_videoio -lopencv_imgcodecs -lopencv_photo -lopencv_imgproc -lopencv_core"
Le code pour implémenter l'analyse JSON et créer un lot pour le transfert vers le modèle est donné sous le spoiler. Tout d'abord, ajoutez le répertoire local du projet pour rechercher les fichiers d'en-tête (nécessaires pour ndjson):
Sys.setenv("PKG_CXXFLAGS" = paste0("-I", normalizePath(file.path("src"))))
Implémentation de la conversion JSON en tensor en C ++ // [[Rcpp::plugins(cpp14)]] // [[Rcpp::plugins(opencv)]] // [[Rcpp::depends(xtensor)]] // [[Rcpp::depends(RcppThread)]] #include <xtensor/xjson.hpp> #include <xtensor/xadapt.hpp> #include <xtensor/xview.hpp> #include <xtensor-r/rtensor.hpp> #include <opencv2/core/core.hpp> #include <opencv2/highgui/highgui.hpp> #include <opencv2/imgproc/imgproc.hpp> #include <Rcpp.h> #include <RcppThread.h> // using RcppThread::parallelFor; using json = nlohmann::json; using points = xt::xtensor<double,2>; // JSON using strokes = std::vector<points>; // JSON using xtensor3d = xt::xtensor<double, 3>; // using xtensor4d = xt::xtensor<double, 4>; // using rtensor3d = xt::rtensor<double, 3>; // R using rtensor4d = xt::rtensor<double, 4>; // R // // const static int SIZE = 256; // // . https://en.wikipedia.org/wiki/Pixel_connectivity#2-dimensional const static int LINE_TYPE = cv::LINE_4; // const static int LINE_WIDTH = 3; // // https://docs.opencv.org/3.1.0/da/d54/group__imgproc__transform.html#ga5bb5a1fea74ea38e1a5445ca803ff121 const static int RESIZE_TYPE = cv::INTER_LINEAR; // OpenCV- template <typename T, int NCH, typename XT=xt::xtensor<T,3,xt::layout_type::column_major>> XT to_xt(const cv::Mat_<cv::Vec<T, NCH>>& src) { // std::vector<int> shape = {src.rows, src.cols, NCH}; // size_t size = src.total() * NCH; // cv::Mat xt::xtensor XT res = xt::adapt((T*) src.data, size, xt::no_ownership(), shape); return res; } // JSON strokes parse_json(const std::string& x) { auto j = json::parse(x); // if (!j.is_array()) { throw std::runtime_error("'x' must be JSON array."); } strokes res; res.reserve(j.size()); for (const auto& a: j) { // 2- if (!a.is_array() || a.size() != 2) { throw std::runtime_error("'x' must include only 2d arrays."); } // auto p = a.get<points>(); res.push_back(p); } return res; } // // HSV cv::Mat ocv_draw_lines(const strokes& x, bool color = true) { // auto stype = color ? CV_8UC3 : CV_8UC1; // auto dtype = color ? CV_32FC3 : CV_32FC1; auto bg = color ? cv::Scalar(0, 0, 255) : cv::Scalar(255); auto col = color ? cv::Scalar(0, 255, 220) : cv::Scalar(0); cv::Mat img = cv::Mat(SIZE, SIZE, stype, bg); // size_t n = x.size(); for (const auto& s: x) { // size_t n_points = s.shape()[1]; for (size_t i = 0; i < n_points - 1; ++i) { // cv::Point from(s(0, i), s(1, i)); // cv::Point to(s(0, i + 1), s(1, i + 1)); // cv::line(img, from, to, col, LINE_WIDTH, LINE_TYPE); } if (color) { // col[0] += 180 / n; } } if (color) { // RGB cv::cvtColor(img, img, cv::COLOR_HSV2RGB); } // float32 [0, 1] img.convertTo(img, dtype, 1 / 255.0); return img; } // JSON xtensor3d process(const std::string& x, double scale = 1.0, bool color = true) { auto p = parse_json(x); auto img = ocv_draw_lines(p, color); if (scale != 1) { cv::Mat out; cv::resize(img, out, cv::Size(), scale, scale, RESIZE_TYPE); cv::swap(img, out); out.release(); } xtensor3d arr = color ? to_xt<double,3>(img) : to_xt<double,1>(img); return arr; } // [[Rcpp::export]] rtensor3d cpp_process_json_str(const std::string& x, double scale = 1.0, bool color = true) { xtensor3d res = process(x, scale, color); return res; } // [[Rcpp::export]] rtensor4d cpp_process_json_vector(const std::vector<std::string>& x, double scale = 1.0, bool color = false) { size_t n = x.size(); size_t dim = floor(SIZE * scale); size_t channels = color ? 3 : 1; xtensor4d res({n, dim, dim, channels}); parallelFor(0, n, [&x, &res, scale, color](int i) { xtensor3d tmp = process(x[i], scale, color); auto view = xt::view(res, i, xt::all(), xt::all(), xt::all()); view = tmp; }); return res; }
Ce code doit être placé dans le src/cv_xt.cpp et compilé avec la commande Rcpp::sourceCpp(file = "src/cv_xt.cpp", env = .GlobalEnv) ; vous aurez également besoin de nlohmann/json.hpp du référentiel pour fonctionner . Le code est divisé en plusieurs fonctions:
to_xt - une fonction de modèle pour convertir la matrice d'image ( cv::Mat ) en le tenseur xt::xtensor ;parse_json - la fonction analyse une chaîne JSON, extrait les coordonnées des points et les place dans un vecteur;ocv_draw_lines - ocv_draw_lines des lignes multicolores à partir du vecteur de points reçu;process - combine les fonctions ci-dessus et ajoute également la possibilité de mettre à l'échelle l'image résultante;cpp_process_json_str - un wrapper sur la fonction de process , qui exporte le résultat vers un objet R (tableau multidimensionnel);cpp_process_json_vector - un wrapper sur la fonction cpp_process_json_str , qui vous permet de traiter un vecteur chaîne en mode multi-thread.
Pour dessiner des lignes multicolores, le modèle de couleur HSV a été utilisé, suivi d'une conversion en RVB. Testez le résultat:
arr <- cpp_process_json_str(tmp_data[4, drawing]) dim(arr) # [1] 256 256 3 plot(magick::image_read(arr))

Comparaison de la vitesse des implémentations en R et C ++ res_bench <- bench::mark( r_process_json_str(tmp_data[4, drawing], scale = 0.5), cpp_process_json_str(tmp_data[4, drawing], scale = 0.5), check = FALSE, min_iterations = 100 ) # cols <- c("expression", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # expression min median max `itr/sec` total_time n_itr # <chr> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 r_process_json_str 3.49ms 3.55ms 4.47ms 273. 490ms 134 # 2 cpp_process_json_str 1.94ms 2.02ms 5.32ms 489. 497ms 243 library(ggplot2) # res_bench <- bench::press( batch_size = 2^(4:10), { .data <- tmp_data[sample(seq_len(.N), batch_size), drawing] bench::mark( r_process_json_vector(.data, scale = 0.5), cpp_process_json_vector(.data, scale = 0.5), min_iterations = 50, check = FALSE ) } ) res_bench[, cols] # expression batch_size min median max `itr/sec` total_time n_itr # <chr> <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 r 16 50.61ms 53.34ms 54.82ms 19.1 471.13ms 9 # 2 cpp 16 4.46ms 5.39ms 7.78ms 192. 474.09ms 91 # 3 r 32 105.7ms 109.74ms 212.26ms 7.69 6.5s 50 # 4 cpp 32 7.76ms 10.97ms 15.23ms 95.6 522.78ms 50 # 5 r 64 211.41ms 226.18ms 332.65ms 3.85 12.99s 50 # 6 cpp 64 25.09ms 27.34ms 32.04ms 36.0 1.39s 50 # 7 r 128 534.5ms 627.92ms 659.08ms 1.61 31.03s 50 # 8 cpp 128 56.37ms 58.46ms 66.03ms 16.9 2.95s 50 # 9 r 256 1.15s 1.18s 1.29s 0.851 58.78s 50 # 10 cpp 256 114.97ms 117.39ms 130.09ms 8.45 5.92s 50 # 11 r 512 2.09s 2.15s 2.32s 0.463 1.8m 50 # 12 cpp 512 230.81ms 235.6ms 261.99ms 4.18 11.97s 50 # 13 r 1024 4s 4.22s 4.4s 0.238 3.5m 50 # 14 cpp 1024 410.48ms 431.43ms 462.44ms 2.33 21.45s 50 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = expression, color = expression)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() + scale_color_discrete(name = "", labels = c("cpp", "r")) + theme(legend.position = "bottom")
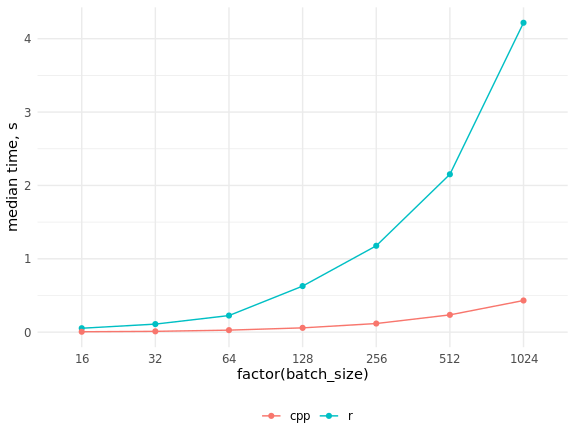
Comme vous pouvez le voir, l'augmentation de vitesse s'est avérée très importante et il n'est pas possible de rattraper le code C ++ en parallélisant le code R.
3. Itérateurs pour décharger des lots de la base de données
R a une réputation bien méritée en tant que langage de traitement des données situées dans la RAM, tandis que Python est davantage caractérisé par un traitement itératif des données, ce qui facilite et simplifie la mise en œuvre de calculs hors cœur (calculs utilisant la mémoire externe). Classiques et pertinents pour nous dans le contexte du problème décrit, un exemple de tels calculs est les réseaux de neurones profonds, formés par la méthode de la descente du gradient avec approximation du gradient à chaque étape par une petite portion d'observations, ou un mini-lot.
Les frameworks d'apprentissage en profondeur écrits en Python ont des classes spéciales qui implémentent des itérateurs basés sur des données: tableaux, images dans des dossiers, formats binaires, etc. Vous pouvez utiliser des options prédéfinies ou écrire les vôtres pour des tâches spécifiques. Dans R, nous pouvons tirer pleinement parti de la bibliothèque Keras Python avec ses différents backends en utilisant le package du même nom, qui à son tour fonctionne au-dessus du package réticulé . Ce dernier mérite un grand article séparé; il vous permet non seulement d'exécuter du code Python à partir de R, mais fournit également le transfert d'objets entre les sessions R et Python, effectuant automatiquement toutes les conversions de type nécessaires.
Nous nous sommes débarrassés de la nécessité de stocker toutes les données dans la RAM en raison de l'utilisation de MonetDBLite, tout le travail du «réseau neuronal» sera effectué par le code Python original, nous n'avons qu'à écrire un itérateur basé sur les données, car il n'y a pas de prêt pour une telle situation dans R ou Python. : ( R ). R numpy-, keras .
:
train_generator <- function(db_connection = con, samples_index, num_classes = 340, batch_size = 32, scale = 1, color = FALSE, imagenet_preproc = FALSE) { # checkmate::assert_class(con, "DBIConnection") checkmate::assert_integerish(samples_index) checkmate::assert_count(num_classes) checkmate::assert_count(batch_size) checkmate::assert_number(scale, lower = 0.001, upper = 5) checkmate::assert_flag(color) checkmate::assert_flag(imagenet_preproc) # , dt <- data.table::data.table(id = sample(samples_index)) # dt[, batch := (.I - 1L) %/% batch_size + 1L] # dt <- dt[, if (.N == batch_size) .SD, keyby = batch] # i <- 1 # max_i <- dt[, max(batch)] # sql <- sprintf( "PREPARE SELECT drawing, label_int FROM doodles WHERE id IN (%s)", paste(rep("?", batch_size), collapse = ",") ) res <- DBI::dbSendQuery(con, sql) # keras::to_categorical to_categorical <- function(x, num) { n <- length(x) m <- numeric(n * num) m[x * n + seq_len(n)] <- 1 dim(m) <- c(n, num) return(m) } # function() { # if (i > max_i) { dt[, id := sample(id)] data.table::setkey(dt, batch) # i <<- 1 max_i <<- dt[, max(batch)] } # ID batch_ind <- dt[batch == i, id] # batch <- DBI::dbFetch(DBI::dbBind(res, as.list(batch_ind)), n = -1) # i <<- i + 1 # JSON batch_x <- cpp_process_json_vector(batch$drawing, scale = scale, color = color) if (imagenet_preproc) { # c [0, 1] [-1, 1] batch_x <- (batch_x - 0.5) * 2 } batch_y <- to_categorical(batch$label_int, num_classes) result <- list(batch_x, batch_y) return(result) } }
, , , , ( scale = 1 256256 , scale = 0.5 — 128128 ), ( color = FALSE , color = TRUE ) , imagenet-. , [0, 1] [-1, 1], keras .
, data.table samples_index , , SQL- . keras::to_categorical() . , , steps_per_epoch keras::fit_generator() , if (i > max_i) .
, , JSON- ( cpp_process_json_vector() , C++) , . one-hot , , . data.table — "" data.table - R.
Core i5 :
library(Rcpp) library(keras) library(ggplot2) source("utils/rcpp.R") source("utils/keras_iterator.R") con <- DBI::dbConnect(drv = MonetDBLite::MonetDBLite(), Sys.getenv("DBDIR")) ind <- seq_len(DBI::dbGetQuery(con, "SELECT count(*) FROM doodles")[[1L]]) num_classes <- DBI::dbGetQuery(con, "SELECT max(label_int) + 1 FROM doodles")[[1L]] # train_ind <- sample(ind, floor(length(ind) * 0.995)) # val_ind <- ind[-train_ind] rm(ind) # scale <- 0.5 # res_bench <- bench::press( batch_size = 2^(4:10), { it1 <- train_generator( db_connection = con, samples_index = train_ind, num_classes = num_classes, batch_size = batch_size, scale = scale ) bench::mark( it1(), min_iterations = 50L ) } ) # cols <- c("batch_size", "min", "median", "max", "itr/sec", "total_time", "n_itr") res_bench[, cols] # batch_size min median max `itr/sec` total_time n_itr # <dbl> <bch:tm> <bch:tm> <bch:tm> <dbl> <bch:tm> <int> # 1 16 25ms 64.36ms 92.2ms 15.9 3.09s 49 # 2 32 48.4ms 118.13ms 197.24ms 8.17 5.88s 48 # 3 64 69.3ms 117.93ms 181.14ms 8.57 5.83s 50 # 4 128 157.2ms 240.74ms 503.87ms 3.85 12.71s 49 # 5 256 359.3ms 613.52ms 988.73ms 1.54 30.5s 47 # 6 512 884.7ms 1.53s 2.07s 0.674 1.11m 45 # 7 1024 2.7s 3.83s 5.47s 0.261 2.81m 44 ggplot(res_bench, aes(x = factor(batch_size), y = median, group = 1)) + geom_point() + geom_line() + ylab("median time, s") + theme_minimal() DBI::dbDisconnect(con, shutdown = TRUE)

, ( 32 ). /dev/shm , . , /etc/fstab , tmpfs /dev/shm tmpfs defaults,size=25g 0 0 . , df -h .
, :
test_generator <- function(dt, batch_size = 32, scale = 1, color = FALSE, imagenet_preproc = FALSE) { # checkmate::assert_data_table(dt) checkmate::assert_count(batch_size) checkmate::assert_number(scale, lower = 0.001, upper = 5) checkmate::assert_flag(color) checkmate::assert_flag(imagenet_preproc) # dt[, batch := (.I - 1L) %/% batch_size + 1L] data.table::setkey(dt, batch) i <- 1 max_i <- dt[, max(batch)] # function() { batch_x <- cpp_process_json_vector(dt[batch == i, drawing], scale = scale, color = color) if (imagenet_preproc) { # c [0, 1] [-1, 1] batch_x <- (batch_x - 0.5) * 2 } result <- list(batch_x) i <<- i + 1 return(result) } }
4.
mobilenet v1 , . keras , , R. : (batch, height, width, 3) , . Python , , ( , keras- ):
mobilenet v1 library(keras) top_3_categorical_accuracy <- custom_metric( name = "top_3_categorical_accuracy", metric_fn = function(y_true, y_pred) { metric_top_k_categorical_accuracy(y_true, y_pred, k = 3) } ) layer_sep_conv_bn <- function(object, filters, alpha = 1, depth_multiplier = 1, strides = c(2, 2)) { # NB! depth_multiplier != resolution multiplier # https://github.com/keras-team/keras/issues/10349 layer_depthwise_conv_2d( object = object, kernel_size = c(3, 3), strides = strides, padding = "same", depth_multiplier = depth_multiplier ) %>% layer_batch_normalization() %>% layer_activation_relu() %>% layer_conv_2d( filters = filters * alpha, kernel_size = c(1, 1), strides = c(1, 1) ) %>% layer_batch_normalization() %>% layer_activation_relu() } get_mobilenet_v1 <- function(input_shape = c(224, 224, 1), num_classes = 340, alpha = 1, depth_multiplier = 1, optimizer = optimizer_adam(lr = 0.002), loss = "categorical_crossentropy", metrics = c("categorical_crossentropy", top_3_categorical_accuracy)) { inputs <- layer_input(shape = input_shape) outputs <- inputs %>% layer_conv_2d(filters = 32, kernel_size = c(3, 3), strides = c(2, 2), padding = "same") %>% layer_batch_normalization() %>% layer_activation_relu() %>% layer_sep_conv_bn(filters = 64, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 128, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 128, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 256, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 256, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 512, strides = c(1, 1)) %>% layer_sep_conv_bn(filters = 1024, strides = c(2, 2)) %>% layer_sep_conv_bn(filters = 1024, strides = c(1, 1)) %>% layer_global_average_pooling_2d() %>% layer_dense(units = num_classes) %>% layer_activation_softmax() model <- keras_model( inputs = inputs, outputs = outputs ) model %>% compile( optimizer = optimizer, loss = loss, metrics = metrics ) return(model) }
. , , , . , imagenet-. , . get_config() ( base_model_conf$layers — R- ), from_config() :
base_model_conf <- get_config(base_model) base_model_conf$layers[[1]]$config$batch_input_shape[[4]] <- 1L base_model <- from_config(base_model_conf)
keras imagenet- :
get_model <- function(name = "mobilenet_v2", input_shape = NULL, weights = "imagenet", pooling = "avg", num_classes = NULL, optimizer = keras::optimizer_adam(lr = 0.002), loss = "categorical_crossentropy", metrics = NULL, color = TRUE, compile = FALSE) { # checkmate::assert_string(name) checkmate::assert_integerish(input_shape, lower = 1, upper = 256, len = 3) checkmate::assert_count(num_classes) checkmate::assert_flag(color) checkmate::assert_flag(compile) # keras model_fun <- get0(paste0("application_", name), envir = asNamespace("keras")) # if (is.null(model_fun)) { stop("Model ", shQuote(name), " not found.", call. = FALSE) } base_model <- model_fun( input_shape = input_shape, include_top = FALSE, weights = weights, pooling = pooling ) # , if (!color) { base_model_conf <- keras::get_config(base_model) base_model_conf$layers[[1]]$config$batch_input_shape[[4]] <- 1L base_model <- keras::from_config(base_model_conf) } predictions <- keras::get_layer(base_model, "global_average_pooling2d_1")$output predictions <- keras::layer_dense(predictions, units = num_classes, activation = "softmax") model <- keras::keras_model( inputs = base_model$input, outputs = predictions ) if (compile) { keras::compile( object = model, optimizer = optimizer, loss = loss, metrics = metrics ) } return(model) }
. : get_weights() R- , ( - ), set_weights() . , , .
mobilenet 1 2, resnet34. , SE-ResNeXt. , , ( ).
5.
, docopt :
doc <- ' Usage: train_nn.R --help train_nn.R --list-models train_nn.R [options] Options: -h --help Show this message. -l --list-models List available models. -m --model=<model> Neural network model name [default: mobilenet_v2]. -b --batch-size=<size> Batch size [default: 32]. -s --scale-factor=<ratio> Scale factor [default: 0.5]. -c --color Use color lines [default: FALSE]. -d --db-dir=<path> Path to database directory [default: Sys.getenv("db_dir")]. -r --validate-ratio=<ratio> Validate sample ratio [default: 0.995]. -n --n-gpu=<number> Number of GPUs [default: 1]. ' args <- docopt::docopt(doc)
docopt http://docopt.org/ R. Rscript bin/train_nn.R -m resnet50 -c -d /home/andrey/doodle_db ./bin/train_nn.R -m resnet50 -c -d /home/andrey/doodle_db , train_nn.R ( resnet50 128128 , /home/andrey/doodle_db ). , . , mobilenet_v2 keras R - R- — , .
RStudio ( tfruns ). , RStudio.
6.
. R- .
« », . , NVIDIA, CUDA+cuDNN — , tensorflow/tensorflow:1.12.0-gpu , R-.
- :
Dockerfile FROM tensorflow/tensorflow:1.12.0-gpu MAINTAINER Artem Klevtsov <aaklevtsov@gmail.com> SHELL ["/bin/bash", "-c"] ARG LOCALE="en_US.UTF-8" ARG APT_PKG="libopencv-dev r-base r-base-dev littler" ARG R_BIN_PKG="futile.logger checkmate data.table rcpp rapidjsonr dbi keras jsonlite curl digest remotes" ARG R_SRC_PKG="xtensor RcppThread docopt MonetDBLite" ARG PY_PIP_PKG="keras" ARG DIRS="/db /app /app/data /app/models /app/logs" RUN source /etc/os-release && \ echo "deb https://cloud.r-project.org/bin/linux/ubuntu ${UBUNTU_CODENAME}-cran35/" > /etc/apt/sources.list.d/cran35.list && \ apt-key adv --keyserver keyserver.ubuntu.com --recv-keys E084DAB9 && \ add-apt-repository -y ppa:marutter/c2d4u3.5 && \ add-apt-repository -y ppa:timsc/opencv-3.4 && \ apt-get update && \ apt-get install -y locales && \ locale-gen ${LOCALE} && \ apt-get install -y --no-install-recommends ${APT_PKG} && \ ln -s /usr/lib/R/site-library/littler/examples/install.r /usr/local/bin/install.r && \ ln -s /usr/lib/R/site-library/littler/examples/install2.r /usr/local/bin/install2.r && \ ln -s /usr/lib/R/site-library/littler/examples/installGithub.r /usr/local/bin/installGithub.r && \ echo 'options(Ncpus = parallel::detectCores())' >> /etc/R/Rprofile.site && \ echo 'options(repos = c(CRAN = "https://cloud.r-project.org"))' >> /etc/R/Rprofile.site && \ apt-get install -y $(printf "r-cran-%s " ${R_BIN_PKG}) && \ install.r ${R_SRC_PKG} && \ pip install ${PY_PIP_PKG} && \ mkdir -p ${DIRS} && \ chmod 777 ${DIRS} && \ rm -rf /tmp/downloaded_packages/ /tmp/*.rds && \ rm -rf /var/lib/apt/lists/* COPY utils /app/utils COPY src /app/src COPY tests /app/tests COPY bin/*.R /app/ ENV DBDIR="/db" ENV CUDA_HOME="/usr/local/cuda" ENV PATH="/app:${PATH}" WORKDIR /app VOLUME /db VOLUME /app CMD bash
; . /bin/bash /etc/os-release . .
-, . , , , :
#!/bin/sh DBDIR=${PWD}/db LOGSDIR=${PWD}/logs MODELDIR=${PWD}/models DATADIR=${PWD}/data ARGS="--runtime=nvidia --rm -v ${DBDIR}:/db -v ${LOGSDIR}:/app/logs -v ${MODELDIR}:/app/models -v ${DATADIR}:/app/data" if [ -z "$1" ]; then CMD="Rscript /app/train_nn.R" elif [ "$1" = "bash" ]; then ARGS="${ARGS} -ti" else CMD="Rscript /app/train_nn.R $@" fi docker run ${ARGS} doodles-tf ${CMD}
- , train_nn.R ; — "bash", . : CMD="Rscript /app/train_nn.R $@" .
, , , .
7. GPU Google Cloud
(. , @Leigh.plt ODS-). , 1 GPU GPU . GoogleCloud ( ) - , $300. 4V100 SSD , . , . K80. — SSD c, dev/shm .
, GPU. CPU , :
with(tensorflow::tf$device("/cpu:0"), { model_cpu <- get_model( name = model_name, input_shape = input_shape, weights = weights, metrics =(top_3_categorical_accuracy, compile = FALSE ) })
( ) GPU, :
model <- keras::multi_gpu_model(model_cpu, gpus = n_gpu) keras::compile( object = model, optimizer = keras::optimizer_adam(lr = 0.0004), loss = "categorical_crossentropy", metrics = c(top_3_categorical_accuracy) )
, , , GPU .
tensorboard , :
# log_file_tmpl <- file.path("logs", sprintf( "%s_%d_%dch_%s.csv", model_name, dim_size, channels, format(Sys.time(), "%Y%m%d%H%M%OS") )) # model_file_tmpl <- file.path("models", sprintf( "%s_%d_%dch_{epoch:02d}_{val_loss:.2f}.h5", model_name, dim_size, channels )) callbacks_list <- list( keras::callback_csv_logger( filename = log_file_tmpl ), keras::callback_early_stopping( monitor = "val_loss", min_delta = 1e-4, patience = 8, verbose = 1, mode = "min" ), keras::callback_reduce_lr_on_plateau( monitor = "val_loss", factor = 0.5, # lr 2 patience = 4, verbose = 1, min_delta = 1e-4, mode = "min" ), keras::callback_model_checkpoint( filepath = model_file_tmpl, monitor = "val_loss", save_best_only = FALSE, save_weights_only = FALSE, mode = "min" ) )
8.
, , :
- keras (
lr_finder fast.ai ); , R , , ; - , GPU;
- , imagenet-;
- one cycle policy discriminative learning rates (osine annealing , skeydan ).
:
- ( ) . data.table in-place , , . .
- R C++ Rcpp . RcppThread RcppParallel , , R .
- Rcpp C++, . xtensor CRAN, , R C++. — ++ RStudio.
- docopt . , .. . RStudio , IDE .
- , . .
- Google Cloud — , .
- , R C++, bench — .
, .