 Protocole expérimental et implémentation d'un algorithme de tri sur un ordinateur ADN programmable
Protocole expérimental et implémentation d'un algorithme de tri sur un ordinateur ADN programmableLes scientifiques expérimentent depuis longtemps le stockage d'informations dans l'ADN et le traitement de ces informations. Par exemple, des scientifiques de l'Université de Washington et de Microsoft ont récemment construit le
«premier ADN Winchester au monde» (
photo ). Cette conception est capable pour la première fois de fournir l'enregistrement et la lecture d'informations dans le stockage d'ADN sans intervention humaine. Une réalisation très importante, étant donné que l'ADN peut enregistrer des informations avec une densité de
2,2 pétaoctets par gramme . L'ADN est un conteneur compact avec une densité d'enregistrement des milliers de fois supérieure à celle des supports existants.
Cependant, tous les systèmes d'ADN existants ont un problème: ce sont tous des développements propriétaires uniques qui manquent complètement de flexibilité. Si nous comparons avec la technologie du silicium, chaque groupe de chercheurs à partir de zéro développe une nouvelle architecture informatique, pour laquelle vous devez écrire de nouveaux logiciels. Mais les choses peuvent changer grâce au
premier ordinateur ADN programmable développé à UC Davis, au California Institute of Technology et à l'Université de Maynooth.
Le premier ordinateur programmable ADN est décrit dans un
article scientifique publié le 20 mars 2019 dans la revue Nature. Les auteurs ont montré qu'à l'aide d'un simple déclencheur, le même ensemble de base de molécules d'ADN est capable de mettre en œuvre de nombreux algorithmes différents. Bien que l'étude soit une expérience purement de laboratoire, les algorithmes moléculaires programmables à l'avenir peuvent être utilisés, par exemple, pour programmer des robots à ADN qui
délivrent déjà avec succès
des médicaments aux cellules cancéreuses .
«Il s'agit de l'un des travaux phares dans ce domaine»,
explique Torsten-Lars Schmidt, professeur adjoint de biophysique expérimentale à l'Université de Kent, qui n'a pas participé à la recherche. "Ils avaient l'habitude de démontrer l'auto-assemblage algorithmique, mais pas à ce degré de complexité."
Dans les ordinateurs électroniques, les bits sont des unités binaires d'information. Ils représentent l'état physique discret de l'équipement de base, par exemple, la présence ou l'absence d'un courant électrique. Ces bits, ou plutôt des signaux électriques, passent par des circuits constitués d'éléments logiques qui effectuent une opération sur un ou plusieurs bits d'entrée et produisent un bit en sortie.
En combinant ces blocs de construction simples encore et encore, les ordinateurs peuvent exécuter des programmes incroyablement complexes. L'idée de l'informatique ADN est de remplacer les signaux électriques par des liaisons chimiques et le silicium par des acides nucléiques pour créer un logiciel biomoléculaire.
 Hiérarchie abstraite de l'architecture et mise en œuvre pratique du circuit logique IBC complet à 6 bits (circuit booléen itéré)
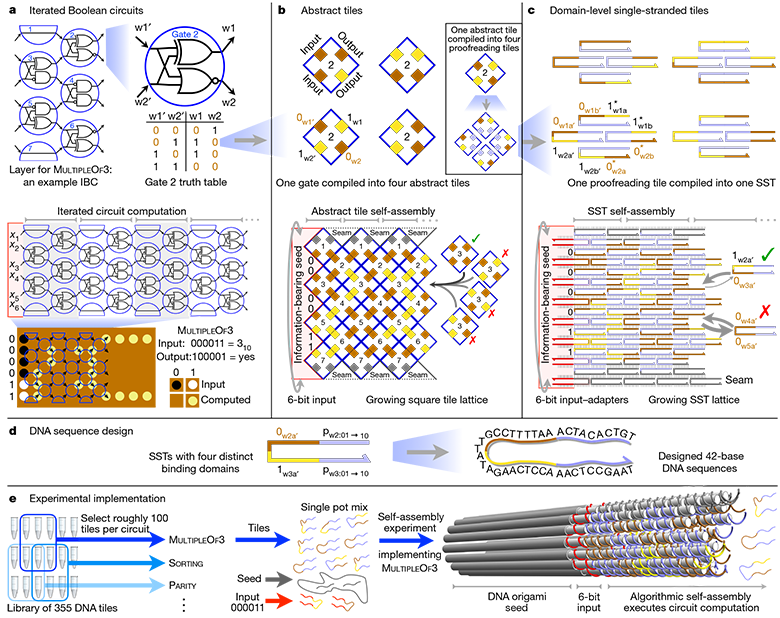
Hiérarchie abstraite de l'architecture et mise en œuvre pratique du circuit logique IBC complet à 6 bits (circuit booléen itéré)Selon Eric Winfrey, scientifique au California Institute of Technology et co-auteur de l'article, les algorithmes moléculaires utilisent les possibilités naturelles de traitement des informations dans l'ADN, mais au lieu de laisser la nature prendre les rênes de ses propres mains, les calculs dans l'ADN sont effectués conformément à un programme écrit par l'homme.
Au cours des 20 dernières années, plusieurs expériences réussies avec des algorithmes moléculaires ont été menées, par exemple, pour jouer au tic-tac-toe ou assembler des molécules de formes diverses. Dans chaque cas, un développement minutieux de la séquence d'ADN était nécessaire afin d'exécuter un algorithme spécifique qui générerait la structure d'ADN. Dans ce cas, la différence est que les chercheurs ont développé un système dans lequel les mêmes fragments d'ADN de base peuvent être commandés pour créer
des algorithmes complètement différents - et, par conséquent, pour obtenir des résultats complètement différents.
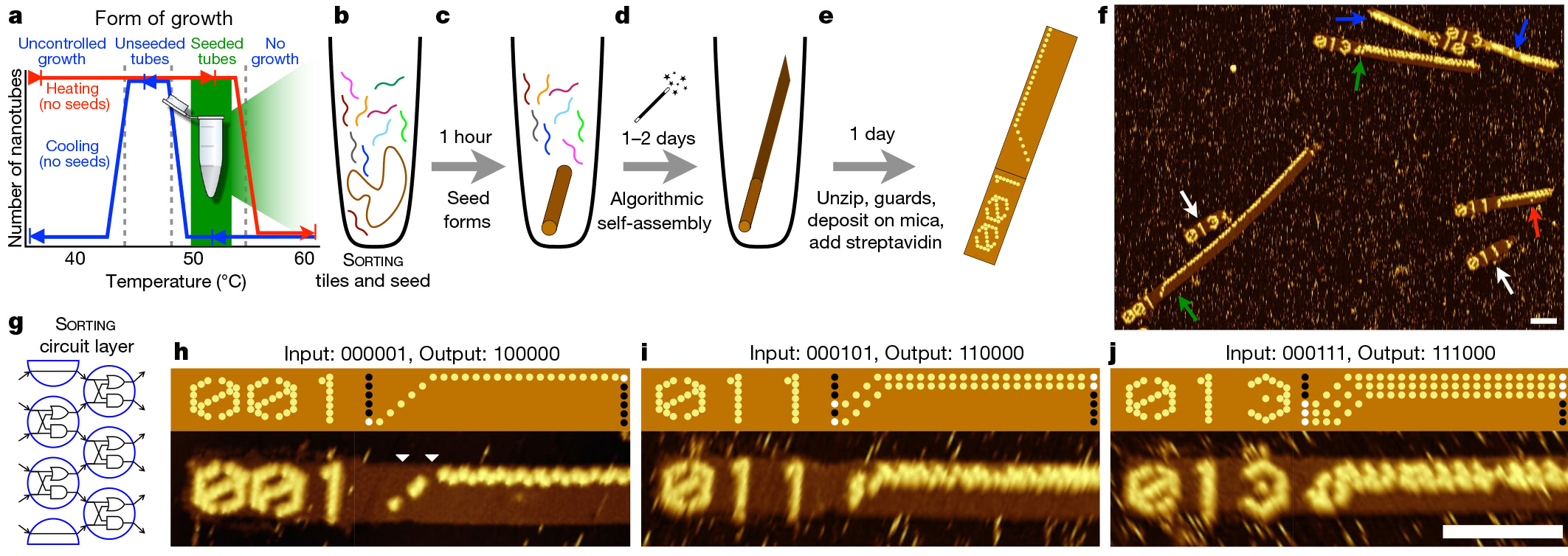
Le processus commence par la technique de l'ADN origami, c'est-à-dire le pliage d'une longue chaîne d'ADN dans la forme souhaitée. Cette tranche pliée fonctionne comme une graine, qui exécute une chaîne d'assemblage algorithmique. La graine reste pratiquement inchangée, quel que soit l'algorithme. Pour chaque expérience, seuls de petits changements y sont apportés en plusieurs séquences.
 Reprogrammer le circuit logique
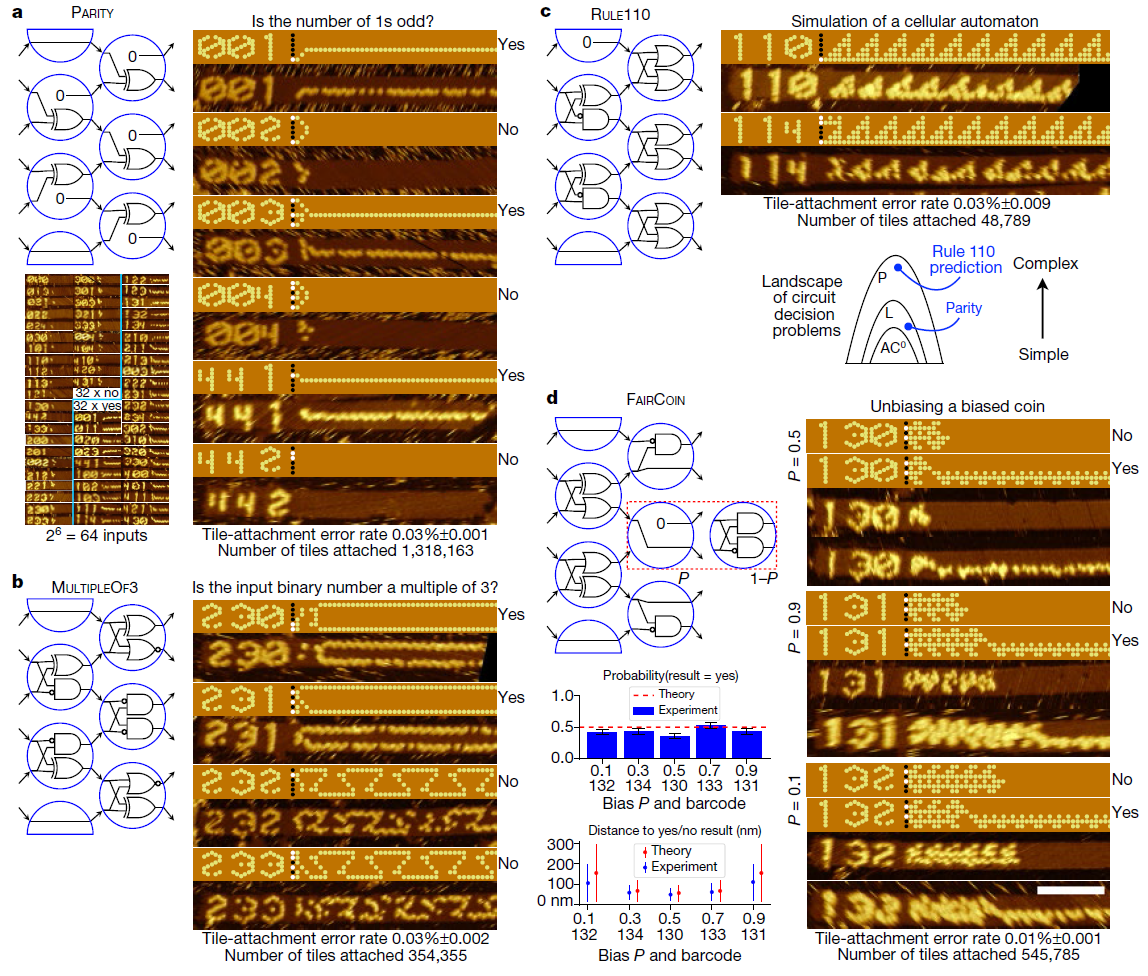
Reprogrammer le circuit logiqueAprès avoir créé la «graine», elle est ajoutée à la solution avec des centaines d'autres brins d'ADN, appelés tuiles d'ADN. Les scientifiques ont développé 355 de ces tuiles. Chacun a un arrangement unique de bases azotées. En conséquence, pour chaque algorithme, les chercheurs choisissent simplement un ensemble différent de tuiles de départ. Étant donné que ces fragments d'ADN sont joints pendant le processus d'assemblage, ils forment un circuit qui implémente l'algorithme moléculaire sélectionné sur les bits d'entrée fournis par la «graine».
En utilisant ce système, les chercheurs ont développé et testé 21 algorithmes pour effectuer des tâches telles que reconnaître une division par trois,
choisir un leader , générer des modèles et compter de 0 à 63. Tous ces algorithmes sont mis en œuvre en utilisant différentes combinaisons des mêmes 355 tuiles d'ADN.
Bien sûr, il n'est pas facile d'écrire du code en déposant des fragments d'ADN dans un tube à essai, mais si le processus est automatisé, les futurs programmeurs moléculaires n'ont même pas à penser à la biomécanique, car les programmeurs d'aujourd'hui n'ont pas besoin de comprendre la physique des transistors pour écrire de bons programmes.