Avant de lire cette mise à niveau, il est conseillé de vous familiariser avec le code source publié précédemment .Le tout premier et le plus populaire séquenceur de pores nanion MinION (le minimum fixé au Royaume-Uni est de 1000 $, en Russie - 150 000 roubles), développé par Oxford Nanopore Technologies (ONT), fonctionne sur des cellules jetables, dont chacune coûte 900 $ (en Russie - 135 mille roubles). Ces cellules vous permettent de numériser de l'ADN de 10 à 20 Go en 2 à 3 jours. C'est trop peu pour séquencer le génome humain (vous avez besoin de> 100 Go), mais trop pour toutes les autres fins de diagnostic clinique (<1 Go est suffisant).
Récemment, a commencé à vendre des modèles plus productifs (et plus chers) - PromethION 24/48 (165 000 $ / 285 000 $), destinés principalement au séquençage en ligne du génome complet. Plus chers sont leurs consommables plus efficaces (2 000 $ / unité,> 100 Go). Certes, ils peuvent coûter moins cher en vrac (1800000 $ pour 2880 pièces, 625 $ / pièces), mais de telles acquisitions (200 ... 250 millions de roubles par lot) ne sont pas autorisées pour le budget russe.
Et pas seulement pour le russe. Par conséquent, la plus grande attention des visiteurs de la récente conférence ONT (22-24 mai) a été attirée par l'annonce du début des ventes de Flongle, un adaptateur pour le séquenceur MinION, qui permet de travailler avec des cellules jetables moins efficaces (~ 1 Go), mais relativement bon marché (90 $).
 https://nanoporetech.com/products
https://nanoporetech.com/productsL'apparition de Flongle peut provoquer une augmentation explosive de l'utilisation du NGS (séquençage de nouvelle génération) dans la pratique clinique et déplacer les diagnostics conventionnels par PCR, dont la taille du marché en Russie dépasse le milliard de roubles, et dans le monde, elle est mesurée en milliards de dollars. Mais pour la plupart des patients russes, les diagnostics NGS resteront toujours un luxe inadmissible, car le coût des cellules pour Flongle sur le marché intérieur sera de plus de 10 000 roubles. Et si nous y ajoutons le coût des réactifs consommables, les coûts de transport et les frais généraux, le coût estimé du séquençage d'un échantillon d'ADN / ARN ne tombera pas en dessous de 20 000 roubles.
Cela implique la nécessité de développer des séquenceurs de nanopores nationaux et de leur fournir des consommables et des réactifs bon marché.
Nanorider
Il est possible de réduire considérablement le coût du séquençage des nanopores en réutilisant les cellules pour Flongle et en important la substitution des réactifs nécessaires pour travailler avec eux. Mais vous devez d'abord obtenir un appareil de test qui vous permet de surveiller la formation de membranes lipidiques bicouches (BLM) dans 126 puits de la cellule de travail, et d'évaluer la quantité et la qualité des nanopores incorporés dans ces membranes. Un tel dispositif ("Nanorider"?) Devrait suivre les changements dans la conductivité électrique (et / ou l'impédance) des puits individuels pendant la formation de BLM sur eux, et enregistrer les courants pico-ampères circulant à travers les canaux ioniques uniques.
Il est possible de composer un lecteur connecté à l'ordinateur via USB 2.0 à partir d'éléments standards: FPGA (100 ... 200 $), amplificateurs de courant (MAX9923FEUB - 200 ... 300 roubles), ADC (1 ... 2 mille roubles), etc ... Il faut développer uniquement une carte avec un plot de contact non standard (10x13) type LGA (Land Grid Array) pour l'amarrage avec une cellule de Flongle. Ou avec son homologue domestique simplifié contenant un plus petit nombre (16 ... 32) de puits de capteur.
 Touch Pad Cell Flongle
Touch Pad Cell FlongleRéduire la productivité de conception des cellules de 4 à 8 fois (jusqu'à 10 à 20 Mo) n'est pas essentiel pour résoudre de nombreux problèmes (détermination des agents pathogènes, typage HLA, établissement de la paternité, identification, etc.), mais il vous permet de collecter un séquenceur à nanopores sur des amplificateurs conventionnels courants picoampères. Et se passer de l'utilisation de puces spéciales à 512 canaux, dont les Oxfordiens ont investi des dizaines de millions de dollars dans le développement. Certes, chaque nanoreader multicanal doit contenir plus d'une douzaine d'amplificateurs de courant + ADC, mais cela peut encore coûter plusieurs fois moins que la combinaison propriétaire MinION + Flongle (> 200 mille roubles). Quant aux faibles performances du nanoreader, pour les utilisateurs russes qui n'ont pas d'ordinateurs décents (médecins, biologistes, biohackers, etc.), il peut même être utile.
Kilorider
Les amplificateurs de courant picoampères 512 canaux développés par les Oxfordiens sont contenus dans chaque cellule jetable pour le MinION. Les piles à déchets peuvent être jetées, mais il est préférable d'utiliser les puces qu'elles contiennent pour produire des analogues de Flongle caractérisés par une productivité accrue. Si ces puces sont équipées de contacts de surface, elles pourront fonctionner avec des cellules de contact contenant 2048 (512x4) puits de capteur.
La conception de cellules avec de telles LGA ne sera pas facile, mais vous devez tenir compte du fait que les exigences de contact ici sont moins strictes que les processeurs et les cartes mères, dans lesquelles un mauvais contact peut désactiver l'ensemble du système. Pour un séquenceur, l'inopérabilité de même la moitié des contacts cellulaires peut être considérée comme acceptable. La moitié restante est suffisante pour assurer des performances au niveau de 5 ... 10 Go.
Le développement logique de cette idée sera la combinaison de plusieurs lecteurs similaires dans un seul appareil. Cela vous permettra d'obtenir un séquenceur analogique GridION X5, conçu pour un travail simultané avec cinq (ou plus) cellules réutilisables de production nationale. Un tel séquenceur peut être demandé dans de nombreux laboratoires de diagnostic clinique.
 https://store.nanoporetech.com/devices
https://store.nanoporetech.com/devicesLes cellules de séquenceur jetables PromethION contiennent 9 000 puits de capteur sur puces. Et les puces elles-mêmes sont des amplificateurs à 3000 canaux de courants picoampères. Le flux d'informations qu'ils diffusent avec 24 (PromethION 24) ou 48 (PromethION 48) cellules d'information est si important qu'il ne peut pas être transféré sous forme brute vers un ordinateur externe ordinaire. Par conséquent, les séquenceurs de ce type doivent être équipés de leur propre supercalculateur. Mais si le séquenceur ne fonctionne qu'avec une seule cellule, il peut être connecté à un ordinateur portable de jeu classique avec un port Thunderbolt 3.0.
La modification des puces libres (éjectées) de l'ONT pour fonctionner avec des cellules de contact n'est pas un problème insoluble. Mais les séquenceurs basés sur l'utilisation de ces puces ne peuvent fonctionner qu'avec le logiciel ONT. Et les efforts qui devront être consacrés à la refonte de ces programmes peuvent annuler les avantages de l'utilisation de puces standard. Et développer de telles puces à partir de zéro est trop cher.
Mega Rider
Cette année, les tests bêta du séquenceur de nanopores d'AXBIO (USA) devraient commencer. Il utilise des puces avec un million de canaux d'amplification de courant produites au Japon par la société israélienne Tower Semiconductor Ltd ... Les consommables et la vente de ces séquenceurs seront assurés par la branche chinoise d'AXBIO.

Aux États-Unis et dans l'Union européenne, plus de 7 000 brevets sont consacrés au séquençage des nanopores, ce qui crée des risques financiers imprévisibles pour les entreprises locales qui tentent de gérer ces technologies. Apparemment, c'est la principale raison d'une aussi forte internationalisation de ce développement. Et cela permet aux entreprises et laboratoires russes de participer sans aucune restriction aux tests et à l'amélioration de la technologie AXBIO, ainsi qu'à la production (ou la collecte) de tels séquenceurs sur leur territoire.
Téraoctets de données brutes obtenues par un séquenceur nanoporeux à puces mégapixels (méga lecteur), il est souhaitable de filtrer. Et transférez sur l'ordinateur uniquement les lectures les plus longues et de la plus haute qualité. Cela améliorera non seulement la qualité du séquençage, mais réduira également les exigences pour l'ordinateur, qui est également important pour la production de masse et l'utilisation généralisée des séquenceurs génomiques. De plus, la productivité excessive des méga-lecteurs peut réduire la durée de séquençage du génome humain de 2 ... 3 jours (pour ONT) à plusieurs heures.
Somme de la technologie
Le succès de l'ONT a été facilité par l'organisation d'une optimisation en pipeline parallèle des cinq principaux éléments de la technologie de séquençage des nanopores - membranes, nanopores, «moteurs» (hélicases), conditions de séquençage et algorithmes de décodage pour les signaux de lecture.
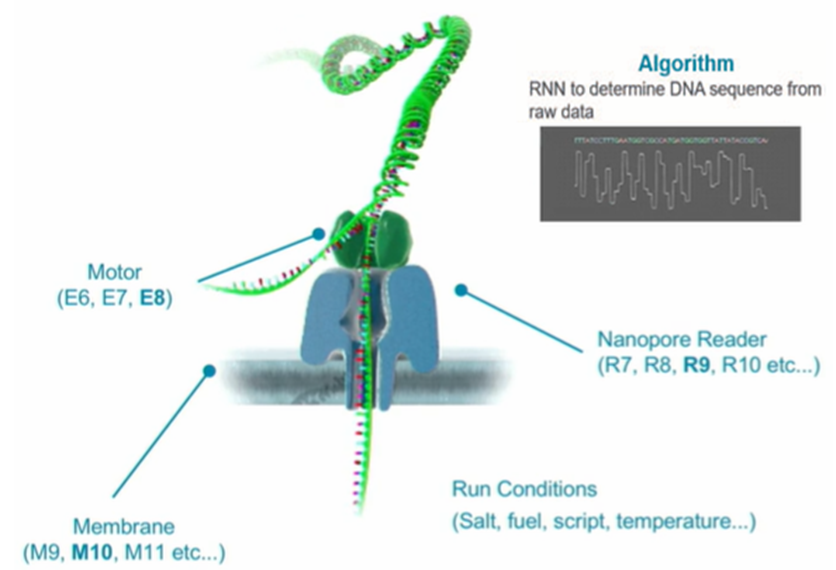 https://nanoporetech.com/resource-centre/videos/sub1000
https://nanoporetech.com/resource-centre/videos/sub1000La durée de conservation garantie des cellules pour le séquençage des nanopores dépend principalement de la stabilité de la membrane lipidique bicouche. Maintenant, c'est 6 semaines, bien que ces membranes soient généralement détruites en quelques jours. Pour augmenter leur durée de conservation, des additifs polymères hydrophobes, des copolymères séquencés tensioactifs, du cholestérol, des antioxydants, une réticulation d'acides gras, etc. sont utilisés, et de telles modifications peuvent interférer avec la formation de canaux ioniques. Par conséquent, il est très difficile d'obtenir une stabilité BLM.
Résoudre radicalement ce problème permet la formation de membranes dans les cellules de travail du séquenceur et l'introduction de nanopores dedans immédiatement avant utilisation. L'auto-préparation des cellules pour le travail offrira l'opportunité de leur régénération et réutilisation. Quant à la durée de conservation, pour les cellules sèches elle est quasiment illimitée, et les réactifs nécessaires à leur activation et à leur régénération peuvent être stockés pendant des mois.
Le goulot d'étranglement dans la technologie du séquençage des nanofils est en même temps le goulot d'étranglement du nanopore, dont dépend la qualité des informations lues. Par conséquent, l'une des tâches principales de l'ONT était la recherche d'une protéine membranaire, qui permet d'obtenir des résultats de qualité acceptable.
La première de ces protéines était le CsgG, une protéine de la membrane externe présente dans de nombreuses bactéries à Gram négatif. Neuf de ses sous-unités forment un canal ionique dans la membrane avec de larges vestibules d'entrée et de sortie et un goulot d'étranglement. Mais il n'a été possible d'obtenir des nanopores de qualité suffisamment élevée (R9, R9.4.1, R9.5.1) qu'après avoir dénombré et analysé des centaines de variantes CsgG modifiées. Cet été, des cellules avec les pores de la prochaine génération (R10 ou R10b) devraient apparaître en vente, avec une précision accrue dans la lecture des répétitions d'homopolymères.
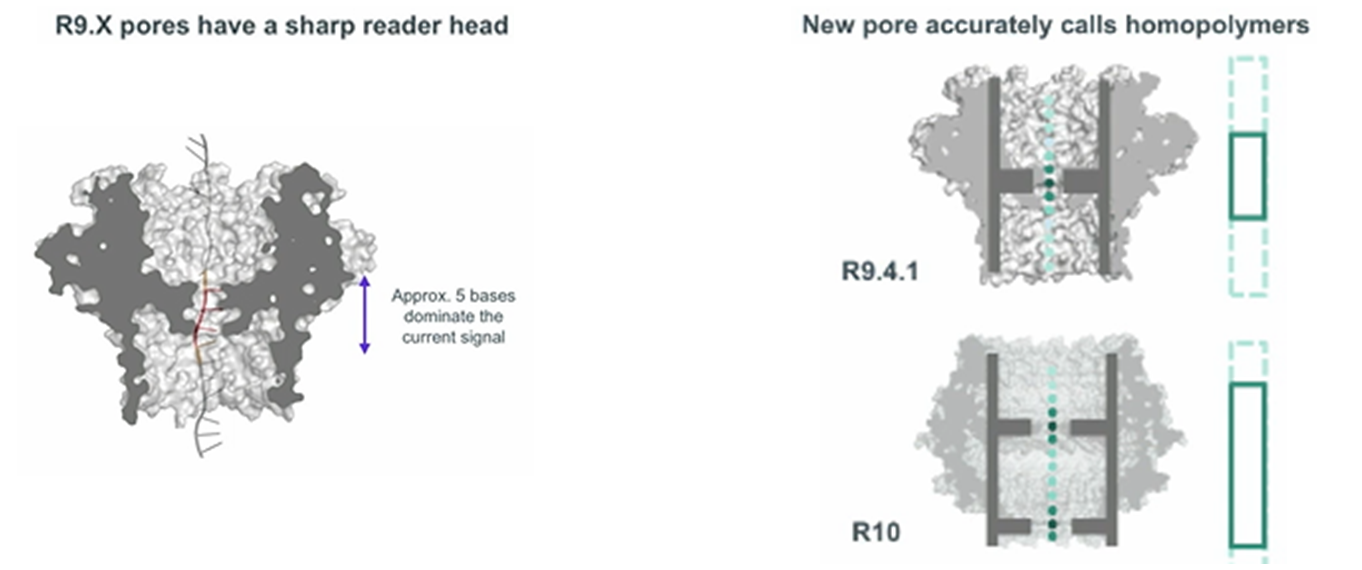 https://nanoporetech.com/about-us/news/london-calling-clive-brown-and-team-plenary
https://nanoporetech.com/about-us/news/london-calling-clive-brown-and-team-plenaryL'ONT est un composant obligatoire de la technologie de séquençage des nanopores de l'ONT, une hélicase, un moteur moléculaire qui déroule l'ADN double brin et inhibe la progression de l'ADN simple brin à travers un nanopore. La vitesse maximale de déroulement de l'ADN par un tel moteur atteint désormais 450 paires de nucléotides par seconde, bien que la vitesse des composants électroniques MinION lui permette d'être portée à 1000 nucléotides par seconde. Théoriquement, l'accélération du travail de l'hélicase peut doubler les performances de MinION, mais en pratique, cela peut conduire à un mauvais séquençage. Par conséquent, il est peu probable que des hélicases plus rapides et plus efficaces améliorent les performances des séquenceurs de nanopores tels que MinION et PromethION.
La qualité du séquençage dépend de nombreuses conditions - l'amplitude de la tension appliquée à la membrane, la conductivité électrique et la composition ionique du mélange réactionnel, la quantité et la nature des impuretés obstruant les pores, la capacité de nettoyer les pores obstrués en modifiant la polarité du potentiel, la structure des adaptateurs utilisés, etc., etc., etc. ... Par conséquent, nanopore le séquençage nécessite une optimisation minutieuse de tous ses paramètres et compositions de solutions de travail (conditions d'exécution), ce qui peut nuire à la qualité de la numérisation des séquences nucléotidiques awns. Par exemple, il a été récemment montré qu'une diminution de la concentration d'ATP clivé par l'hélicase diminue progressivement la précision des résultats de séquençage, et maintenant les Oxfordiens tentent d'éliminer ce problème en introduisant un système de régénération d'ATP dans le mélange réactionnel.
Le cinquième élément de la technologie de séquençage des nanopores est l'intelligence artificielle construite sur des réseaux de neurones récurrents (RNN) et sur l'utilisation de certains algorithmes et programmes spéciaux qui améliorent la reconnaissance des répétitions homogènes (Flip-flop, Medaka). En juillet, la prochaine version des programmes Guppi et MinKNOW devrait apparaître, dans laquelle les Oxfordiens promettent d'ajouter la capacité de déterminer les bases méthylées (5mC et 6mA). Ceci est important non seulement pour les études épigénomiques, mais aussi pour améliorer la reconnaissance des bases conventionnelles.
Les volumes importants d'informations lues par les séquenceurs de nanopores et la complexité de leur traitement imposent des exigences accrues sur le matériel de ces logiciels et systèmes matériels. Par conséquent, tous les ordinateurs ne conviennent pas pour connecter MinION, la dernière génération d'accélérateurs graphiques est installée dans GridION et PromethION, et Oxford Nanopore Technologies est devenu l'un des premiers acheteurs d'un grand lot de modules Jetson AGX Xavier (NVIDIA) conçus pour les appareils dotés d'intelligence artificielle.
Plus récemment, la technologie des nanopores n'était considérée que comme un complément utile à des technologies de séquençage de fluorescence et de semi-conducteurs plus précises. Mais une augmentation progressive de la précision de lecture de l'ADN, combinée à une grande longueur de séquences lisibles, lui a permis, sinon de devancer tous ses concurrents, de déterminer au moins le principal vecteur de développement des technologies de séquençage génomique.

Il peut prendre un certain temps pour maîtriser tous les éléments de la technologie de séquençage des nanopores, mais ce n'est pas le plus gros problème. Le principal problème est le manque de financement ciblé pour de tels développements intégrés en Russie. Certes, le programme fédéral scientifique et technique pour le développement des technologies génétiques pour 2019-2027 récemment approuvé (section «Orientations de mise en œuvre du programme») indique qu'à court terme (3 ... 6 ans), un prototype de dispositif de séquençage génomique haute performance sera développé. Mais trouver cette seule ligne sur 30 pages de texte n'est pas si facile. Plus facile à négocier avec les Chinois sur l'achat de séquenceurs AXBIO.
Encore plus simple est de trouver des artisans qui peuvent assembler le nanorider le plus simple "sur le genou". Et puis marcher d'une main tendue à la recherche d'investisseurs qui acceptent de financer le développement de la technologie domestique de séquençage des nanopores.