Je suis tombé sur
un article sur le blog de la société School of Data et j'ai décidé de vérifier les capacités de la bibliothèque Fast.ai sur le même jeu de données que celui mentionné dans l'article. Ici, vous ne trouverez pas d'arguments sur l'importance de diagnostiquer correctement et en temps opportun la pneumonie, si les radiologues seront nécessaires dans les conditions du développement technologique, si la prédiction d'un réseau neuronal peut être considérée comme un diagnostic médical, etc. L'objectif principal est de montrer que l'apprentissage automatique dans les bibliothèques modernes peut être assez simple (nécessite littéralement quelques lignes de code) et donne d'excellents résultats. Souvenons-nous du résultat de l'article (précision = 0,84, rappel = 0,96) et voyons ce qui se passe avec nous.
Nous prenons les données pour la formation
d'ici . Les données sont 5856 rayons X répartis en deux classes - avec ou sans signes de pneumonie. La tâche du réseau neuronal est de nous fournir un classificateur binaire de haute qualité d'images radiographiques pour déterminer les signes de pneumonie.
Nous commençons par importer les bibliothèques et certains paramètres standard:
%reload_ext autoreload %autoreload 2 %matplotlib inline from fastai.vision import * from fastai.metrics import error_rate import os
Ensuite, déterminez la taille du lot. Lors de l'apprentissage sur le GPU, il est important de le choisir de manière à ce que votre mémoire ne soit pas pleine. Si nécessaire, il peut être réduit de moitié.
bs = 64
Mise à jour importante:Comme indiqué à juste titre dans les commentaires ci-dessous, il est important de contrôler clairement les données sur lesquelles le modèle sera formé et sur lesquelles nous testerons son efficacité. Nous allons former le modèle dans les images dans les dossiers train et val, et valider dans les images dans le dossier test, similaire à ce qui a été fait
ici .
Nous déterminons les chemins d'accès à nos données
path = Path('storage/chest_xray') path.ls()
et vérifiez que tous les dossiers sont en place (le dossier val a été déplacé vers train):
Out: [PosixPath('storage/chest_xray/train'), PosixPath('storage/chest_xray/test')]
Nous préparons nos données pour le «chargement» dans le réseau neuronal. Il est important de noter que Fast.ai propose plusieurs méthodes pour faire correspondre le libellé de l'image. La méthode from_folder nous dit que les étiquettes doivent être tirées du nom du dossier dans lequel se trouve l'image.
Le paramètre de taille signifie que nous redimensionnons toutes les images à une taille de 299x299 (nos algorithmes fonctionnent avec des images carrées). La fonction get_transforms nous donne une augmentation d'image pour augmenter la quantité de données d'entraînement (nous laissons les paramètres par défaut ici).
np.random.seed(5) data = ImageDataBunch.from_folder(path, train = 'train', valid = 'test', size=299, bs=bs, ds_tfms=get_transforms()).normalize(imagenet_stats)
Regardons les données:
data.show_batch(rows=3, figsize=(6,6))
Pour vérifier, nous regardons quelles classes nous avons obtenues et quelle distribution quantitative d'images entre le train et la validation:
data.classes, data.c, len(data.train_ds), len(data.valid_ds)
Out: (['NORMAL', 'PNEUMONIA'], 2, 5232, 624)
Nous définissons un modèle de formation basé sur l'architecture Resnet50:
learn = cnn_learner(data, models.resnet50, metrics=error_rate)
et commencer à apprendre en 8 époques sur la base d'
une politique à cycle unique :
learn.fit_one_cycle(8)
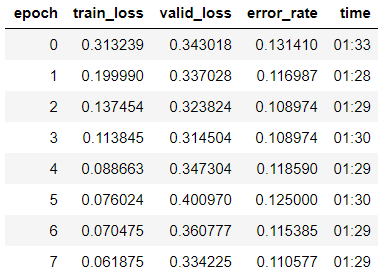
On voit que nous avons déjà obtenu une précision de 89% sur l'échantillon de validation. Nous allons noter les poids de notre modèle pour l'instant et essayer d'améliorer le résultat.
learn.save('step-1-50')
«Dégivrez» tout le modèle, car avant cela, nous avons entraîné le modèle uniquement sur le dernier groupe de couches, et les poids des autres ont été tirés du modèle pré-formé sur Imagenet et «gelé»:
learn.unfreeze()
Nous recherchons le taux d'apprentissage optimal pour continuer à apprendre:
learn.lr_find() learn.recorder.plot()
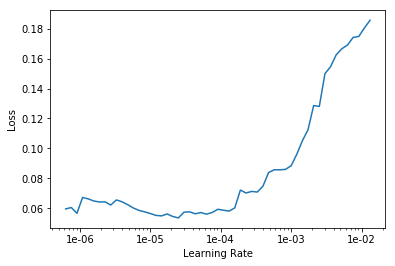
Nous commençons la formation pour 10 époques avec des taux d'apprentissage différents pour chaque groupe de couches.
learn.fit_one_cycle(10, max_lr=slice(1e-6, 1e-4))
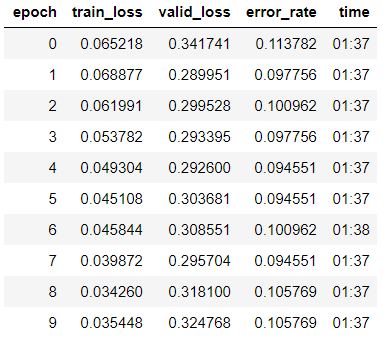
Nous constatons que la précision de notre modèle a légèrement augmenté à 89,4% dans l'échantillon de validation.
Nous notons les poids.
learn.save('step-2-50')
Construire une matrice de confusion:
interp = ClassificationInterpretation.from_learner(learn) interp.plot_confusion_matrix()
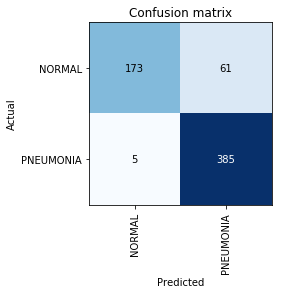
À ce stade, nous rappelons que le paramètre de précision seul est insuffisant, en particulier pour les classes déséquilibrées. Par exemple, si dans la vie réelle une pneumonie ne survient que chez 0,1% des personnes qui subissent un examen aux rayons X, le système peut simplement révéler l'absence de pneumonie dans tous les cas et sa précision sera au niveau de 99,9% avec une utilité absolument nulle.
C'est là que les mesures de précision et de rappel entrent en jeu:
- TP - vraie prédiction positive;
- TN - vraie prédiction négative;
- FP - prédiction de faux positifs;
- FN - Prédiction de faux négatifs.
On voit que le résultat que nous avons obtenu est même légèrement supérieur à celui mentionné dans l'article. Dans les travaux ultérieurs sur la tâche, il convient de rappeler que le rappel est un paramètre extrêmement important dans les problèmes médicaux, car Les fausses erreurs négatives sont les plus dangereuses du point de vue du diagnostic (ce qui signifie que nous pouvons simplement «ignorer» un diagnostic dangereux).