La partie précédente (sur la régression linéaire, la descente de gradient et comment tout cela fonctionne) -
habr.com/en/post/471458Dans cet article, je vais d'abord montrer la solution au problème de classification, comme on dit, «plumes», sans bibliothèques tierces pour SGD, LogLoss et calculer les gradients, puis en utilisant la bibliothèque PyTorch.
Objectif: pour deux caractéristiques catégorielles décrivant le jaune et la symétrie, déterminer à quelle classe (pomme ou poire) appartient l'objet (apprendre au modèle à classer les objets).
Pour commencer, téléchargez notre jeu de données:
import pandas as pd data = pd.read_csv("https://raw.githubusercontent.com/DLSchool/dlschool_old/master/materials/homeworks/hw04/data/apples_pears.csv") data.head(10)
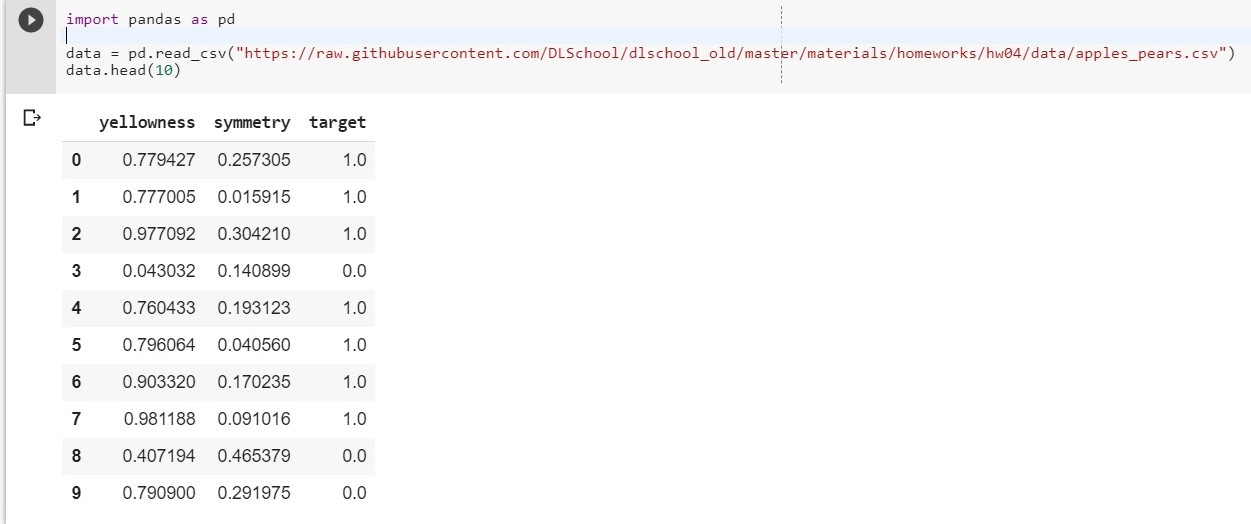
Soit: x1 - jaune, x2 - symétrie, y = targer
Nous composons la fonction y = w1 * x1 + w2 * x2 + w0
(w0 sera considéré comme le biais (eng. - biais))
Maintenant, notre tâche se réduit à trouver les poids w1, w2 et w0, qui décrivent le plus précisément la dépendance de y sur x1 et x2.
Nous utilisons la fonction de perte logarithmique:
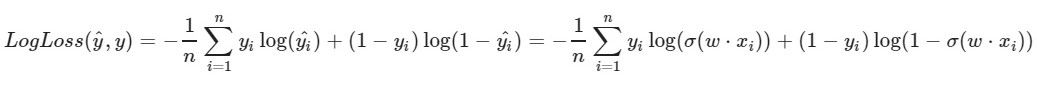
Le paramètre de gauche de la fonction est la prédiction avec les poids actuels w1, w2, w0
Le bon paramètre de la fonction est la valeur correcte (la classe est 0 ou 1)
σ (x) est la
fonction d'activation sigmoïde de x
log (x) - le logarithme naturel de x
Il est clair que plus la valeur de la fonction de perte est petite, mieux nous avons choisi les poids w1, w2, w0. Pour ce faire, choisissez une
descente de gradient stochastique .
Je note que la formule de LogLoss prendra un aspect différent étant donné que dans SGD, nous sélectionnons un élément et non une sélection entière (ou un sous-échantillon, comme dans le cas de la descente de gradient en mini-lot):
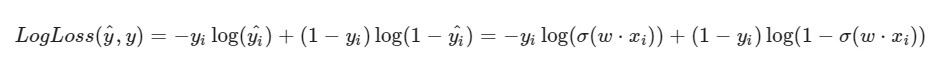 Progression de la solution:
Progression de la solution:Les poids initiaux w1, w2, w0 reçoivent des valeurs aléatoires
Nous prenons un certain i-ème objet de notre ensemble de données (par exemple, aléatoire), calculons LogLoss pour lui (avec nos w1, w2 et w0, auxquels nous avons initialement attribué des valeurs aléatoires), puis nous calculons les dérivées partielles pour chacun des poids w1, w2 et w0, puis mettez à jour chacun des poids.
Un peu de préparation: import pandas as pd import numpy as np X = data.iloc[:,:2].values
Réalisation: import random np.random.seed(62) w1 = np.random.randn(1) w2 = np.random.randn(1) w0 = np.random.randn(1) print(w1, w2, w0)
[0.49671415] [-0.1382643] [0.64768854]
[0,87991625] [-1,14098372] [0,22355905]
* _grad est la dérivée du poids correspondant. J'écrirai la formule générale:
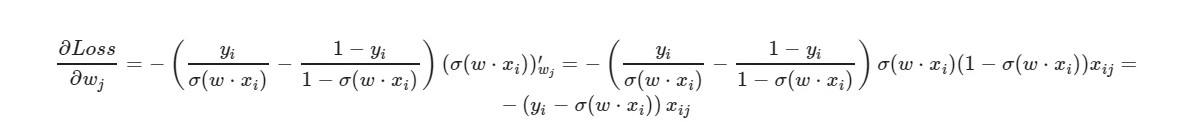
Pour le terme libre w0 - le facteur x est omis (pris égal à un).
En utilisant la formule finale de la dérivée, nous pouvons voir que nous n'avons pas besoin de calculer explicitement la fonction de perte (nous n'avons besoin que de dérivées partielles).
Vérifions le nombre d'objets de l'ensemble de formation que notre modèle donne les bonnes réponses et le nombre - les mauvaises.
i = 0 correct = 0 incorrect = 0 for item in y: if(np.around(x1[i] * w1 + x2[i] * w2 + w0) == item): correct += 1 else: incorrect += 1 i = i + 1 print(correct, incorrect)
925 75
np.around (x) - arrondit la valeur de x. Pour nous: si x> 0,5, alors la valeur est 1. Si x ≤ 0,5, alors la valeur est 0.
Et que ferons-nous si le nombre de caractéristiques de l'objet est de 5? 10? 100? Et nous aurons la quantité appropriée de poids (plus un pour le biais). Il est clair que travailler manuellement avec chaque poids, calculer les gradients pour cela n'est pas pratique.
Nous utiliserons la populaire bibliothèque PyTorch.
PyTorch = NumPy +
CUDA + Autograd (calcul automatique des gradients)
Implémentation de PyTorch:
import torch import numpy as np from torch.nn import Linear, Sigmoid def make_train_step(model, loss_fn, optimizer): def train_step(x, y): model.train() yhat = model(x) loss = loss_fn(yhat, y) loss.backward() optimizer.step() optimizer.zero_grad() return loss.item() return train_step X = torch.FloatTensor(data.iloc[:,:2].values) y = torch.FloatTensor(data['target'].values.reshape((-1, 1))) from torch import optim, nn neuron = torch.nn.Sequential( Linear(2, out_features=1), Sigmoid() ) print(neuron.state_dict()) lr = 0.1 n_epochs = 10000 loss_fn = nn.MSELoss(reduction="mean") optimizer = optim.SGD(neuron.parameters(), lr=lr) train_step = make_train_step(neuron, loss_fn, optimizer) for epoch in range(n_epochs): loss = train_step(X, y) print(neuron.state_dict()) print(loss)
OrderedDict ([('0.weight', tenseur ([[- 0.4148, -0.5838]]))), ('0.bias', tenseur ([0.5448])]]))
OrderedDict ([('0.weight', tenseur ([[[5.4915, -8.2156]]))), ('0.bias', tenseur ([- 1.1130])]]])
0,03930133953690529
Assez bonne perte sur l'échantillon d'essai.
Ici,
MSELoss est sélectionné comme fonction de perte.
Plus sur LinearEn bref: nous donnons 2 paramètres à l'entrée (nos x1 et x2 comme dans l'exemple précédent) et nous obtenons un paramètre (y) à la sortie, qui, à son tour, est alimenté à l'entrée de la fonction d'activation. Et puis ils sont déjà calculés: la valeur de la fonction d'erreur, les gradients. À la fin - les poids sont mis à jour.
Matériaux utilisés dans l'article