Un peu plus d'un an s'est écoulé depuis que le MIT a annoncé la sortie du langage polyvalent haute performance Julia . Depuis lors, la langue a gagné en popularité: elle est utilisée dans plus de 1500 universités (dans certaines, elle est enseignée comme première langue d'enseignement), et les domaines d'application couvrent les diagnostics médicaux et la planification de missions spatiales aux problèmes urgents tels que l' optimisation du trafic des autobus scolaires .
Il n'est pas difficile de deviner que l'un des principaux domaines d'activité de nombreux projets est l'apprentissage automatique, pour lequel Julia possède de nombreux outils puissants , et un projet assez intéressant a récemment été publié - General Probability Programming System «GEN» .
Aujourd'hui, nous ferons attention, comme son nom l'indique, au package Flux , qui fournit toute la puissance des réseaux de neurones. Nous allons essayer de passer du traitement et de la recherche d'ensembles d'images à un réseau neuronal formé pour obtenir un classificateur complet!
L'installation
Téléchargez le kit de distribution sur le site officiel et installez l'interpréteur Julia ( REPL ) sur votre ordinateur.
Pour que le gestionnaire de packages fonctionne correctement, les utilisateurs de Windows 7 / Windows Server 2012 doivent également installer:
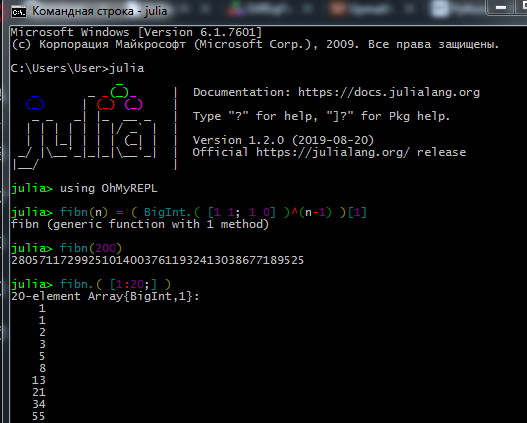
Le processus de travail dans REPL ressemble à ceci:
Les vrais utilisateurs de données et machines-lingologues préfèrent Jupyter . Ici, vous pouvez regarder l'installation, ainsi que trouver des leçons interactives pour l'auto-apprentissage avec des devoirs en russe (liens vers des tutoriels originaux et un guide de la langue là-bas).
Ici, vous pouvez voir comment travailler avec le bloc-notes Jupyter.
En cas de problèmes d'installation- La connexion ne peut pas être établie - vérifiez vos droits d'accès (avez-vous des restrictions sur l'écriture dans des dossiers sur C: \, connectez-vous en tant qu'administrateur ou démarrez Julia en mode administrateur), si vous utilisez un proxy, assurez-vous qu'il est configuré non seulement pour le navigateur
- Certains packages n'aiment pas l'alphabet cyrillique dans le chemin du fichier, donc à cause du nom d'utilisateur en russe, j'ai eu beaucoup de problèmes
- Si le package Interact n'affiche pas les résultats, vous avez peut-être installé WebIO de manière incorrecte, ce qui peut être résolu
- Pour que certains packages fonctionnent correctement sous Windows, les chemins d'accès à Julia et Jupyter doivent être entrés dans des variables d'environnement.
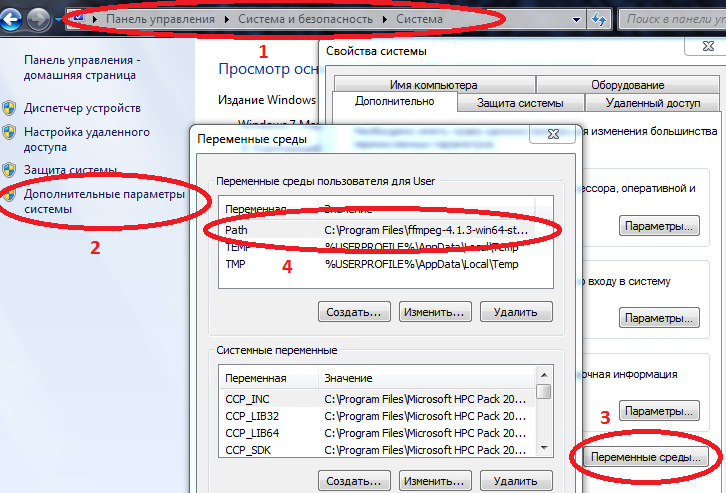
Ordinateur / Propriétés système / Paramètres système avancés / Variables d'environnement / Chemin d'accès (Créer sinon) et y ajouter le chemin d'accès à julia.exe
Exemple C: \ Users \ User \ AppData \ Local \ Julia-1.2.0 \ bin
si Path a déjà des valeurs, séparez-les par un point-virgule.
Maintenant, si vous conduisez julia dans la console de commande ( cmd ), l'interpréteur démarre.
Après avoir installé tout ce dont vous avez besoin, vous pouvez procéder au téléchargement des packages dont vous avez besoin aujourd'hui. Entrez les commandes dans REPL ou Jupyter
Code using Pkg pkgs = ["Plots", "TextParse", "CSV", "DataFrames", "ImageMagick", "Images", "Interact", "Flux"] for p in pkgs Pkg.add(p) end for p in pkgs Pkg.build(p) end
Après avoir appris les bases du langage (travailler avec des tableaux, créer des fonctions, télécharger des packages, tracer des graphiques), vous pouvez passer au matériel suivant.
Chargement et traitement des données
La collecte et l'organisation des données est un art distinct. En ce qui concerne Julia, le réseau a beaucoup de matériel obsolète, mais pour commencer, vous pouvez essayer le tutoriel ci - dessus , et pour une étude plus approfondie, lisez le livre Data Science with Julia (dans le domaine public)
Et aujourd'hui, peut-être, nous travaillerons avec des données déjà préparées: un ensemble de données à partir d'un grand nombre de photographies de fruits sous différents angles - qui voulait un fruit frais?


En fait, c'est la tâche - nous apprendrons au réseau neuronal à distinguer les pommes des bananes!
Tout d'abord, téléchargez des images de test:
using Images fnames = [ "data/10_100.jpg", "data/107_100.jpg", "data/yellow_apple_2.jpg", "data/8_100.jpg", "data/104_100.jpg", "data/3_100.jpg" ]

En quoi les objets sur les photos diffèrent-ils les uns des autres? D'abord par la forme, ensuite par la couleur, puis par les textures et autres attributs. L'analyse d'image est un sujet intéressant en soi, et la classification peut être faite non seulement par les neurones, mais aussi, par exemple, par des ondelettes . Nous commencerons par la couleur de signe la plus simple.
Comme vous le savez, les images sont stockées dans la mémoire de l'ordinateur sous forme de tableaux, dans notre cas ce sont des matrices, dont chaque cellule contient trois nombres, indiquant les quantités de couleurs rouge, vert et bleu dans chaque pixel de l'image. Voyons la quantité moyenne de chaque couleur dans ces images:
using Statistics: mean M1 = [ mean(float.(c.(img))) for c = [red,green,blue], img = fruits ] 3×6 Array{Float32,2}: 0.570278 0.652852 0.977111 0.835252 0.903998 0.842564 0.338118 0.468729 0.950773 0.806882 0.880692 0.755442 0.322406 0.379424 0.835212 0.707626 0.799643 0.761916
Nous examinons attentivement la première ligne - cela ne vous dérange pas? Une pomme jaune et des bananes sont plus rouges que des pommes de la variété Breburn! Comment ça?! Allez, fais des mines d'acide, peut-être que les écoliers lisent ce tutoriel, ou des élèves plus jeunes du Ballet and Tractor Institute. Par conséquent, nous essaierons d'éviter les omissions. Le fait est que l'arrière-plan de chaque image est blanc, et en notation RVB , il est représenté par les valeurs (1,1,1). Et comme il y a plus de 6 arrière-plans sur les 3 images de tableau de bord, plus la coloration des bananes et la pomme jaune contient également du rouge, il s'avère que les deux premières images perdent en rouge. Pour plus de clarté, nous avons divisé les images en couleurs de base:
function tweaking(img) R = colorview( RGB, red.(img),zeroarray,zeroarray ) G = colorview( RGB, zeroarray,green.(img),zeroarray ) B = colorview( RGB, zeroarray,zeroarray, blue.(img) ) [R; G; B] end tweaking( hcat(fruits...) )

Avez-vous déjà entendu le mot cryptique "base"? On peut donc dire que ces images sont disposées en RVB . Le plus noir - moins une certaine couleur, et comme nous nous y attendions, le fond avec sa richesse rend le calcul des moyennes bruyant. Supprimez-le.
function remove_background(img) mtrx = copy( channelview(img) ) for i = 1:size(mtrx, 2), j = 1:size(mtrx, 3) if reduce(&, mtrx[:,i,j] .> [0.8, 0.8, 0.8])

M3 = [ mean(float.(c.(img))) for c = [red,green,blue], img = greyfruits ] 3×6 Array{Float32,2}: 0.451008 0.532696 0.578967 0.527727 0.52849 0.500276 0.218805 0.348609 0.552679 0.499192 0.505136 0.412946 0.203528 0.260142 0.439354 0.400631 0.424784 0.419291
La différence dans la superficie occupée par chaque objet continue d'affecter, mais en général, on peut conclure que les bananes sont des pommes plus vertes ( et bleues ). Ce sera le critère d'évaluation, c'est-à-dire un signe. Jetons maintenant un œil au reste des images:
pth = "C:\\Users\\User\\Desktop\\Banana"
Pour chaque image, on neutralise la contribution de l'arrière-plan, on retrouve la quantité moyenne de chaque couleur, en se souvenant simultanément des tailles d'image ...
dataz = [] for fname in fnames img_i = load("$pth\\$fname") gbimg = remove_background(img_i) colorz = [ mean(c.(gbimg)) for c = [red,green,blue] ] inform = [size(gbimg, 1) size(gbimg, 2) colorz' ] push!(dataz, inform) end dataz
... et ensuite vous pouvez organiser nos données dans des structures pratiques pour le travail - trames de données:
using DataFrames, CSV banans = DataFrame( vcat(dataz...), [:height, :width, :red, :green, :blue] ) CSV.write("data/bananas.csv", banans)
apples = CSV.read("data/Apple_Braeburn.csv")
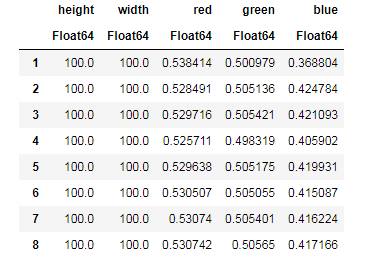
Desc = describe(apples, :all)
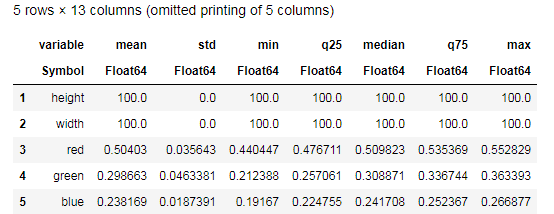
Essayez de comprendre les données fournies par la fonction describe() et comparez avec un tableau similaire pour les bananes. Eh bien, quel genre d'analyse de données peut être sans graphiques?
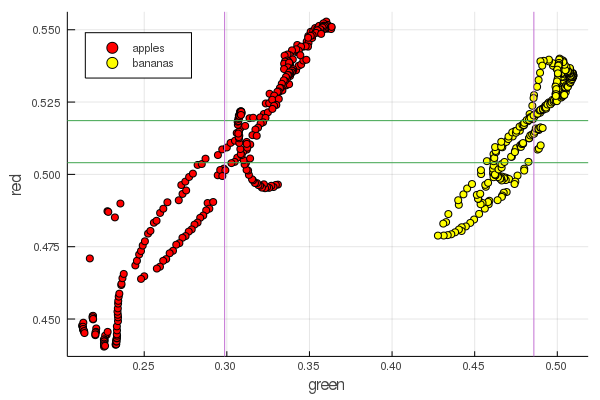
function plot2features(clr) x_apples = apples[:, :green] x_banans = banans[:, :green] y_apples = apples[:, clr] y_banans = banans[:, clr] scatter(x_apples, y_apples, lab = "apples", colour = :red) scatter!(x_banans, y_banans, lab = "bananas", legend = :topleft, colour = :yellow) hline!([mean(y_apples), mean(y_banans) ], lab = "" ) vline!([mean(x_apples), mean(x_banans) ], lab = "" ) xaxis!("green") yaxis!("$clr") end plot2features(:red)
plot2features(:blue)
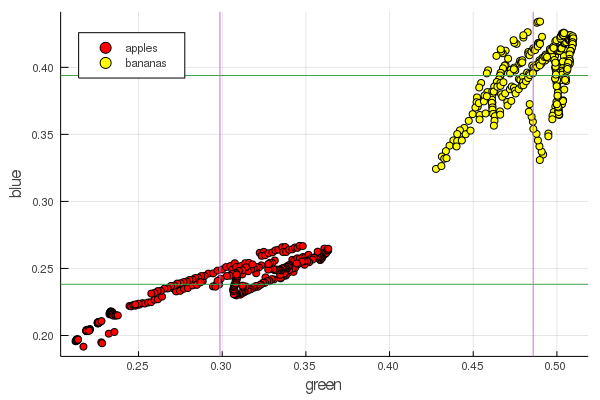
Le rouge mi-banane est très proche de la mi-pomme. Mais sur le deuxième graphique, l'isolement des fruits est immédiatement plus clairement tracé par deux caractéristiques de couleur à la fois. Les séparations peuvent être améliorées par une renormalisation correcte, par exemple, nos valeurs vertes passent de 0,2 à 0,55, et si vous effectuez la conversion
puis nous obtenons les données redimensionnées de [0,1], ce qui augmentera l'écart entre ces tas grappes de points.
Perceptron
La tâche de classification consiste à définir un modèle et à sélectionner des paramètres pour lesquels diverses données recevront de manière unique une évaluation de leur appartenance à une classe particulière. En termes simples, nous devons introduire une certaine fonction et définir ses paramètres de sorte qu'elle sépare nos pommes des bananes.
Le modèle le plus célèbre et le plus populaire à ces fins est le neurone artificiel McCulloch-Pitts, développé au début des années 40. Par la suite, Frank Rosenblatt a proposé un réseau neuronal formé - le perceptron. Il n'est pas difficile de trouver des explications complètes sur les réseaux de neurones, y compris sur cette ressource (par exemple les réseaux de neurones pour les débutants , l' utilisation des réseaux de neurones dans la reconnaissance d'images , les réseaux de neurones, les principes fondamentaux de fonctionnement, la variété et la topologie )
Sélection du sigmoïde comme fonction d'activation et réglage des sorties des objets classés (fruits) en fonction de ses sorties
sélectionner ces paramètres et de sorte que les valeurs de sortie du sigmoïde pour les données reçues correspondent à la notation ci-dessus
using Interact sigmo(x,w,b) = 1 / (1 + exp(-w*x+b)) r_apples, g_apples, b_apples = apples[:, :red], apples[:, :green], apples[:, :blue] r_banans, g_banans, b_banans = banans[:, :red], banans[:, :green], banans[:, :blue]; @manipulate for w in 10:1:60, b in -5:1:25 plot(x->sigmo(x,w,b), 0, 1, label="Model", legend = :topleft, lw=3) scatter!(g_apples[1:5], zeros(10), label="Apple", colour = :red) scatter!(g_banans[1:5], ones(10), label="Banana", colour = :yellow) end
foon(x) = sigmo(x,60,24) plot(foon, 0, 1, label="Model", legend = :topleft, lw=3) scatter!(foon, g_apples, label="Apple", colour = :red) scatter!(foon, g_banans, label="Banana", colour = :yellow) xaxis!("green")
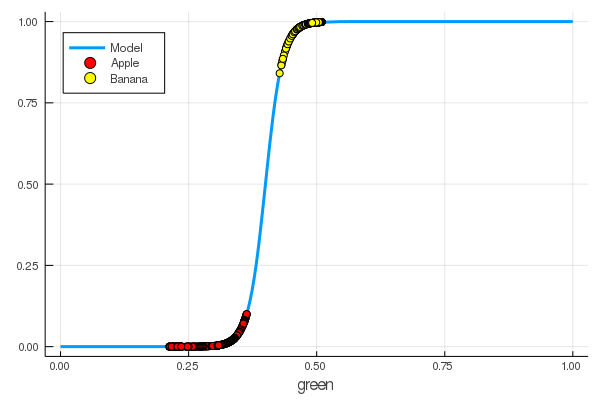
Nous avons appris manuellement à un neurone à distinguer les pommes des bananes par la quantité de vert!
Naturellement, la volonté d'automatiser ce processus. Nous introduisons la fonction de perte
Maintenant, le processus d'apprentissage consistera à minimiser cette fonction:
Code apples_mean_green = mean(g_apples) banans_mean_green = mean(g_banans) L(w, b) = (0 - sigmo(apples_mean_green,w,b))^2 + (1 - sigmo(banans_mean_green,w,b))^2 w_range = 10:0.5:30 b_range = 0:0.5:20 L_values = [L(w,b) for b in b_range, w in w_range] @manipulate for w in w_range, b in b_range p1 = surface(w_range, b_range, L_values, xlabel="b", ylabel="w", cam=(80,40), cbar=false, leg=false) scatter!(p1, [w], [b], [L(w,b)+1e-2], markersize=5, color = :blue) p2 = plot(x->sigmo(x,w,b), 0, 1, label="Model", legend = :topleft, lw=3) scatter!(p2, [apples_mean_green], [0.0], label="Apple", markersize=10) scatter!(p2, [banans_mean_green], [1.0], label="Banana", markersize=10, xlim=(0,1), ylim=(0,1)) plot(p1, p2, layout=(2,1)) end
Plus tôt, nous avons étudié les packages pour Julia qui permettent de résoudre des problèmes d'optimisation par différentes méthodes. Heureusement, l'essentiel est déjà dans l'environnement Flux!
Flux
using Flux
Tout d'abord, nous présentons les données pour la formation sous une forme digestible:
Y = [zeros(length(g_apples)); ones(length(g_banans)) ] |> permutedims X = [g_apples; g_banans] |> permutedims;
Ensuite dans l'ordre:
- Nous créons un ensemble de données de formation en combinant les données d'entrée avec les bonnes réponses concernant la classification de ces données
- Nous définissons les paramètres W et b par des matrices de valeurs aléatoires (il y a un signe à l'entrée et un à la sortie, donc les matrices ont une taille de 1 x 1 )
- En tant que modèle, nous définissons une couche dense - un perceptron avec une fonction d'activation sigmoïdale
- Nous définissons la fonction de perte - la somme des différences au carré (vous pouvez toujours utiliser le
Flux.crossentropy() plus populaire) - Comme méthode d'optimisation, nous choisissons la descente de gradient . Il faut un paramètre - la vitesse de descente
- Nous avons défini une fonction d'évaluation qui arrondira les valeurs des sorties du modèle et les comparera avec les bonnes réponses.
- Et imprimez les paramètres de notre modèle non formé
dataz = [(X, Y)] W = param(rand(1)) b = param(rand(1)) model = Dense(W, b, σ) loss(x, y) = mse(model(x), y) opt = Descent(0.1) accuracy(x, y) = mean( round.(model(x)) .== y ) params(model) Params([[0.3372841444115968] (tracked), [0.8430399003786011] (tracked)])
Voyons quelle est la sortie de la fonction de perte pour nos données.
loss(X, Y)
Et vérifiez les résultats de la fonction d'évaluation
accuracy(X, Y) 0.5
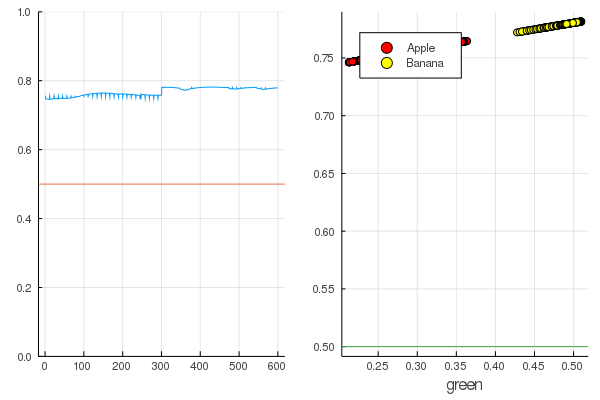
Le résultat est assez naturel - les sorties sont réparties assez uniformément et la moitié des données est correctement classée:
Code modeldataz(x) = x |> model |> data |> permutedims
modelX = modeldataz(X) modelapples = modeldataz(g_apples') modelbanans = modeldataz(g_banans') plot(modelX, legend = false) hline!([0.5]) p1 = yaxis!((0,1)) curv = [-1:0.01:1;]' |> modeldataz plot( [-1:0.01:1;], curv, label="Model", legend = :topleft, lw=3) scatter!(g_apples, modelapples, label="Apple", colour = :red) scatter!(g_banans, modelbanans, label="Banana",colour = :yellow) hline!([0.5], lab = "", legend = :topleft) p2 = xaxis!("green") plot(p1, p2)
Commençons: c'est assez simple. Il vous suffit de crier sur le réseau de neurones: «Train!», Tout en indiquant sur quoi s'entraîner et quoi minimiser, et elle terminera une session de formation. Par conséquent, nous la forcerons à sevrer tout comme il se doit, mais uniquement sans fanatisme, afin qu'il n'y ait pas de recyclage
for i in 1:7000 train!(loss, params(model), dataz, opt) end model.W, model.b ([9.578663260720564] (tracked), [-3.7540362587506464] (tracked))
Les pertes sont devenues beaucoup moins importantes:
loss(X, Y) 0.09152783090457564 (tracked)
Une note vaut mieux:
accuracy(X, Y) 1.0
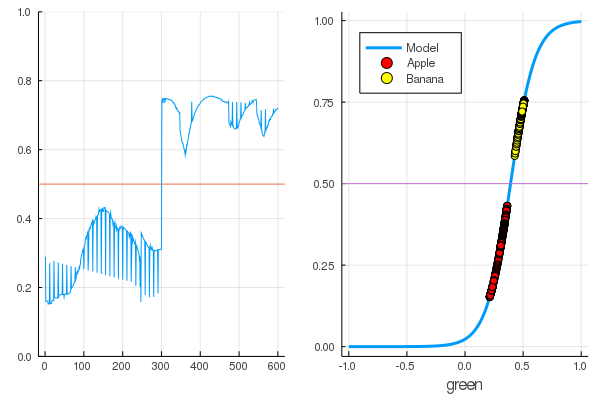
Les données sont divisées et une formation supplémentaire rendra le modèle plus vertical. Vérifiez le modèle formé sur la toute première série de fruits:
function classifier(img) gbimg = remove_background(img) greenmean = mean(float.(green.(gbimg))) answ = data( model( [ greenmean ]' ) )[1] fr = answ > 0.5 ? "Banana" : "Apple" "$fr $(round(200abs(0.5-answ)))%" end hcat(fruits...)

classifier.(fruits) 6-element Array{String,1}: "Apple 68.0%" "Apple 20.0%" "Banana 65.0%" "Banana 47.0%" "Banana 49.0%" "Banana 10.0%"
Une pomme jaune spécialement plantée, bien sûr, n'était pas reconnue correctement, et une banane rouge entrait à peine dans sa catégorie. Mais le neurone ne reçoit qu'un seul chiffre de l'image - la quantité moyenne de vert. Vous pouvez ajouter un autre signe, disons, la quantité de bleu, ce qui rendra le modèle un peu plus adaptable.
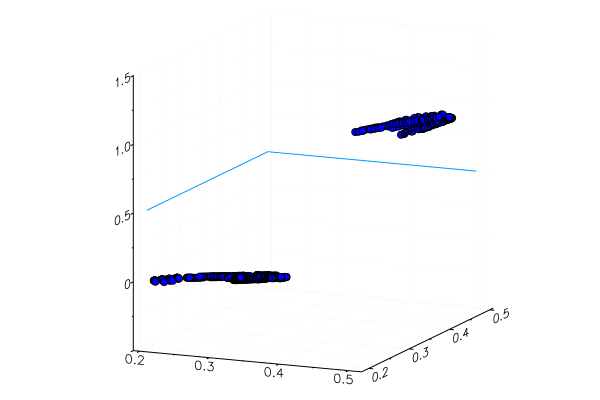
Ou vous pouvez utiliser non pas la représentation RVB, mais HSV (teinte, saturation, valeur), dans laquelle le canal de teinte contiendra des informations sur la couleur de l'image.
Toute la saveur des réseaux de neurones est qu'ils peuvent eux-mêmes distinguer des caractéristiques parfois peu évidentes (corrélation des couleurs, leur distribution, contours et courbes ...), et vous pouvez les aider à l'aide d'heuristiques et de techniques spéciales, qui transforment le travail avec les réseaux de neurones en vrai art.

Pour que le leadership ne se développe pas trop et faire une série d'articles donnons également un exemple de classification des images avec des nombres manuscrits, et le lecteur intéressé généralisera lui-même les connaissances acquises en images avec des fruits et créera son propre réseau de neurones, capable, disons, de marquer des objets dans des natures mortes!
Mnist
using Images using Flux, Flux.Data.MNIST, Statistics using Flux: onehotbatch, onecold, crossentropy, throttle using Base.Iterators: repeated
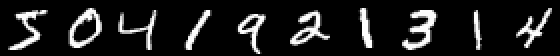
Un exemple est intéressant car il y a déjà dix sorties. Les soi-disant vecteurs One-hot sont utiles ici.
labels = MNIST.labels()
10×60000 Flux.OneHotMatrix{Array{Flux.OneHotVector,1}}: 0 1 0 0 0 0 0 0 0 0 0 0 0 … 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 1 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 1 0 1 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 … 0 0 0 0 0 1 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 0 1 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 1 0 0 0 0 0 0 0
Nous définissons une chaîne de neurones comme un modèle, l' entropie croisée sera une fonction de perte et Adam comme une méthode d'optimisation:
m = Chain( Dense(28^2, 32, relu), Dense(32, 10), softmax) loss(x, y) = crossentropy(m(x), y) accuracy(x, y) = mean(onecold(m(x)) .== onecold(y)) dataset = repeated((X, Y), 20) evalcb = () -> @show(loss(X, Y)) opt = ADAM()
Entraînez-vous en mode économe, mais imprimez les pertes toutes les 10 secondes:
for i = 1:10 Flux.train!(loss, params(m), dataset, opt, cb = throttle(evalcb, 10)) end
accuracy(X, Y) 0.64545
Et vérifiez les données non utilisées dans la formation
Les réseaux de neurones sur Julia sont simples et très excitants! Même s'il n'est pas nécessaire de rechercher les connexions de votre domaine d'activité avec l'apprentissage automatique, vous devriez au moins ressentir cette curiosité, qui est criée sous tous les angles, et les outils ne manqueront pas!
Chaleur CPU modérée!