Si vous lisez la formation sur les auto-encodeurs sur le site keras.io, alors l'un des premiers messages il y a quelque chose comme ça: dans la pratique, les auto-encodeurs ne sont presque jamais utilisés, mais ils sont souvent évoqués dans les formations et les gens viennent, nous avons donc décidé d'écrire notre propre tutoriel à leur sujet:
Leur principale prétention à la gloire vient de leur présence dans de nombreux cours d'initiation à l'apprentissage automatique disponibles en ligne. En conséquence, beaucoup de nouveaux arrivants sur le terrain adorent les auto-encodeurs et n'en ont jamais assez. C'est la raison pour laquelle ce tutoriel existe!
Néanmoins, l'une des tâches pratiques pour lesquelles elles peuvent s'appliquer à soi-même est la recherche d'anomalies, et j'en avais personnellement besoin dans le cadre du projet du soir.
Sur Internet, il existe de nombreux tutoriels sur les auto-encodeurs, pourquoi en écrire un de plus? Eh bien, pour être honnête, il y avait plusieurs raisons à cela:
- Il y avait le sentiment qu'en fait les tutoriels étaient d'environ 3 ou 4, tout le reste a été réécrit avec leurs propres mots;
- Presque tout - sur le MNIST'e qui souffre depuis longtemps avec des images 28x28;
- À mon humble avis - ils ne développent pas une intuition sur la façon dont tout cela devrait fonctionner, mais proposent simplement de répéter;
- Et le facteur le plus important - personnellement, lorsque j'ai remplacé MNIST par mon propre ensemble de données - tout a stupidement cessé de fonctionner .
Ce qui suit décrit mon chemin sur lequel les cônes sont farcis. Si vous prenez l'un des modèles plats proposés (non convolutionnels) de la masse des tutoriels et que vous les collez stupidement, alors, rien, étonnamment, ne fonctionne. Le but de l'article est de comprendre pourquoi et, il me semble, d'obtenir une sorte de compréhension intuitive de la façon dont tout cela fonctionne.
Je ne suis pas un spécialiste de l'apprentissage automatique et j'utilise les approches auxquelles je suis habitué dans le travail quotidien. Pour les scientifiques de données expérimentés, cet article sera probablement complètement sauvage, mais pour les débutants, il me semble, quelque chose de nouveau pourrait surgir.
quel genre de projetEn bref sur le projet, bien que l'article ne parle pas de lui. Il y a un récepteur ADS-B, il capte les données des avions qui volent et les écrit, les avions, les coordonnées à la base. Parfois, les avions se comportent de façon inhabituelle - ils tournent autour pour brûler du carburant avant l'atterrissage, ou tout simplement des vols privés survolent des itinéraires standard (couloirs). Il est intéressant d'isoler d'environ un millier d'avions par jour ceux qui ne se sont pas comportés comme les autres. J'admets pleinement que les écarts de base peuvent être calculés plus facilement, mais j'étais intéressé à essayer la magie réseaux de neurones.
Commençons. J'ai un ensemble de 4000 images en noir et blanc 64x64 pixels, cela ressemble à ceci:

Juste quelques lignes sur fond noir, et dans l'image 64x64 environ 2% des points sont remplis. Si vous regardez beaucoup d'images, alors, bien sûr, il s'avère que la plupart des lignes sont assez similaires.
Je n'entrerai pas dans les détails de la façon dont l'ensemble de données a été chargé, traité, car le but de l'article, encore une fois, n'est pas celui-ci. Montrez simplement un morceau de code effrayant.
Voici, par exemple, le premier modèle proposé avec keras.io, sur lequel ils ont travaillé et formé sur mnist:
Dans mon cas, le modèle est défini comme ceci:
model = tf.keras.Sequential() model.add(tf.keras.Input(shape=(64,64,1))) model.add(tf.keras.layers.Flatten()) model.add(tf.keras.layers.Dense(64*64/10, activation='relu')) model.add(tf.keras.layers.Dense(64*64, activation="sigmoid")) model.add(tf.keras.layers.Reshape(target_shape=(64,64,1)))
Il y a de légères différences que j'aplatis et remodèle directement dans le modèle, et que je «compresse» non pas 25 fois, mais seulement 10. Cela ne devrait rien affecter.
En tant que fonction de perte - erreur quadratique moyenne, l'optimiseur n'est pas fondamental, laissez Adam. Ci-après, nous formons 20 époques, 100 pas par époque.
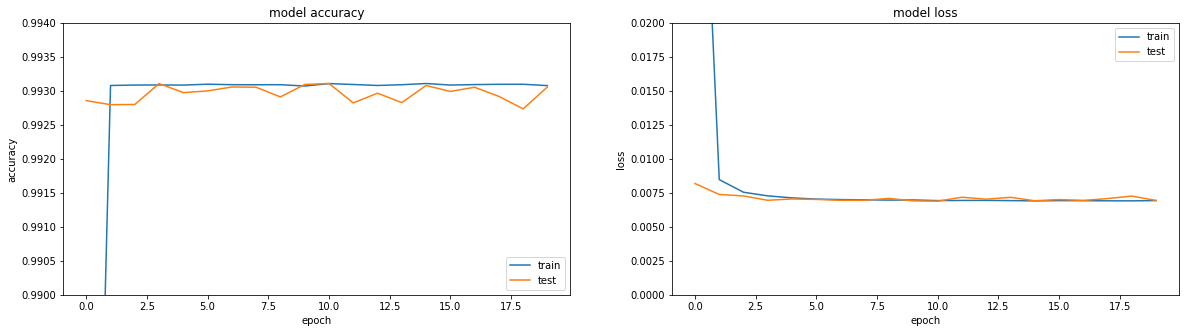
Si vous regardez les métriques - tout est en feu. Précision == 0,993. Si vous regardez les horaires de formation - tout est un peu plus triste, nous atteignons un plateau dans la région de la troisième ère.
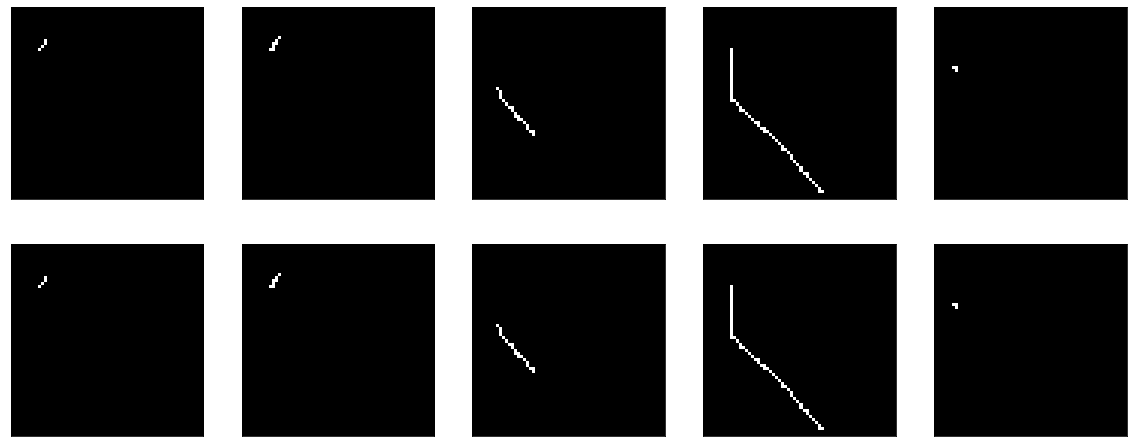
Eh bien, si vous regardez directement le résultat de l'encodeur, vous obtenez une image généralement triste (l'original est en haut et le résultat de l'encodage-décodage est ci-dessous):

En général, lorsque vous essayez de comprendre pourquoi quelque chose ne fonctionne pas, c'est une assez bonne approche pour diviser toutes les fonctionnalités en gros blocs et vérifier chacun d'eux isolément. Alors faisons-le.
Dans l'original du tutoriel - des données plates sont fournies à l'entrée du modèle et elles sont prises à la sortie. Pourquoi ne pas vérifier mes actions sur aplatir et remodeler. Voici un tel modèle sans opération:
model = tf.keras.Sequential() model.add(tf.keras.Input(shape=(64,64,1))) model.add(tf.keras.layers.Flatten()) model.add(tf.keras.layers.Reshape(target_shape=(64,64,1)))
Résultat:
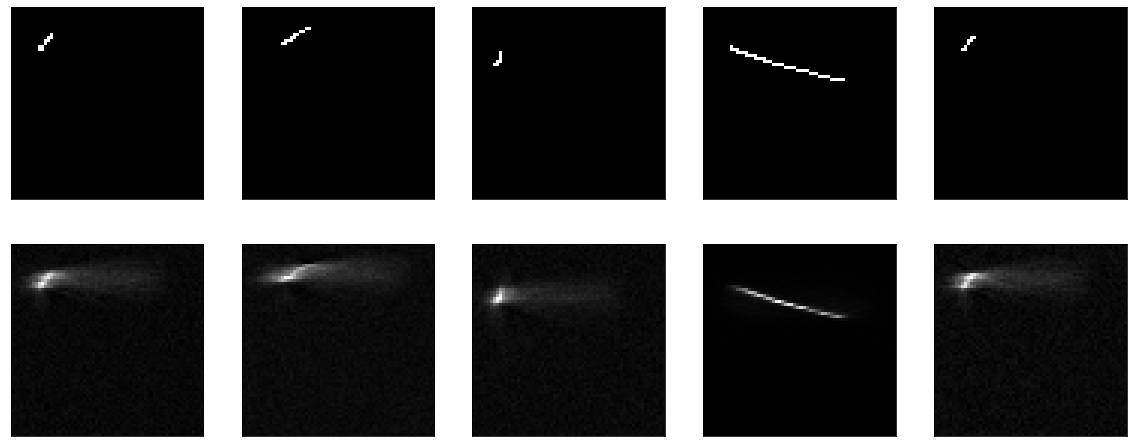
Il n'y a rien à enseigner ici. Eh bien, en même temps, cela a prouvé que ma fonction de visualisation fonctionne également.
Ensuite, essayez de rendre le modèle non-op, mais aussi stupide que possible - il suffit de couper la couche de compression, de laisser une couche de la taille de l'entrée. Comme ils le disent dans tous les tutoriels, disent-ils, il est très important que votre modèle apprenne des fonctionnalités, et pas seulement une fonction d'identité. Eh bien, c'est exactement ce que nous allons essayer d'obtenir, passons simplement l'image résultante à la sortie.
model = tf.keras.Sequential() model.add(tf.keras.Input(shape=(64,64,1))) model.add(tf.keras.layers.Flatten()) model.add(tf.keras.layers.Dense(64*64, activation="sigmoid")) model.add(tf.keras.layers.Reshape(target_shape=(64,64,1)))
Elle apprend quelque chose, une précision == 0.995 et encore une fois elle tombe sur un plateau.
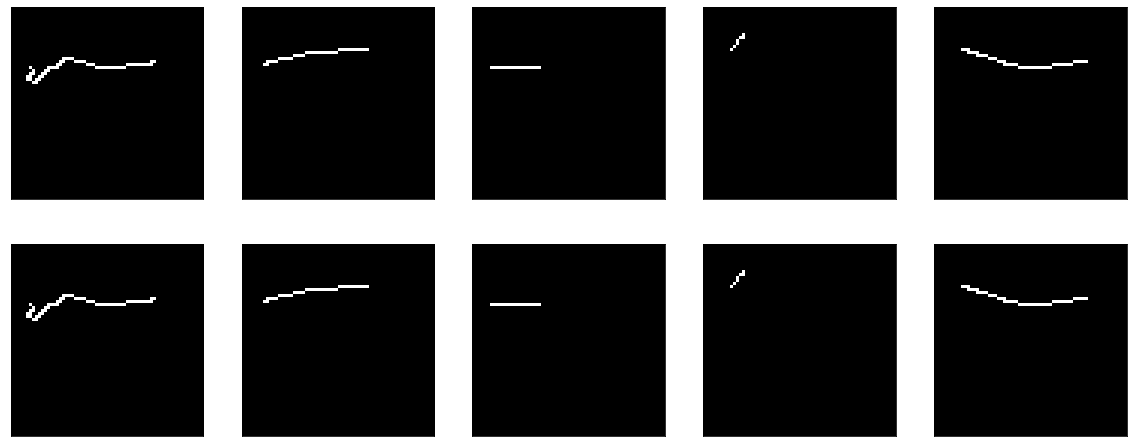
Mais, en général, il est clair que cela ne fonctionne pas très bien. Quoi qu'il en soit - quoi apprendre là-bas, passez l'entrée de la sortie et c'est tout.
Si vous lisez la documentation des keras sur les couches denses, elle décrit ce qu'elles font: output = activation(dot(input, kernel) + bias)
Pour que la sortie coïncide avec l'entrée, deux choses simples suffisent - biais = 0 et noyau - la matrice d'identité (il est important de ne pas laisser la matrice remplie d'unités ici - ce sont des choses très différentes). Heureusement, ceci et cela peuvent être faits assez facilement à partir de la documentation pour le même Dense .
model = tf.keras.Sequential() model.add(tf.keras.Input(shape=(64,64,1))) model.add(tf.keras.layers.Flatten()) model.add(tf.keras.layers.Dense(64*64, activation = "sigmoid", use_bias=False, kernel_initializer = tf.keras.initializers.Identity())) model.add(tf.keras.layers.Reshape(target_shape=(64,64,1)))
Parce que nous réglons le poids tout de suite, alors vous ne pouvez rien apprendre - tout de suite c'est bien:
Mais si vous commencez à vous entraîner, cela commence, à première vue, de manière surprenante - le modèle commence avec une précision == 1.0, mais il tombe rapidement.
Évaluez le résultat avant l'entraînement: 8/Unknown - 1s 140ms/step - loss: 0.2488 - accuracy: 1.0000[0.24875330179929733, 1.0] . Formation:
Epoch 1/20 100/100 [==============================] - 6s 56ms/step - loss: 0.1589 - accuracy: 0.9990 - val_loss: 0.0944 - val_accuracy: 0.9967 Epoch 2/20 100/100 [==============================] - 5s 51ms/step - loss: 0.0836 - accuracy: 0.9964 - val_loss: 0.0624 - val_accuracy: 0.9958 Epoch 3/20 100/100 [==============================] - 5s 50ms/step - loss: 0.0633 - accuracy: 0.9961 - val_loss: 0.0470 - val_accuracy: 0.9958 Epoch 4/20 100/100 [==============================] - 5s 48ms/step - loss: 0.0520 - accuracy: 0.9961 - val_loss: 0.0423 - val_accuracy: 0.9961 Epoch 5/20 100/100 [==============================] - 5s 48ms/step - loss: 0.0457 - accuracy: 0.9962 - val_loss: 0.0357 - val_accuracy: 0.9962
Oui, et ce n'est pas très clair, nous avons déjà un modèle idéal - l'image sort 1 en 1, et la perte (erreur quadratique moyenne) montre presque 0,25.
Soit dit en passant, c'est une question fréquente sur les forums - la perte diminue, mais la précision n'augmente pas, comment cela peut-il être?
Ici, il convient de rappeler une fois de plus la définition de la couche dense: output = activation(dot(input, kernel) + bias) et le mot activation qui y est mentionné, que j'ai si bien ignoré ci-dessus. Avec des poids de la matrice d'identité et sans biais, nous obtenons output = activation(input) .
En fait, la fonction d'activation dans notre code source est déjà indiquée, sigmoïde, je l'ai copiée assez bêtement et c'est tout. Et dans les tutoriels, il est conseillé de l'utiliser partout. Mais vous devez le comprendre.
Pour commencer, vous pouvez lire dans la documentation ce qu'ils en disent: The sigmoid activation: (1.0 / (1.0 + exp(-x))) . Personnellement, cela ne me dit rien, car je ne suis pas fantôme une fois pour construire de tels graphiques dans ma tête.
Mais vous pouvez construire avec des stylos:
import matplotlib.ticker as plticker range_tensor = tf.range(-4, 4, 0.01, dtype=tf.float32) fig, ax = plt.subplots(1,1) plt.plot(range_tensor.numpy(), tf.keras.activations.sigmoid(range_tensor).numpy()) ax.grid(which='major', linestyle='-', linewidth='0.5', color='red') ax.grid(which='minor', linestyle=':', linewidth='0.5', color='black') ax.yaxis.set_major_locator(plticker.MultipleLocator(base=0.5) ) plt.minorticks_on()
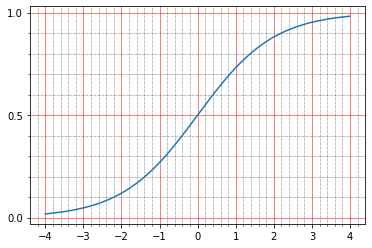
Et ici, il devient clair qu'à zéro, le sigmoïde prend la valeur 0,5, et dans l'unité - environ 0,73. Et les points que nous avons sont noirs (0,0) ou blancs (1,0). Il s'avère donc que l'erreur quadratique moyenne de la fonction d'identité reste non nulle.
Vous pouvez même regarder les stylos, voici une ligne de l'image résultante:
array([0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.7310586, 0.7310586, 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 , 0.5 ], dtype=float32)
Et c'est tout, en fait, très cool, car plusieurs questions apparaissent en même temps:
- pourquoi cela n'était-il pas visible dans la visualisation ci-dessus?
- pourquoi alors précision == 1.0, car les images originales sont 0 et 1.
Avec la visualisation, tout est étonnamment simple. Pour afficher les images, j'ai utilisé matplotlib: plt.imshow(res_imgs[i][:, :, 0]) . Et, comme d'habitude, si vous allez dans la documentation, tout y sera écrit: l' The Normalize instance used to scale scalar data to the [0, 1] range before mapping to colors using cmap. By default, a linear scaling mapping the lowest value to 0 and the highest to 1 is used. The Normalize instance used to scale scalar data to the [0, 1] range before mapping to colors using cmap. By default, a linear scaling mapping the lowest value to 0 and the highest to 1 is used. C'est-à-dire la bibliothèque a soigneusement normalisé mes 0,5 et 0,73 dans la plage de 0 à 1. Modifiez le code:
plt.imshow(res_imgs[i][:, :, 0], norm=matplotlib.colors.Normalize(0.0, 1.0))

Et voici la question avec précision. Pour commencer - par habitude, nous allons dans la documentation, lisons pour tf.keras.metrics.Accuracy et là, il semble qu'ils écrivent compréhensible:
For example, if y_true is [1, 2, 3, 4] and y_pred is [0, 2, 3, 4] then the accuracy is 3/4 or .75.
Mais dans ce cas, notre précision aurait dû être 0. En conséquence, je me suis enterré dans la source et c'est assez clair pour moi:
When you pass the strings 'accuracy' or 'acc', we convert this to one of `tf.keras.metrics.BinaryAccuracy`, `tf.keras.metrics.CategoricalAccuracy`, `tf.keras.metrics.SparseCategoricalAccuracy` based on the loss function used and the model output shape. We do a similar conversion for the strings 'crossentropy' and 'ce' as well.
De plus, dans la documentation sur le site pour une raison quelconque, ce paragraphe ne se trouve pas dans la description de .compile .
Voici un morceau de code de https://github.com/tensorflow/tensorflow/blob/66c48046f169f3565d12e5fea263f6d731f9bfd2/tensorflow/python/keras/engine/compile_utils.py
y_t_rank = len(y_t.shape.as_list()) y_p_rank = len(y_p.shape.as_list()) y_t_last_dim = y_t.shape.as_list()[-1] y_p_last_dim = y_p.shape.as_list()[-1] is_binary = y_p_last_dim == 1 is_sparse_categorical = ( y_t_rank < y_p_rank or y_t_last_dim == 1 and y_p_last_dim > 1) if metric in ['accuracy', 'acc']: if is_binary: metric_obj = metrics_mod.binary_accuracy elif is_sparse_categorical: metric_obj = metrics_mod.sparse_categorical_accuracy else: metric_obj = metrics_mod.categorical_accuracy
y_t est y_true, ou la sortie attendue, y_p est y_predicted, ou le résultat prévu.
Nous avons le format de données: shape=(64,64,1) , il s'avère donc que la précision est considérée comme binary_accuracy. Intérêt pour la façon dont il est considéré:
def binary_accuracy(y_true, y_pred, threshold=0.5): threshold = math_ops.cast(threshold, y_pred.dtype) y_pred = math_ops.cast(y_pred > threshold, y_pred.dtype) return K.mean(math_ops.equal(y_true, y_pred), axis=-1)
C'est drôle qu'ici, nous avons juste de la chance - par défaut, tout est considéré comme une unité supérieure à 0,5, et 0,5 et inférieure - zéro. La précision ressort donc à cent pour cent pour notre modèle d'identité, bien qu'en réalité les chiffres ne soient pas du tout les mêmes. Eh bien, il est clair que si nous le voulons vraiment, nous pouvons corriger le seuil et réduire la précision à zéro, par exemple, seulement ce n'est pas vraiment nécessaire. Il s'agit d'une mesure, elle n'affecte pas la formation, il vous suffit de comprendre que vous pouvez la calculer de mille manières différentes et obtenir des indicateurs complètement différents. À titre d'exemple, vous pouvez extraire diverses mesures avec des stylos et leur transférer nos données:
m = tf.keras.metrics.BinaryAccuracy() m.update_state(x_batch, res_imgs) print(m.result().numpy())
Nous donnera 1.0 .
Et ici
m = tf.keras.metrics.Accuracy() m.update_state(x_batch, res_imgs) print(m.result().numpy())
Nous donnera 0.0 sur les mêmes données.
Soit dit en passant, le même morceau de code peut être utilisé pour jouer avec les fonctions de perte et comprendre comment elles fonctionnent. Si vous lisez les tutoriels sur les auto-encodeurs, ils suggèrent essentiellement d'utiliser l'une des deux fonctions de perte: soit l'erreur quadratique moyenne, soit «binary_crossentropy». Vous pouvez également les regarder en même temps.
Je vous rappelle que pour mse j'ai déjà donné des modèles d' evaluate :
8/Unknown - 2s 221ms/step - loss: 0.2488 - accuracy: 1.0000[0.24876083992421627, 1.0]
C'est-à-dire perte == 0,2448. Voyons pourquoi c'est. Il me semble personnellement que c'est le plus simple et le plus compréhensible: la différence entre y_true et y_predict est soustraite pixel par pixel, chaque résultat est carré, puis la moyenne est recherchée.
tf.keras.backend.mean(tf.math.squared_difference(x_batch[0], res_imgs[0]))
Et en sortie:
<tf.Tensor: shape=(), dtype=float32, numpy=0.24826494>
Ici, l'intuition est très simple - la majorité des pixels vides, le modèle en produit 0,5, ils obtiennent 0,25 - de différence au carré pour eux.
Avec crossenttrtopy binaire, les choses sont un peu plus compliquées, et il y a des articles entiers sur la façon dont cela fonctionne, mais personnellement, il m'a toujours été plus facile de lire les sources, et là, ça ressemble à ceci:
if from_logits: return nn.sigmoid_cross_entropy_with_logits(labels=target, logits=output) if not isinstance(output, (ops.EagerTensor, variables_module.Variable)): output = _backtrack_identity(output) if output.op.type == 'Sigmoid':
Pour être honnête, j'ai creusé la tête sur ces quelques lignes de code pendant très longtemps. Premièrement, il est immédiatement clair que deux implémentations peuvent fonctionner: soit sigmoid_cross_entropy_with_logits sera appelé, soit la dernière paire de lignes fonctionnera. La différence est que sigmoid_cross_entropy_with_logits fonctionne avec les logits (comme son nom l'indique, doh), et le code principal fonctionne avec les probabilités.
Qui sont les logits? Si vous lisez un million d'articles différents sur le sujet, ils mentionneront des définitions mathématiques, des formules, autre chose. En pratique, tout semble étonnamment simple (corrigez-moi si je me trompe). La sortie brute de la prédiction est des logits. Eh bien, ou log-odds, les cotes logarithmiques qui sont mesurées en log istiques sur ses - perroquets logistiques.
Il y a une petite digression - pourquoi y a-t-il des logarithmesLes chances sont le rapport du nombre d'événements dont nous avons besoin au nombre d'événements dont nous n'avons pas besoin (contrairement à la probabilité, qui est le rapport des événements dont nous avons besoin au nombre de tous les événements en général). Par exemple - le nombre de victoires de notre équipe au nombre de ses défaites. Et il y a un problème. Poursuivant l'exemple avec les victoires des équipes, notre équipe peut être mi-perdante et avoir une chance de gagner 1/2 (un à deux), et peut-être extrêmement perdante - et avoir une chance de gagner 1/100. Et dans la direction opposée - moyennement raide et 2/1, plus raide que les plus hautes montagnes - puis 100/1. Et il s'avère que toute la gamme des équipes perdantes est décrite par des nombres de 0 à 1, et des équipes sympas - de 1 à l'infini. Par conséquent, il n'est pas pratique de comparer, il n'y a pas de symétrie, travailler avec cela en général est gênant pour tout le monde, les mathématiques sont laides. Et si vous prenez le logarithme des cotes, alors tout devient symétrique:
ln(1/2) == -0.69 ln(2/1) == 0.69 ln(1/100) == -4.6 ln(100/1) == 4.6
Dans le cas du tensorflow, cela est plutôt arbitraire, car, à proprement parler, la sortie de la couche n'est mathématiquement pas des cotes logarithmiques, mais elle est déjà acceptée. Si la valeur brute est de -∞ à + ∞ - alors logits. Ils peuvent ensuite être convertis en probabilités. Il y a deux options pour cela: softmax et son cas spécial, sigmoid. Softmax - Prenez un vecteur de logits et convertissez-les en un vecteur de probabilités, et même de sorte que la somme de la probabilité de tous les événements qu'il contient se révèle être 1. Sigmoid (dans le cas de tf) prend également un vecteur de logits, mais convertit chacun d'eux en probabilités séparément, indépendamment du reste.
Vous pouvez le voir de cette façon. Il y a des tâches de classification multi-étiquettes, il y a des tâches de classification multi-classes. Multiclasse - c'est si vous avez besoin de déterminer les pommes dans l'image ou les oranges, et peut-être même les ananas. Et multilabel, c'est quand il peut y avoir un vase de fruits sur la photo et vous devez dire qu'il y a des pommes et des oranges dessus, mais qu'il n'y a pas d'ananas. Si nous voulons une multiclasse - nous avons besoin de softmax, si nous voulons une multi-étiquette - nous avons besoin de sigmoid.
Ici, nous avons le cas du multilabel - il est nécessaire que chaque pixel individuel (classe) dise s'il est installé.
Revenons au tensorflow et pourquoi dans la crossentropie binaire (au moins dans les autres fonctions de crossentropie c'est à peu près la même), il y a deux branches globales. La crossentropie fonctionne toujours avec des probabilités, nous en reparlerons un peu plus tard. Ensuite, il y a simplement deux façons: soit les probabilités entrent déjà dans l'entrée, soit les logits arrivent à l'entrée - puis sigmoïde leur est d'abord appliqué pour obtenir la probabilité. Il se trouve que l'application de sigmoïde et le calcul de l'entropie croisée se sont avérés meilleurs que le simple calcul de l'entropie croisée à partir des probabilités (la source de la fonction sigmoid_cross_entropy_with_logits a une conclusion mathématique, plus pour les curieux, vous pouvez google `` entropie croisée de stabilité numérique ''), donc même les développeurs de tensorflow recommandent de ne pas transmettre la probabilité à saisir des fonctions de crossentropie et rendre des logits bruts. Eh bien, dans le code, les fonctions de perte sont vérifiées si la dernière couche est sigmoïde, puis elles la coupent et prennent l'entrée d'activation, plutôt que sa sortie, à calculer, envoyant tout à prendre en compte dans sigmoid_cross_entropy_with_logits .
D'accord, trié, maintenant binary_crossentropy. Il existe deux explications «intuitives» populaires qui mesurent l'entropie croisée.
Plus formel: imaginez qu'il existe un certain modèle qui pour n classes connaît la probabilité de leur occurrence (y 0 , y 1 , ..., y n ). Et maintenant dans la vie, chacune de ces classes a surgi k n fois (k 1 , k 1 , ..., k n ). La probabilité d'un tel événement est le produit de la probabilité pour chaque classe individuelle - (y 1 ^ k 1 ) (y 2 ^ k 2 ) ... (y n ^ k n ). En principe - il s'agit déjà d'une définition normale de l'entropie croisée - la probabilité d'un ensemble de données est exprimée en termes de probabilité d'un autre ensemble de données. Le problème avec cette définition est qu'elle se révélera être de 0 à 1 et sera souvent très petite; il n'est pas pratique de comparer de telles valeurs.
Si nous prenons le logarithme de cela, alors k 1 log (y 1 ) + k 2 log (y 2 ) sortira et ainsi de suite. La plage de valeurs devient de -∞ à 0. Multipliez tout cela par -1 / n - et la plage de 0 à + ∞ apparaît, en outre, parce que il est exprimé comme la somme des valeurs pour chaque classe, le changement dans chaque classe se reflète dans la valeur globale d'une manière très prévisible.
Plus simple: l'entropie croisée montre combien de bits supplémentaires sont nécessaires pour exprimer l'échantillon en termes de modèle d'origine. Si nous étions là pour faire un logarithme avec la base 2, alors nous irions directement en bits. Nous utilisons des logarithmes naturels partout, donc ils montrent le nombre de nat ( https://en.wikipedia.org/wiki/Nat_(unit )), pas de bits.
L'entropie croisée binaire, à son tour, est un cas particulier de l'entropie croisée ordinaire, lorsque le nombre de classes est de deux. Ensuite, nous avons suffisamment de connaissances sur la probabilité d'occurrence d'une classe - y 1 , et la probabilité d'occurrence de la seconde sera (1-y 1 ).
Mais, il me semble, un peu dérapé. Permettez-moi de vous rappeler que la dernière fois que nous avons essayé de construire un encodeur automatique d'identité, il nous a montré une belle image et même une précision de 1,0, mais en fait, les chiffres se sont révélés horribles. Pour les besoins de l'expérience, vous pouvez effectuer quelques tests supplémentaires:
1) l'activation peut être supprimée, il y aura une identité propre
2) vous pouvez essayer d'autres fonctions d'activation, par exemple la même relu
Sans activation:
model = tf.keras.Sequential() model.add(tf.keras.Input(shape=(64,64,1))) model.add(tf.keras.layers.Flatten()) model.add(tf.keras.layers.Dense(64*64, use_bias=False, kernel_initializer=tf.keras.initializers.Identity())) model.add(tf.keras.layers.Reshape(target_shape=(64,64,1)))
Nous obtenons le modèle d'identité parfait:
model.evaluate(x=val.map(lambda x: (x,x)).batch(BATCH_SIZE, drop_remainder=True))
La formation, en passant, ne mènera à rien, car la perte == 0,0.
Maintenant avec relu. Son graphique ressemble à ceci:
import matplotlib.ticker as plticker range_tensor = tf.range(-4, 4, 0.01, dtype=tf.float32) fig, ax = plt.subplots(1,1) plt.plot(range_tensor.numpy(), tf.keras.activations.relu(range_tensor).numpy()) ax.grid(which='major', linestyle='-', linewidth='0.5', color='red') ax.grid(which='minor', linestyle=':', linewidth='0.5', color='black') ax.yaxis.set_major_locator(plticker.MultipleLocator(base=1) ) plt.minorticks_on()
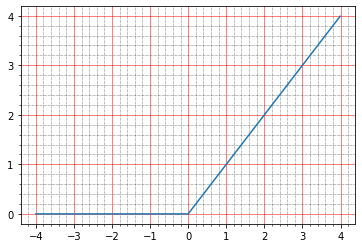
En dessous de zéro - zéro, au dessus - y = x, c'est-à-dire en théorie, nous devrions obtenir le même effet qu'en l'absence d'activation - un modèle idéal.
model = tf.keras.Sequential() model.add(tf.keras.Input(shape=(64,64,1))) model.add(tf.keras.layers.Flatten()) model.add(tf.keras.layers.Dense(64*64, activation='relu', use_bias=False, kernel_initializer=tf.keras.initializers.Identity())) model.add(tf.keras.layers.Reshape(target_shape=(64,64,1))) model.compile(optimizer="adam", loss="binary_crossentropy", metrics=["accuracy"]) model.evaluate(x=val.map(lambda x: (x,x)).batch(BATCH_SIZE, drop_remainder=True))
D'accord, nous avons compris le modèle d'identité, même avec une partie de la théorie, il est devenu plus clair. Essayons maintenant de former le même modèle pour qu'il devienne identité.
Pour le plaisir, je vais mener cette expérience sur trois fonctions d'activation. Pour commencer - relu, car il s'est montré bien plus tôt (tout est comme avant, mais le kernel_initializer est supprimé, donc par défaut ce sera glorot_uniform ):
model = tf.keras.Sequential() model.add(tf.keras.Input(shape=(64,64,1))) model.add(tf.keras.layers.Flatten()) model.add(tf.keras.layers.Dense(64*64, activation='relu', use_bias=False)) model.add(tf.keras.layers.Reshape(target_shape=(64,64,1)))
Il apprend à merveille:
Le résultat était assez bon, précision: 0,9999, perte (mse): 2e-04 après 20 époques et vous pouvez vous entraîner plus loin.

Ensuite, essayez avec sigmoid:
model = tf.keras.Sequential() model.add(tf.keras.Input(shape=(64,64,1))) model.add(tf.keras.layers.Flatten()) model.add(tf.keras.layers.Dense(64*64, activation='sigmoid', use_bias=False)) model.add(tf.keras.layers.Reshape(target_shape=(64,64,1)))
J'ai déjà enseigné quelque chose de similaire auparavant, à la seule différence que le biais est désactivé ici. Il étudie abondamment, monte sur un plateau dans la région de la 50e ère, précision: 0.9970, perte: 0.01 après 60 époques.
Le résultat n'est pas encore impressionnant:
Eh bien, vérifiez également tanh:
model = tf.keras.Sequential() model.add(tf.keras.Input(shape=(64,64,1))) model.add(tf.keras.layers.Flatten()) model.add(tf.keras.layers.Dense(64*64, activation='tanh', use_bias=False)) model.add(tf.keras.layers.Reshape(target_shape=(64,64,1)))
Le résultat est comparable à relu - précision: 0,9999, perte: 6e-04 après 20 époques, et vous pouvez vous entraîner davantage:
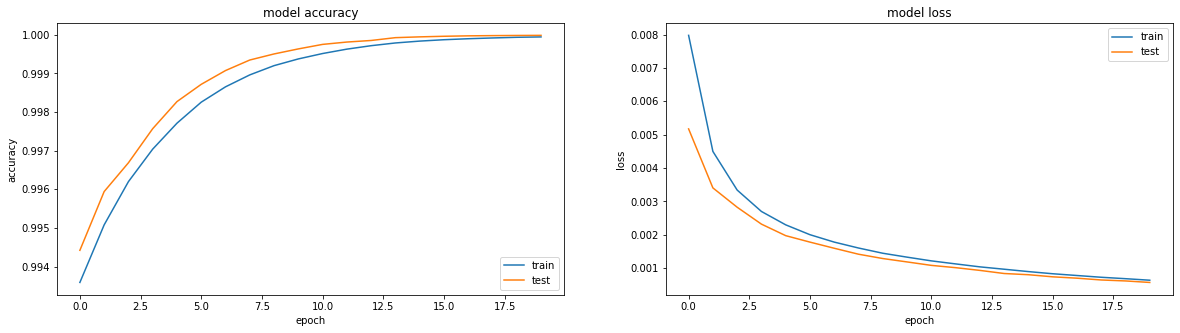

En fait, je suis tourmenté par la question de savoir si quelque chose peut être fait pour que sigmoïde affiche un résultat comparable. Exclusivement par intérêt sportif.
Par exemple, vous pouvez essayer d'ajouter BatchNormalization:
model = tf.keras.Sequential() model.add(tf.keras.Input(shape=(64,64,1))) model.add(tf.keras.layers.Flatten()) model.add(tf.keras.layers.Dense(64*64, activation='sigmoid', use_bias=False)) model.add(tf.keras.layers.BatchNormalization()) model.add(tf.keras.layers.Reshape(target_shape=(64,64,1)))
Et puis une sorte de magie se produit. À la 13e ère, précision: 1.0. Et les résultats ardents:
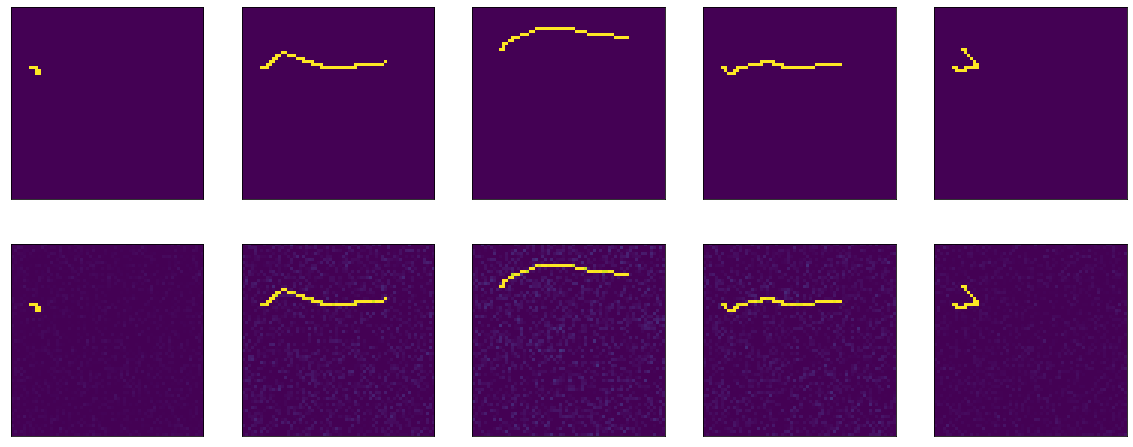
III ... sur cette falaise, je terminerai la première partie, parce que le texte est trop dofig, et on ne sait pas si quelqu'un en a besoin ou non. Dans la deuxième partie, je vais comprendre ce qui s'est passé, expérimenter différents optimiseurs, essayer de construire un encodeur-décodeur honnête, me cogner la tête sur la table. J'espère que quelqu'un était intéressé et utile.