MIT ने उच्च प्रदर्शन वाली सामान्य प्रयोजन की भाषा जूलिया को छोड़ने की घोषणा करने के बाद एक साल से थोड़ा अधिक समय बीत चुका है। तब से, भाषा ने लोकप्रियता हासिल की है: इसका उपयोग 1,500 से अधिक विश्वविद्यालयों में किया जाता है (कुछ में इसे शिक्षा की पहली भाषा के रूप में पढ़ाया जाता है), और मेडिकल डायग्नोस्टिक्स और अंतरिक्ष मिशन योजना से आवेदन कवर के क्षेत्र जैसे स्कूल बस यातायात के अनुकूलन जैसी समस्याओं को दबाने के लिए।
कई परियोजनाओं की गतिविधि के प्रमुख क्षेत्रों में से एक, यह अनुमान लगाना मुश्किल नहीं है, मशीन लर्निंग है, जिसके लिए जूलिया के पास कई शक्तिशाली उपकरण हैं , और एक दिलचस्प परियोजना हाल ही में प्रकाशित हुई है - जनरल प्रोबेबिलिटी प्रोग्रामिंग सिस्टम "जेन" ।
आज हम ध्यान देंगे, जैसा कि नाम से पता चलता है, फ्लक्स पैकेज, जो तंत्रिका नेटवर्क की सभी शक्ति प्रदान करता है। हम एक पूर्ण क्लासिफायरियर प्राप्त करने के लिए एक प्रशिक्षित तंत्रिका नेटवर्क में छवियों के प्रसंस्करण और शोध सेट से जाने की कोशिश करेंगे!
स्थापना
आधिकारिक साइट से वितरण किट डाउनलोड करें और अपने कंप्यूटर पर जूलिया दुभाषिया ( REPL ) स्थापित करें।
पैकेज मैनेजर के सही ढंग से काम करने के लिए, विंडोज 7 / विंडोज सर्वर 2012 के उपयोगकर्ताओं को भी इंस्टॉल करना होगा:
REPL में काम करने की प्रक्रिया कुछ इस प्रकार है:
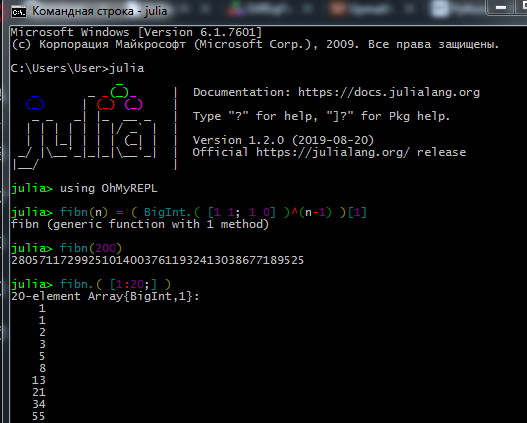
ट्रू डेट्रायटिस्ट और मशीन-लिंगोलॉजिस्ट जुपिटर को पसंद करते हैं। यहां आप स्थापना के बारे में देख सकते हैं , साथ ही रूसी में असाइनमेंट (मूल ट्यूटोरियल के लिंक और वहां की भाषा के लिए एक गाइड) के साथ स्व-अध्ययन के लिए इंटरैक्टिव पाठ भी पा सकते हैं।
यहां आप देख सकते हैं कि जुपिटर नोटबुक के साथ कैसे काम किया जाए।
यदि स्थापना समस्याओं- कनेक्शन स्थापित नहीं किया जा सकता - अपने एक्सेस अधिकारों की जांच करें (क्या आपके पास C: \ पर फ़ोल्डर्स लिखने पर प्रतिबंध है, व्यवस्थापक के रूप में लॉग इन करें या व्यवस्थापक मोड में जूलिया शुरू करें), यदि प्रॉक्सी का उपयोग कर रहे हैं, तो सुनिश्चित करें कि यह न केवल ब्राउज़र के लिए कॉन्फ़िगर किया गया है।
- कुछ पैकेज फ़ाइल पथ में सिरिलिक वर्णमाला पसंद नहीं करते हैं, इसलिए रूसी में उपयोगकर्ता नाम के कारण मुझे बहुत सारी समस्याएं थीं
- यदि इंटरैक्शन पैकेज परिणाम प्रदर्शित नहीं करता है, तो आपने WebIO को गलत तरीके से स्थापित किया हो सकता है, जिसे ठीक किया जा सकता है
- कुछ पैकेजों के लिए विंडोज पर सही ढंग से काम करने के लिए, जूलिया और जुपिटर के मार्गों को पर्यावरण चर में दर्ज किया जाना चाहिए।
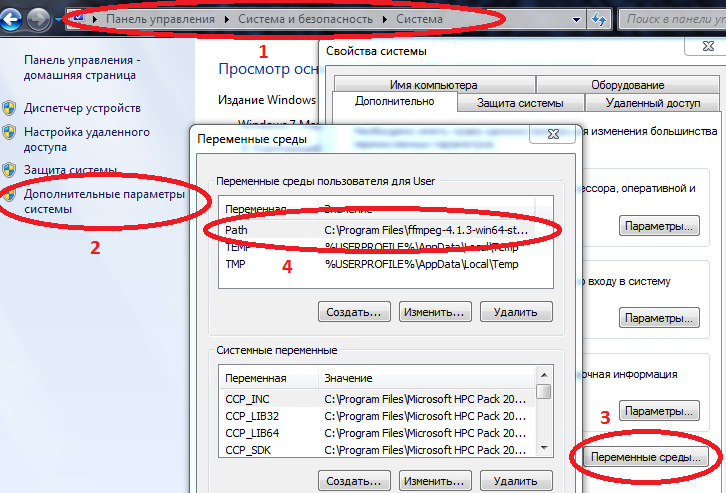
कंप्यूटर / सिस्टम गुण / उन्नत सिस्टम पैरामीटर / पर्यावरण चर / पथ (बनाएँ नहीं तो) और वहाँ julia.exe के लिए पथ जोड़ें
उदाहरण C: \ Users \ User \ AppData \ Local \ Julia-1.2.0 \ bin
यदि पथ में पहले से ही मान हैं, तो उन्हें अर्धविराम से अलग करें।
अब यदि आप julia को कमांड कंसोल ( cmd ) में चलाते हैं, तो दुभाषिया शुरू हो जाएगा।
अपनी ज़रूरत की हर चीज़ को इंस्टॉल करने के बाद, आप आज ज़रूरी पैकेज डाउनलोड कर सकते हैं। REPL या Jupyter में कमांड दर्ज करें
कोड using Pkg pkgs = ["Plots", "TextParse", "CSV", "DataFrames", "ImageMagick", "Images", "Interact", "Flux"] for p in pkgs Pkg.add(p) end for p in pkgs Pkg.build(p) end
भाषा की मूल बातें सीखने (सरणियों के साथ काम करने, फ़ंक्शन बनाने, पैकेज डाउनलोड करने, ग्राफ़ की साजिश रचने) के बाद, आप बाद की सामग्री के लिए आगे बढ़ सकते हैं।
डेटा लोडिंग और प्रोसेसिंग
डेटा एकत्र करना और व्यवस्थित करना एक अलग कला है। जूलिया के बारे में, नेटवर्क में बहुत सारी पुरानी सामग्री है, लेकिन शुरुआत के लिए आप उपरोक्त ट्यूटोरियल की कोशिश कर सकते हैं, और अधिक गहन अध्ययन के लिए, जूलिया (पब्लिक डोमेन में) के साथ डेटा साइंस की पुस्तक पढ़ें।
और आज, शायद, हम पहले से तैयार किए गए डेटा के साथ काम करेंगे: विभिन्न कोणों से फलों की तस्वीरों की एक बड़ी संख्या से एक डेटासेट - जो एक ताजा फल चाहते थे?


वास्तव में यह कार्य है - हम केले से सेब को अलग करने के लिए तंत्रिका नेटवर्क सिखाएंगे!
पहली चीजें पहले, कुछ परीक्षण छवियां अपलोड करें:
using Images fnames = [ "data/10_100.jpg", "data/107_100.jpg", "data/yellow_apple_2.jpg", "data/8_100.jpg", "data/104_100.jpg", "data/3_100.jpg" ]

चित्रों में वस्तुएं एक दूसरे से कैसे भिन्न होती हैं? पहला, रूप से, दूसरा रंग से, और फिर बनावट और अन्य विशेषताओं द्वारा। छवि विश्लेषण अपने आप में एक दिलचस्प विषय है, और वर्गीकरण न केवल न्यूरॉन्स द्वारा, बल्कि तरंगों द्वारा भी कहा जा सकता है। हम सबसे सरल संकेत - रंग से शुरू करेंगे।
जैसा कि आप जानते हैं, छवियों को कंप्यूटर मेमोरी में सरणियों के रूप में संग्रहीत किया जाता है, हमारे मामले में ये मैट्रीस हैं, जिनमें से प्रत्येक सेल में तीन नंबर होते हैं, जो छवि के प्रत्येक पिक्सेल में लाल, हरे और नीले रंगों की मात्रा का संकेत देते हैं। आइए देखें इन चित्रों में प्रत्येक रंग की औसत मात्रा:
using Statistics: mean M1 = [ mean(float.(c.(img))) for c = [red,green,blue], img = fruits ] 3×6 Array{Float32,2}: 0.570278 0.652852 0.977111 0.835252 0.903998 0.842564 0.338118 0.468729 0.950773 0.806882 0.880692 0.755442 0.322406 0.379424 0.835212 0.707626 0.799643 0.761916
हम पहली पंक्ति को ध्यान से देखते हैं - क्या यह आपको परेशान नहीं करता है? एक पीला सेब और केले ब्रेबर्न किस्म के सेब की तुलना में लाल होते हैं! ऐसा कैसे! चलो, खट्टा खानों को बनाओ, शायद स्कूली बच्चे इस ट्यूटोरियल को पढ़ रहे हैं, या बैले और ट्रैक्टर संस्थान के युवा छात्र। इसलिए, हम चूक से बचने की कोशिश करेंगे। तथ्य यह है कि प्रत्येक तस्वीर की पृष्ठभूमि सफेद है, और आरजीबी अंकन में यह मूल्यों (1,1,1) द्वारा दर्शाया गया है। और चूंकि 3 डैश छवियों पर 6 से अधिक पृष्ठभूमि हैं, साथ ही केले और पीले सेब के रंग में भी लाल रंग शामिल हैं, यह पता चला है कि पहले दो चित्र लाल रंग में खो जाते हैं। स्पष्टता के लिए, हम चित्रों को मूल रंगों में विभाजित करते हैं:
function tweaking(img) R = colorview( RGB, red.(img),zeroarray,zeroarray ) G = colorview( RGB, zeroarray,green.(img),zeroarray ) B = colorview( RGB, zeroarray,zeroarray, blue.(img) ) [R; G; B] end tweaking( hcat(fruits...) )

क्या आपने कभी क्रिप्टिक शब्द "आधार" सुना है? तो, हम कह सकते हैं कि इन छवियों को एक RGB बेस में रखा गया है। ब्लैकर - कम एक निश्चित रंग, और जैसा कि हमने उम्मीद की थी, इसकी समृद्धि के साथ पृष्ठभूमि औसत शोर की गणना करती है। इसे हटा दें।
function remove_background(img) mtrx = copy( channelview(img) ) for i = 1:size(mtrx, 2), j = 1:size(mtrx, 3) if reduce(&, mtrx[:,i,j] .> [0.8, 0.8, 0.8])

M3 = [ mean(float.(c.(img))) for c = [red,green,blue], img = greyfruits ] 3×6 Array{Float32,2}: 0.451008 0.532696 0.578967 0.527727 0.52849 0.500276 0.218805 0.348609 0.552679 0.499192 0.505136 0.412946 0.203528 0.260142 0.439354 0.400631 0.424784 0.419291
प्रत्येक वस्तु के कब्जे वाले क्षेत्र में अंतर अभी भी प्रभावित कर रहा है, लेकिन सामान्य तौर पर, यह निष्कर्ष निकाला जा सकता है कि केले हरियाली ( और नीले ) सेब हैं। यह मूल्यांकन मानदंड होगा, वह है - एक संकेत। अब आइए बाकी तस्वीरों पर एक नजर डालते हैं:
pth = "C:\\Users\\User\\Desktop\\Banana"
प्रत्येक छवि के लिए, हम पृष्ठभूमि के योगदान को बेअसर करते हैं, हम प्रत्येक रंग की औसत राशि पाते हैं, साथ ही साथ छवियों के आकार को याद करते हैं ...
dataz = [] for fname in fnames img_i = load("$pth\\$fname") gbimg = remove_background(img_i) colorz = [ mean(c.(gbimg)) for c = [red,green,blue] ] inform = [size(gbimg, 1) size(gbimg, 2) colorz' ] push!(dataz, inform) end dataz
... और फिर आप हमारे डेटा को कार्य के लिए सुविधाजनक संरचनाओं में व्यवस्थित कर सकते हैं - डेटा फ़्रेम:
using DataFrames, CSV banans = DataFrame( vcat(dataz...), [:height, :width, :red, :green, :blue] ) CSV.write("data/bananas.csv", banans)
apples = CSV.read("data/Apple_Braeburn.csv")
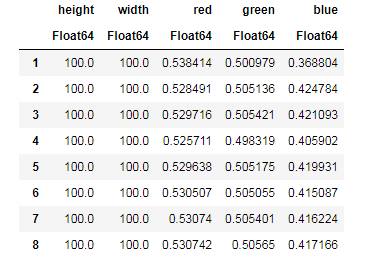
Desc = describe(apples, :all)
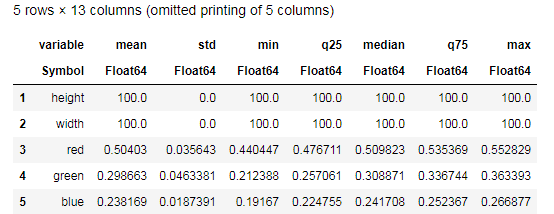
describe() फ़ंक्शन द्वारा प्रदान किए गए डेटा को समझने और केले के लिए एक समान तालिका के साथ तुलना करने का प्रयास करें। खैर, ग्राफ़ के बिना किस तरह का डेटा विश्लेषण हो सकता है?
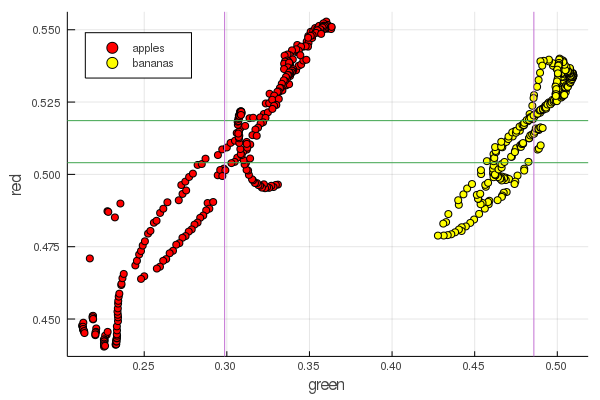
function plot2features(clr) x_apples = apples[:, :green] x_banans = banans[:, :green] y_apples = apples[:, clr] y_banans = banans[:, clr] scatter(x_apples, y_apples, lab = "apples", colour = :red) scatter!(x_banans, y_banans, lab = "bananas", legend = :topleft, colour = :yellow) hline!([mean(y_apples), mean(y_banans) ], lab = "" ) vline!([mean(x_apples), mean(x_banans) ], lab = "" ) xaxis!("green") yaxis!("$clr") end plot2features(:red)
plot2features(:blue)
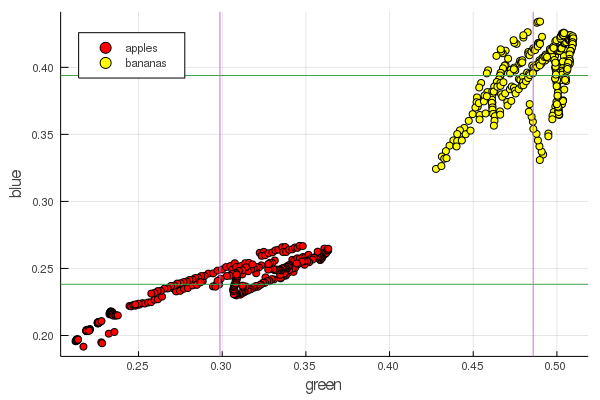
मिड-केला लाल, मध्य-सेब के मूल्य के बहुत करीब है। लेकिन दूसरे चार्ट में, एक ही बार में दो रंग विशेषताओं द्वारा फलों का अलगाव तुरंत अधिक स्पष्ट रूप से पता लगाया जाता है। पृथक्करण को सही पुनर्जलीकरण द्वारा सुधारा जा सकता है, उदाहरण के लिए, हमारे हरे रंग के मान 0.2 से 0.55 तक बदल जाते हैं, और यदि आप रूपांतरण करते हैं
तब हमें डेटा [0,1] द्वारा पुनः प्राप्त होता है, जो इन दोनों के बीच अंतर को बढ़ाएगा ढेर बिंदुओं के समूह।
perceptron
वर्गीकरण कार्य में एक मॉडल को परिभाषित करने और मापदंडों का चयन करना शामिल है, जिसके लिए विभिन्न डेटा विशिष्ट रूप से एक विशेष वर्ग से संबंधित हैं। सीधे शब्दों में कहें, हमें एक निश्चित कार्य शुरू करने और इसके मापदंडों को निर्धारित करने की आवश्यकता है ताकि यह हमारे सेब को केले से अलग कर दे।
इन उद्देश्यों के लिए सबसे प्रसिद्ध और लोकप्रिय मॉडल मैककुलोच-पिट्स कृत्रिम न्यूरॉन है, जिसे 1940 के दशक की शुरुआत में विकसित किया गया था। इसके बाद, फ्रैंक रोसेनब्लट ने एक प्रशिक्षित तंत्रिका नेटवर्क का प्रस्ताव रखा - पेसेप्ट्रॉन। तंत्रिका संसाधनों के बारे में व्यापक स्पष्टीकरण प्राप्त करना मुश्किल नहीं है, इस संसाधन पर (उदाहरण के लिए शुरुआती के लिए तंत्रिका नेटवर्क, छवि मान्यता में तंत्रिका नेटवर्क का उपयोग , तंत्रिका नेटवर्क, संचालन के मूलभूत सिद्धांत, विविधता और टोपोलॉजी )
सिग्मॉइड को सक्रियण फ़ंक्शन के रूप में चुनना और इसके आउटपुट के अनुसार वर्गीकृत वस्तुओं (फलों) के आउटपुट सेट करना
ऐसे मापदंडों का चयन करें और ताकि प्राप्त आंकड़ों के लिए सिग्मॉइड का आउटपुट मान उपरोक्त संकेतन के अनुरूप हो
using Interact sigmo(x,w,b) = 1 / (1 + exp(-w*x+b)) r_apples, g_apples, b_apples = apples[:, :red], apples[:, :green], apples[:, :blue] r_banans, g_banans, b_banans = banans[:, :red], banans[:, :green], banans[:, :blue]; @manipulate for w in 10:1:60, b in -5:1:25 plot(x->sigmo(x,w,b), 0, 1, label="Model", legend = :topleft, lw=3) scatter!(g_apples[1:5], zeros(10), label="Apple", colour = :red) scatter!(g_banans[1:5], ones(10), label="Banana", colour = :yellow) end
foon(x) = sigmo(x,60,24) plot(foon, 0, 1, label="Model", legend = :topleft, lw=3) scatter!(foon, g_apples, label="Apple", colour = :red) scatter!(foon, g_banans, label="Banana", colour = :yellow) xaxis!("green")
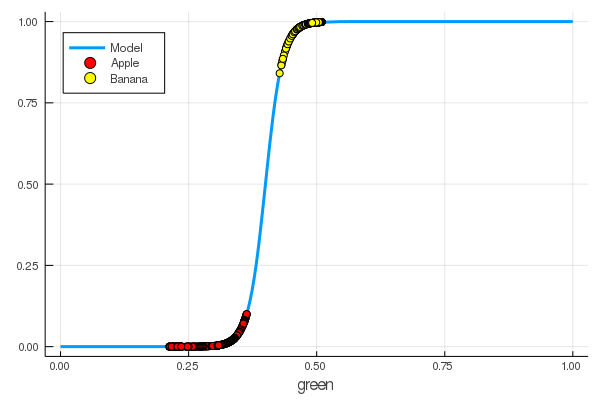
हमने मैन्युअल रूप से हरे रंग की मात्रा से केले को सेब से अलग करने के लिए एक न्यूरॉन सिखाया!
स्वाभाविक रूप से, इस प्रक्रिया को स्वचालित करने की इच्छा। हम नुकसान फ़ंक्शन का परिचय देते हैं
अब सीखने की प्रक्रिया इस समारोह को कम करने में शामिल होगी:
कोड apples_mean_green = mean(g_apples) banans_mean_green = mean(g_banans) L(w, b) = (0 - sigmo(apples_mean_green,w,b))^2 + (1 - sigmo(banans_mean_green,w,b))^2 w_range = 10:0.5:30 b_range = 0:0.5:20 L_values = [L(w,b) for b in b_range, w in w_range] @manipulate for w in w_range, b in b_range p1 = surface(w_range, b_range, L_values, xlabel="b", ylabel="w", cam=(80,40), cbar=false, leg=false) scatter!(p1, [w], [b], [L(w,b)+1e-2], markersize=5, color = :blue) p2 = plot(x->sigmo(x,w,b), 0, 1, label="Model", legend = :topleft, lw=3) scatter!(p2, [apples_mean_green], [0.0], label="Apple", markersize=10) scatter!(p2, [banans_mean_green], [1.0], label="Banana", markersize=10, xlim=(0,1), ylim=(0,1)) plot(p1, p2, layout=(2,1)) end
इससे पहले हमने जूलिया के लिए पैकेजों का अध्ययन किया जो विभिन्न तरीकों से अनुकूलन समस्याओं को हल करने की अनुमति देता है। सौभाग्य से, आवश्यक पहले से ही फ्लक्स वातावरण में हैं!
फ्लक्स
using Flux
सबसे पहले, हम एक सुपाच्य रूप में प्रशिक्षण के लिए डेटा प्रस्तुत करते हैं:
Y = [zeros(length(g_apples)); ones(length(g_banans)) ] |> permutedims X = [g_apples; g_banans] |> permutedims;
अगले क्रम में:
- हम इन डेटा के वर्गीकरण के संबंध में सही उत्तरों के साथ इनपुट डेटा को जोड़कर एक प्रशिक्षण डेटासेट बनाते हैं
- हम यादृच्छिक मानों के मैट्रिक्स द्वारा डब्ल्यू और बी को निर्धारित करते हैं (इनपुट में एक संकेत है और आउटपुट पर एक है, इसलिए मैट्रिक्स आकार में 1 x 1 हैं )
- एक मॉडल के रूप में, हम एक घने परत सेट करते हैं - एक सिग्माइडल सक्रियण फ़ंक्शन के साथ एक अवधारण
- हमने नुकसान फ़ंक्शन सेट किया है - चुकता अंतर का योग (आप अभी भी अधिक लोकप्रिय
Flux.crossentropy() उपयोग कर सकते हैं Flux.crossentropy() ) - एक अनुकूलन विधि के रूप में, हम ढाल वंश का चयन करते हैं । यह एक पैरामीटर लेता है - वंश की गति
- हम एक मूल्यांकन फ़ंक्शन सेट करते हैं जो मॉडल आउटपुट के मूल्यों को गोल करेगा और उनकी तुलना सही उत्तरों के साथ करेगा।
- और हमारे अप्रशिक्षित मॉडल के मापदंडों को प्रिंट करें
dataz = [(X, Y)] W = param(rand(1)) b = param(rand(1)) model = Dense(W, b, σ) loss(x, y) = mse(model(x), y) opt = Descent(0.1) accuracy(x, y) = mean( round.(model(x)) .== y ) params(model) Params([[0.3372841444115968] (tracked), [0.8430399003786011] (tracked)])
आइए देखें कि हमारे डेटा के लिए नुकसान फ़ंक्शन का आउटपुट क्या है।
loss(X, Y)
और मूल्यांकन समारोह के परिणामों की जांच करें
accuracy(X, Y) 0.5
परिणाम काफी स्वाभाविक है - आउटपुट को समान रूप से वितरित किया जाता है और डेटा का आधा सही ढंग से वर्गीकृत किया जाता है:
कोड modeldataz(x) = x |> model |> data |> permutedims
modelX = modeldataz(X) modelapples = modeldataz(g_apples') modelbanans = modeldataz(g_banans') plot(modelX, legend = false) hline!([0.5]) p1 = yaxis!((0,1)) curv = [-1:0.01:1;]' |> modeldataz plot( [-1:0.01:1;], curv, label="Model", legend = :topleft, lw=3) scatter!(g_apples, modelapples, label="Apple", colour = :red) scatter!(g_banans, modelbanans, label="Banana",colour = :yellow) hline!([0.5], lab = "", legend = :topleft) p2 = xaxis!("green") plot(p1, p2)
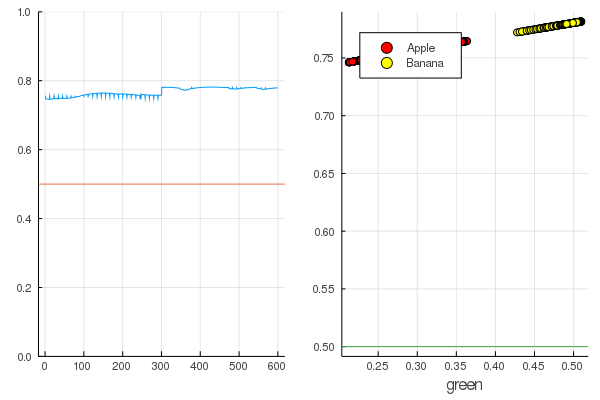
चलो शुरू करें: यह बहुत सरल है। आपको केवल तंत्रिका नेटवर्क पर चिल्लाने की आवश्यकता है: "ट्रेन!", जबकि यह इंगित करने के लिए कि ट्रेन को क्या करना है और क्या कम करना है, और वह एक प्रशिक्षण सत्र पूरा करेगी। इसलिए, हम उसे सब कुछ ठीक करने के लिए मजबूर करेंगे, जैसा कि उसे होना चाहिए, लेकिन केवल कट्टरता के बिना, ताकि कोई पीछे न हटे
for i in 1:7000 train!(loss, params(model), dataz, opt) end model.W, model.b ([9.578663260720564] (tracked), [-3.7540362587506464] (tracked))
नुकसान बहुत कम हो गए हैं:
loss(X, Y) 0.09152783090457564 (tracked)
एक रेटिंग बेहतर है:
accuracy(X, Y) 1.0
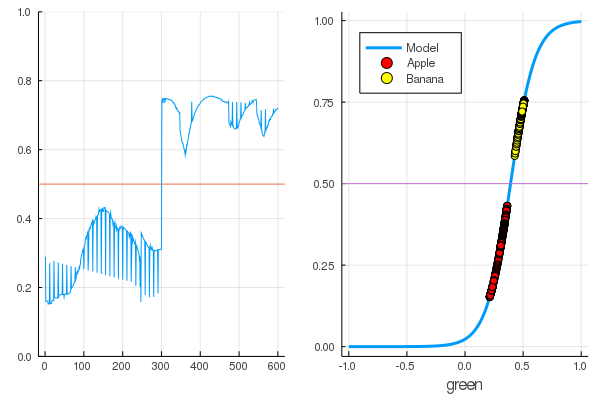
डेटा विभाजित है, और आगे के प्रशिक्षण से मॉडल फ़ंक्शन अधिक ऊर्ध्वाधर हो जाएगा। फलों के पहले सेट पर प्रशिक्षित मॉडल की जाँच करें:
function classifier(img) gbimg = remove_background(img) greenmean = mean(float.(green.(gbimg))) answ = data( model( [ greenmean ]' ) )[1] fr = answ > 0.5 ? "Banana" : "Apple" "$fr $(round(200abs(0.5-answ)))%" end hcat(fruits...)

classifier.(fruits) 6-element Array{String,1}: "Apple 68.0%" "Apple 20.0%" "Banana 65.0%" "Banana 47.0%" "Banana 49.0%" "Banana 10.0%"
एक विशेष रूप से लगाए गए पीले सेब, निश्चित रूप से, सही ढंग से पहचाने नहीं गए थे, और एक लाल केला मुश्किल से अपनी श्रेणी में प्रवेश किया था। लेकिन न्यूरॉन को तस्वीर से केवल एक नंबर मिलता है - हरे रंग की औसत मात्रा। आप एक और संकेत जोड़ सकते हैं, कह सकते हैं, नीले रंग की मात्रा, जो मॉडल को थोड़ा अधिक अनुकूल बना देगा।
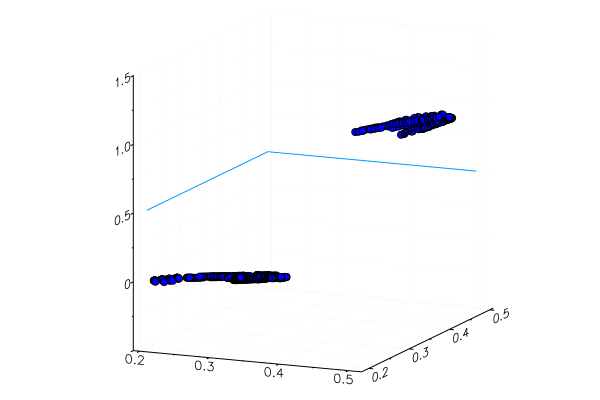
या आप आरजीबी प्रतिनिधित्व का उपयोग नहीं कर सकते हैं, लेकिन एचएसवी (ह्यू, संतृप्ति, मूल्य), जिसमें ह्यू चैनल में छवि के रंग के बारे में जानकारी होगी।
तंत्रिका नेटवर्क की पूरी व्याख्या यह है कि वे स्वयं उन विशेषताओं को अलग कर सकते हैं जो कभी-कभी बहुत स्पष्ट नहीं होती हैं (रंग सहसंबंध, उनका वितरण, रूपरेखा और घटता ...), और आप विशेष उत्तराधिकार और तकनीकों की मदद से उनकी मदद कर सकते हैं, जो तंत्रिका नेटवर्क के साथ काम करते हैं। असली कला।

ताकि नेतृत्व बहुत अधिक न बढ़े और लेखों की एक श्रृंखला करते हैं आइए हम हस्तलिखित संख्याओं के साथ चित्रों के वर्गीकरण का एक उदाहरण भी देते हैं, और इच्छुक पाठक स्वयं फलों के साथ छवियों में प्राप्त ज्ञान को सामान्य बनाएंगे और अपने स्वयं के तंत्रिका नेटवर्क का निर्माण करेंगे, जो अभी भी जीवन में वस्तुओं को चिह्नित करने में सक्षम, कहते हैं!
MNIST
using Images using Flux, Flux.Data.MNIST, Statistics using Flux: onehotbatch, onecold, crossentropy, throttle using Base.Iterators: repeated
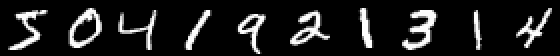
एक उदाहरण दिलचस्प है कि पहले से ही दस निकास हैं। तथाकथित वन-हॉट वैक्टर यहां काम आते हैं।
labels = MNIST.labels()
10×60000 Flux.OneHotMatrix{Array{Flux.OneHotVector,1}}: 0 1 0 0 0 0 0 0 0 0 0 0 0 … 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 1 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 1 0 1 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 … 0 0 0 0 0 1 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 0 1 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 1 0 0 0 0 0 0 0
हम एक मॉडल के रूप में न्यूरॉन्स की एक श्रृंखला को परिभाषित करते हैं, क्रॉस-एन्ट्रापी एक नुकसान फ़ंक्शन होगा, और एडम एक अनुकूलन विधि के रूप में:
m = Chain( Dense(28^2, 32, relu), Dense(32, 10), softmax) loss(x, y) = crossentropy(m(x), y) accuracy(x, y) = mean(onecold(m(x)) .== onecold(y)) dataset = repeated((X, Y), 20) evalcb = () -> @show(loss(X, Y)) opt = ADAM()
एक बख्शते मोड में ट्रेन, लेकिन हर 10 सेकंड में नुकसान का प्रिंट आउट:
for i = 1:10 Flux.train!(loss, params(m), dataset, opt, cb = throttle(evalcb, 10)) end
accuracy(X, Y) 0.64545
और प्रशिक्षण में उपयोग नहीं किए गए डेटा की जांच करें
जूलिया पर तंत्रिका नेटवर्क सरल और बहुत रोमांचक है! यहां तक कि अगर आपके गतिविधि और मशीन सीखने के क्षेत्र के बीच कनेक्शन देखने की कोई आवश्यकता नहीं है, तो आपको कम से कम इस जिज्ञासा को महसूस करना चाहिए, जो सभी कोणों से चिल्लाया जाता है, और उपकरणों की कोई कमी नहीं होगी!
सभी मध्यम सीपीयू गर्मी!