
Ketika menulis artikel tentang pengembangan detektor anomali, saya menerapkan salah satu algoritma yang disebut Gas Neural Penambahan.
Masuk Sastra Soviet segmen Internet Rusia, topik ini dibahas agak buruk, dan hanya ada satu artikel , dan itupun dengan aplikasi algoritma ini.
Jadi apa sebenarnya algoritma gas neural yang bertambah itu?
Pendahuluan
IGNG, seperti GNG , adalah algoritma pengelompokan adaptif.
Algoritma itu sendiri dijelaskan dalam sebuah artikel oleh Prudent dan Ennadji untuk 2005 .
Seperti GNG, ada banyak vektor data X atau menghasilkan fungsi f(t) , yang menyediakan vektor dari data yang didistribusikan secara acak (parameter t - waktu, atau nomor sampel dalam sampel).
Algoritma tidak memberlakukan batasan tambahan pada data ini.
Tetapi di dalam sangat berbeda dari GNG.
Algoritma ini juga menarik karena sedikit lebih akurat daripada neurogenesis model GNG.
Deskripsi algoritma
Algoritma memecah banyak data menjadi cluster.
Dibandingkan dengan GNG, keunggulannya adalah tingkat konvergensi yang lebih tinggi.
Gagasan yang menjadi dasar algoritme:
- Teori Resonansi Adaptif : Pertama, neuron terdekat dicari, dan jika perbedaannya tidak melebihi ambang batas ("parameter kewaspadaan"), bobot disesuaikan atau, jika tidak, koordinat neuron dalam ruang data diubah. Jika ambang batas belum diatasi, neuron baru dibuat yang lebih baik mendekati nilai sampel data.
- Baik koneksi dan neuron memiliki parameter usia (GNG hanya memiliki koneksi), yang pada awalnya adalah nol, tetapi meningkat saat Anda belajar.
- Neuron tidak segera muncul: pertama embrio (atau neuron germinal) muncul, usia meningkat dengan setiap iterasi, sampai matang. Setelah pelatihan, hanya neuron dewasa yang berpartisipasi dalam klasifikasi .
Siklus utama
Pekerjaan dimulai dengan grafik kosong. Parameter sigma diinisialisasi oleh standar deviasi sampel pelatihan:
sigma= sqrt frac1N jumlah LimitNi=1 kiri(xi− barx kanan)2
Dimana: barx - rata-rata di antara koordinat sampel.
Loop utama pada setiap langkah mengurangi nilai sigma , yang merupakan ambang kedekatan, dan menghitung perbedaan antara tingkat kualitas pengelompokan sebelumnya dan tingkat yang diperoleh setelah pengelompokan oleh prosedur IGNG .
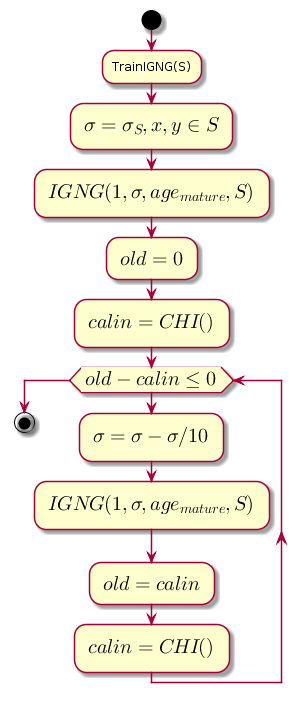
Kode bagan.@startuml start :TrainIGNG(S); :<latex>\sigma = \sigma_S,x,y \in S</latex>; :<latex>IGNG(1, \sigma, age_{mature}, S)</latex>; :<latex>old = 0</latex>; :<latex>calin = CHI()</latex>; while (<latex>old - calin \leq 0</latex>) :<latex>\sigma=\sigma - \sigma / 10</latex>; :<latex>IGNG(1, \sigma, age_{mature}, S)</latex>; :<latex>old = calin</latex>; :<latex>calin = CHI()</latex>; endwhile stop @enduml
CHI adalah indeks Kalinsky-Kharabaz, menunjukkan kualitas pengelompokan:
CHI= fracB/(c−1)W/(n−c)
Dimana:
- n - jumlah sampel data.
- c - jumlah cluster (dalam hal ini, jumlah neuron).
- B - matriks dispersi internal (jumlah jarak kuadrat antara koordinat neuron dan rata-rata semua data).
- W - matriks dispersi eksternal (jumlah jarak kuadrat antara semua data dan neuron terdekat).
Semakin besar nilai indeks, semakin baik, karena jika perbedaan antara indeks setelah pengelompokan dan sebelum negatif, adalah mungkin untuk mengasumsikan bahwa indeks menjadi positif dan melebihi yang sebelumnya, yaitu. pengelompokan berhasil diselesaikan.
Prosedur IGNG
Ini adalah prosedur dasar dari algoritma.
Ini dibagi menjadi tiga tahap yang saling eksklusif:
- Tidak ditemukan neuron.
- Satu neuron yang memuaskan ditemukan.
- Ditemukan dua yang memenuhi kondisi neuron.
Jika salah satu kondisi berlalu, langkah-langkah lain tidak dilakukan.
Pertama, neuron dicari untuk sampel data perkiraan terbaik:
c1=mnt(dist( xi, omegac))
Di sini dist(x omega,x xi) - fungsi perhitungan jarak, yang biasanya merupakan metrik Euclidean .
Jika neuron tidak ditemukan, atau terlalu jauh dari data, mis. tidak memenuhi kriteria kedekatan dist( xi, omegac) leq sigma , neuron embrionik baru dibuat dengan koordinat yang sama dengan koordinat sampel dalam ruang data.

Jika pemeriksaan kedekatan telah berlalu, neuron kedua dicari dengan cara yang sama dan memeriksa kedekatan dengan sampel data.
Jika neuron kedua tidak ditemukan, itu dibuat.

Jika dua neuron ditemukan yang memenuhi kondisi kedekatan dengan sampel data, koordinatnya dikoreksi sesuai dengan rumus berikut:
epsilon(t)hc,ci= begincases epsilonb, jikac=ci epsilonn, jikaadakoneksiantarac=ci0,diothercase endcases
Delta omegac= epsilon(t)hc,c1 parallel xi− omegac parallel omegac= omegac+ Delta omegac
Dimana:
- epsilon(t) - langkah adaptasi.
- ci Adalah jumlah neuron.
- hc,c1 - Fungsi Neuron Neighborhood c dengan neuron pemenang (dalam hal ini, ia akan mengembalikan 1 untuk tetangga langsung, 0 sebaliknya, karena langkah adaptasi, untuk perhitungan omega akan bukan nol hanya untuk tetangga langsung).
Dengan kata lain, koordinat (berat) dari neuron pemenang diubah menjadi epsilonb∗ Delta omegai , dan semua tetangga langsungnya (yang terhubung dengannya pada satu sisi grafik) pada epsilonn∗ Delta omegai dimana omegai - koordinat neuron yang sesuai sebelum perubahan.
Kemudian koneksi dibuat antara dua neuron pemenang, dan jika sudah dibuat, usianya diatur ulang.
Usia semua hubungan lainnya meningkat.
Semua komunikasi yang usianya melebihi konstanta agemax dihapus.
Setelah itu, semua neuron dewasa yang terisolasi (yang tidak memiliki koneksi dengan yang lain) dihilangkan.
Usia neuron langsung yang berdekatan dengan neuron pemenang semakin meningkat.
Jika usia salah satu dari germline neuron melebihi agemature Ia menjadi neuron yang matang.
Grafik terakhir hanya berisi neuron dewasa.
Kondisi untuk menyelesaikan prosedur IGNG di bawah ini dapat dianggap sebagai jumlah siklus yang tetap.
Algoritma ditunjukkan di bawah ini (gambar dapat diklik):
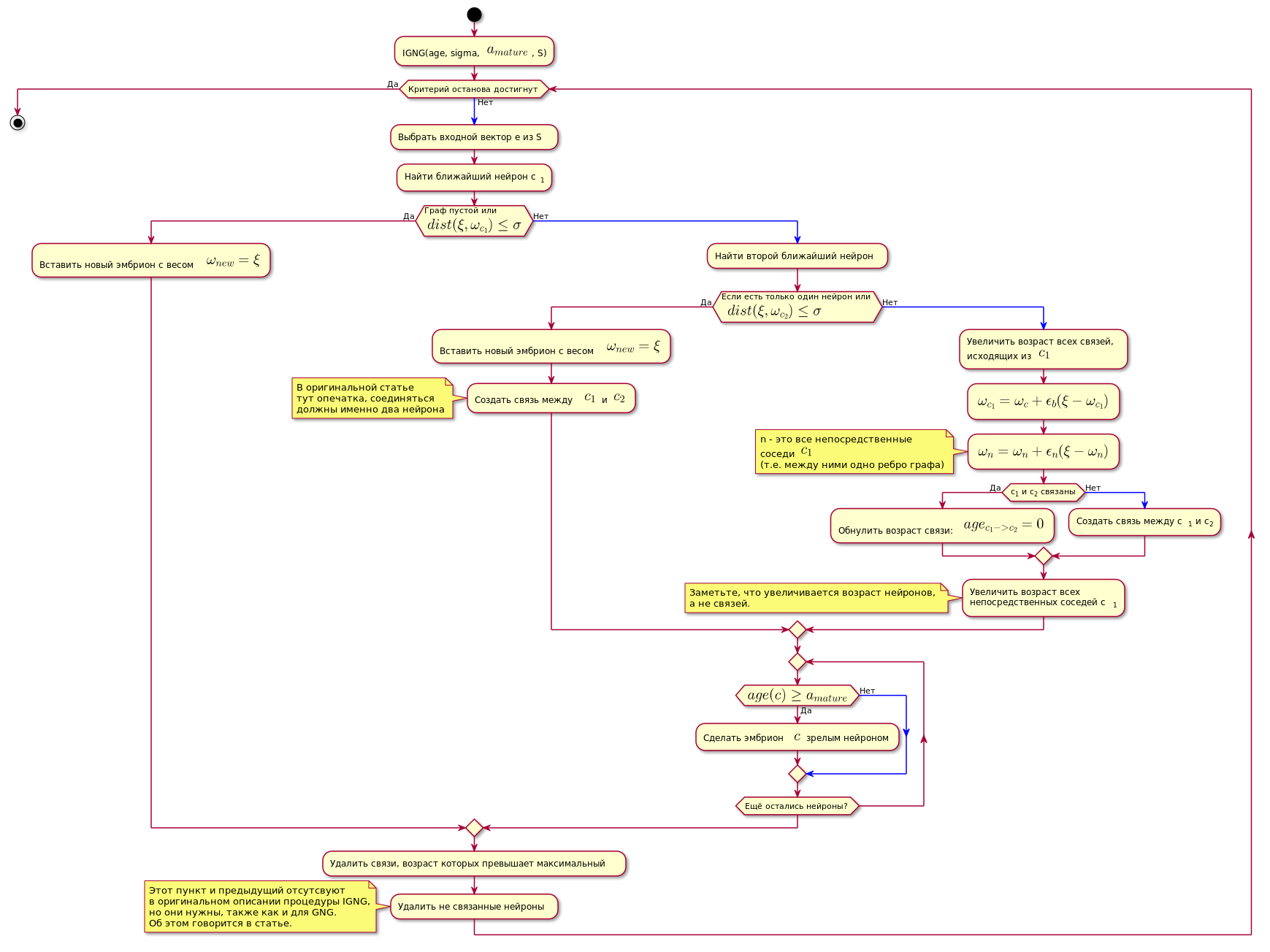
Kode bagan. @startuml skinparam nodesep 10 skinparam ranksep 20 start :IGNG(age, sigma, <latex>a_{mature}</latex>, S); while ( ) is () -[#blue]-> : e S; : c<sub>1</sub>; if ( \n<latex>dist(\xi, \omega_{c_1}) \leq \sigma</latex>) then () : <latex>\omega_{new} = \xi</latex>; else () -[#blue]-> : ; if ( \n <latex>dist(\xi, \omega_{c_2}) \leq \sigma</latex>) then () : <latex>\omega_{new} = \xi</latex>; : <latex>c_1</latex> <latex>c_2</latex>; note , end note else () -[#blue]-> : ,\n <latex>c_1</latex>; :<latex>\omega_{c_1} = \omega_c + \epsilon_b(\xi - \omega_{c_1})</latex>; :<latex>\omega_n = \omega_n + \epsilon_n(\xi - \omega_n)</latex>; note n - <latex>c_1</latex> (.. ) end note if (c<sub>1</sub> c<sub>2</sub> ) then () : : <latex>age_{c_1 -> c_2} = 0</latex>; else () -[#blue]-> : c<sub>1</sub> c<sub>2</sub>; endif : \n c<sub>1</sub>; note , , . end note endif repeat if (<latex>age(c) \geq a_{mature}</latex>) then () : $$ ; else () -[#blue]-> endif repeat while ( ?) endif : , ; : ; note IGNG, , GNG. . endnote endwhile () stop @enduml
Implementasi
Jaringan diimplementasikan dalam Python menggunakan perpustakaan grafik NetworkX . Memotong kode dari prototipe pada artikel sebelumnya diberikan di bawah ini. Ada juga penjelasan singkat untuk kode tersebut.
Jika ada yang tertarik dengan kode lengkap, ini adalah tautan ke repositori .
Contoh algoritma:

Sebagian besar kode class NeuralGas(): __metaclass__ = ABCMeta def __init__(self, data, surface_graph=None, output_images_dir='images'): self._graph = nx.Graph() self._data = data self._surface_graph = surface_graph