Bagaimana jika Anda memiliki ide untuk protein yang keren dan sehat, dan Anda ingin mendapatkannya dalam kenyataan? Misalnya, apakah Anda ingin membuat vaksin melawan
H. pylori (seperti
tim Slovenia di iGEM 2008 ) dengan membuat protein hibrida yang menggabungkan fragmen
E. coli flagellin yang merangsang respons kekebalan dengan
H. pylori flagellin yang biasa?
H. pylori Hybrid Flagellin Design Disampaikan oleh Tim Slovenia di iGEM 2008Yang mengejutkan, kami sangat dekat untuk membuat protein apa pun yang kami inginkan tanpa meninggalkan notebook Jupyter, berkat perkembangan terbaru dalam genomik, biologi sintetis, dan yang terbaru di laboratorium cloud.
Pada artikel ini, saya akan menunjukkan kode Python dari ide protein ke ekspresinya dalam sel bakteri, tanpa menyentuh pipet atau berbicara dengan siapa pun. Total biaya hanya beberapa ratus dolar! Menggunakan
terminologi Vijaya Pande dari A16Z , ini adalah Biologi 2.0.
Lebih khusus lagi, dalam artikel tersebut, kode Python dari lab cloud melakukan hal berikut:
- Sintesis urutan DNA yang mengkodekan protein apa pun yang saya inginkan.
- Mengkloning DNA sintetis ini menjadi vektor yang bisa mengekspresikannya.
- Transformasi bakteri dengan vektor ini dan konfirmasi bahwa ekspresi sedang terjadi.
Pengaturan python
Pertama, pengaturan Python umum yang diperlukan untuk setiap notepad Jupyter. Kami mengimpor beberapa modul Python yang berguna dan membuat beberapa fungsi utilitas, terutama untuk visualisasi data.
Kodeimport re import json import logging import requests import itertools import numpy as np import seaborn as sns import pandas as pd import matplotlib as mpl import matplotlib.pyplot as plt from io import StringIO from pprint import pprint from Bio.Seq import Seq from Bio.Alphabet import generic_dna from IPython.display import display, Image, HTML, SVG def uprint(astr): print(astr + "\n" + "-"*len(astr)) def show_html(astr): return display(HTML('{}'.format(astr))) def show_svg(astr, w=1000, h=1000): SVG_HEAD = '''<?xml version="1.0" standalone="no"?><!DOCTYPE svg PUBLIC "-//W3C//DTD SVG 1.1//EN" "http://www.w3.org/Graphics/SVG/1.1/DTD/svg11.dtd">''' SVG_START = '''<svg viewBox="0 0 {w:} {h:}" version="1.1" xmlns="http://www.w3.org/2000/svg" xmlns:xlink= "http://www.w3.org/1999/xlink">''' return display(SVG(SVG_HEAD + SVG_START.format(w=w, h=h) + astr + '</svg>')) def table_print(rows, header=True): html = ["<table>"] html_row = "</td><td>".join(k for k in rows[0]) html.append("<tr style='font-weight:{}'><td>{}</td></tr>".format('bold' if header is True else 'normal', html_row)) for row in rows[1:]: html_row = "</td><td>".join(row) html.append("<tr style='font-family:monospace;'><td>{:}</td></tr>".format(html_row)) html.append("</table>") show_html(''.join(html)) def clean_seq(dna): dna = re.sub("\s","",dna) assert all(nt in "ACGTN" for nt in dna) return Seq(dna, generic_dna) def clean_aas(aas): aas = re.sub("\s","",aas) assert all(aa in "ACDEFGHIKLMNPQRSTVWY*" for aa in aas) return aas def Images(images, header=None, width="100%"):
Laboratorium cloud
Seperti AWS atau cloud komputasi apa pun, lab cloud memiliki peralatan biologi molekuler, serta robot yang disewa melalui Internet. Anda dapat mengeluarkan instruksi untuk robot Anda dengan mengklik beberapa tombol pada antarmuka atau dengan menulis kode yang memprogramnya sendiri. Tidak perlu menulis protokol Anda sendiri, seperti yang akan saya lakukan di sini, bagian penting dari biologi molekuler adalah tugas rutin standar, jadi biasanya lebih baik bergantung pada protokol alien yang dapat diandalkan yang menunjukkan interaksi yang baik dengan robot.
Baru-baru ini, sejumlah perusahaan dengan lab cloud telah muncul:
Transkriptik ,
Autodesk Wet Lab Accelerator (beta, dan dibangun berdasarkan Transkriptik),
Arcturus BioCloud (beta),
Emerald Cloud Lab (beta),
Synthego (belum dimulai). Bahkan ada perusahaan yang dibangun di atas lab cloud seperti
Desktop Genetics , yang berspesialisasi dalam CRISPR.
Artikel ilmiah tentang penggunaan lab cloud dalam sains nyata mulai muncul.
Pada saat penulisan ini, hanya Transkriptik yang berada dalam domain publik, jadi kami akan menggunakannya. Seperti yang saya pahami, sebagian besar bisnis Transcriptic dibangun di atas otomatisasi protokol umum, dan penulisan protokol Anda sendiri dengan Python (seperti yang akan saya lakukan dalam artikel ini) kurang umum.
 “Sel kerja” transkriptik dengan lemari es di bagian bawah dan berbagai peralatan laboratorium di mimbar
“Sel kerja” transkriptik dengan lemari es di bagian bawah dan berbagai peralatan laboratorium di mimbarSaya akan memberikan instruksi robot Transkriptik pada
protokol otomatis . Autoprotocol adalah bahasa berbasis JSON untuk protokol penulisan robot laboratorium (dan manusia). Autoprotocol terutama dibuat di
perpustakaan Python ini . Bahasa ini awalnya dibuat dan masih didukung oleh Transkriptik, tetapi, seperti yang saya pahami, bahasa ini sepenuhnya terbuka. Ada
dokumentasi yang bagus.
Ide yang menarik adalah Anda dapat menulis instruksi untuk orang-orang di laboratorium terpencil di, katakanlah, China atau India, pada protokol otomatis dan berpotensi mendapatkan beberapa keuntungan dari penggunaan kedua orang (penilaian mereka) dan robot (kurangnya penilaian). Kita perlu menyebutkan
protokol. Di sini , ini adalah upaya untuk membakukan protokol untuk meningkatkan reproduktifitas, tetapi untuk manusia, bukan robot.
"instructions": [ { "to": [ { "well": "water/0", "volume": "500.0:microliter" } ], "op": "provision", "resource_id": "rs17gmh5wafm5p" }, ... ]
Contoh fragmen autoprotocolPengaturan python untuk biologi molekuler
Selain mengimpor perpustakaan standar, saya akan memerlukan beberapa utilitas biologi molekuler tertentu. Kode ini terutama untuk protokol otomatis dan Transkriptik.
Konsep "volume mati" sering ditemukan dalam kode. Ini berarti setetes cairan terakhir yang tidak dapat diambil oleh robot Transkriptik dengan pipet dari tabung (karena mereka tidak dapat melihatnya!). Anda harus menghabiskan banyak waktu untuk memastikan bahwa labu memiliki bahan yang cukup.
Kode import autoprotocol from autoprotocol import Unit from autoprotocol.container import Container from autoprotocol.protocol import Protocol from autoprotocol.protocol import Ref
Sintesis DNA dan biologi sintetis
Terlepas dari hubungannya dengan biologi sintetis modern, sintesis DNA adalah teknologi yang cukup tua. Selama beberapa dekade, kami telah mampu membuat oligonukleotida (yaitu sekuens DNA hingga 200 basa). Namun, itu selalu mahal, dan kimia tidak pernah mengizinkan urutan DNA yang panjang. Baru-baru ini, menjadi mungkin dengan harga yang wajar untuk mensintesis seluruh gen (hingga ribuan basis). Prestasi ini benar-benar membuka era "biologi sintetis".
Genomik Sintetis dari Craig Venter
telah mendorong biologi sintetis yang terjauh dengan
mensintesis seluruh organisme - lebih dari sejuta basa. Seiring meningkatnya panjang DNA, masalahnya bukan lagi sintesis, tetapi perakitan (yaitu, menyatukan urutan DNA yang disintesis). Dengan setiap perakitan, Anda dapat menggandakan panjang DNA (atau lebih), jadi setelah selusin atau lebih iterasi, Anda mendapatkan
molekul yang agak panjang ! Perbedaan antara sintesis dan perakitan harus segera menjadi jelas bagi pengguna akhir.
Hukum Moore?
Harga sintesis DNA turun cukup cepat, dari lebih dari $ 0,30 setahun yang lalu dua menjadi sekitar $ 0,10 hari ini, tetapi sedang berkembang lebih seperti bakteri daripada prosesor. Sebaliknya, harga sekuensing DNA jatuh lebih cepat dari hukum Moore. Target
$ 0,02 per basis ditetapkan sebagai titik belok di mana Anda dapat mengganti banyak manipulasi DNA yang menghabiskan waktu dengan sintesis sederhana. Misalnya, pada harga ini, Anda dapat mensintesis seluruh plasmid 3kb seharga
$ 60 dan melewatkan sekelompok biologi molekuler. Saya harap kami akan mencapai ini dalam beberapa tahun.
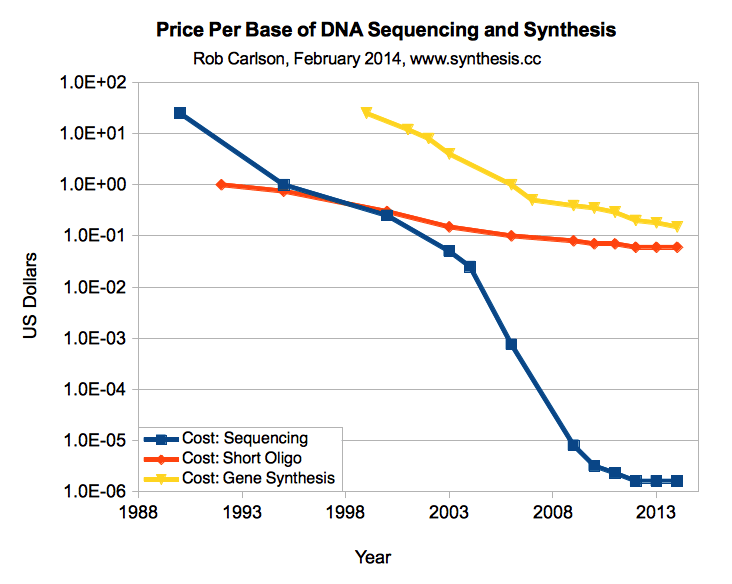 Harga sintesis DNA dibandingkan dengan harga pengurutan DNA, harga untuk 1 basis (Carlson, 2014)
Harga sintesis DNA dibandingkan dengan harga pengurutan DNA, harga untuk 1 basis (Carlson, 2014)Perusahaan sintesis DNA
Ada beberapa perusahaan besar di bidang sintesis DNA: IDT adalah produsen oligonukleotida terbesar, dan juga dapat memproduksi "fragmen gen" (
g hingga 2 kb ) lebih
lama (
gBlocks ).
Gen9 ,
Twist, dan
DNA 2.0 biasanya berspesialisasi dalam urutan DNA yang lebih panjang - ini adalah perusahaan sintesis gen. Ada juga beberapa perusahaan baru yang menarik, seperti
Cambrian Genomics dan
Genesis DNA , yang bekerja pada metode sintesis generasi selanjutnya.
Perusahaan lain, seperti
Amyris ,
Zymergen, dan
Ginkgo Bioworks , menggunakan DNA yang disintesis oleh perusahaan-perusahaan ini untuk bekerja di tingkat tubuh.
Genomik sintetis melakukan ini juga, tetapi ia mensintesis DNA itu sendiri.
Ginkgo baru
- baru ini
membuat kesepakatan dengan Twist untuk menghasilkan 100 juta pangkalan: kesepakatan terbesar yang pernah saya lihat. Ini membuktikan bahwa kita hidup di masa depan, Twist bahkan mengiklankan kode promosi di Twitter: ketika Anda membeli 10 juta basis DNA (hampir seluruh genom ragi!), Anda mendapatkan 10 juta lainnya secara gratis.
 Penawaran Twist Niche Twitter
Penawaran Twist Niche TwitterBagian Satu: Desain Eksperimen
Protein fluoresen hijau
Dalam percobaan ini, kami mensintesis urutan DNA untuk
protein hijau sederhana (GFP). Protein GFP pertama kali ditemukan dalam
ubur -
ubur yang berfluoresensi di bawah sinar ultraviolet. Ini adalah protein yang sangat berguna karena mudah untuk mendeteksi ekspresinya hanya dengan mengukur fluoresensi. Ada opsi GFP yang menghasilkan warna kuning, merah, oranye, dan lainnya.
Sangat menarik untuk melihat bagaimana berbagai mutasi mempengaruhi warna protein, dan ini merupakan masalah pembelajaran mesin yang berpotensi menarik. Baru-baru ini, Anda harus menghabiskan banyak waktu di laboratorium untuk ini, tetapi sekarang saya akan menunjukkan kepada Anda bahwa itu (hampir) semudah mengedit file teks!
Secara teknis, GFP saya adalah Opsi Folder Super (sfGFP) dengan beberapa mutasi untuk meningkatkan kualitas.
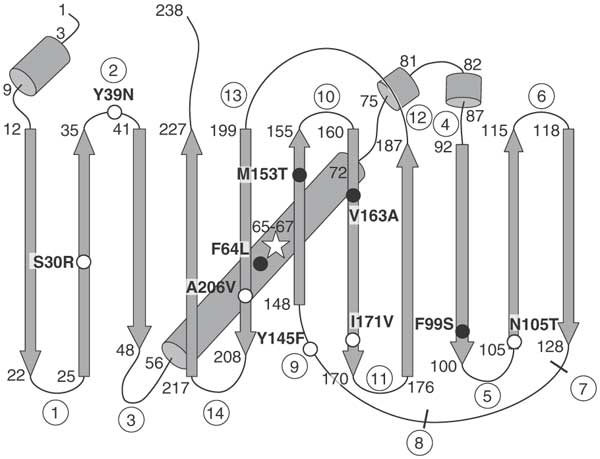 Dalam superfolder-GFP (sfGFP), beberapa mutasi memberikan properti berguna tertentu.
Dalam superfolder-GFP (sfGFP), beberapa mutasi memberikan properti berguna tertentu.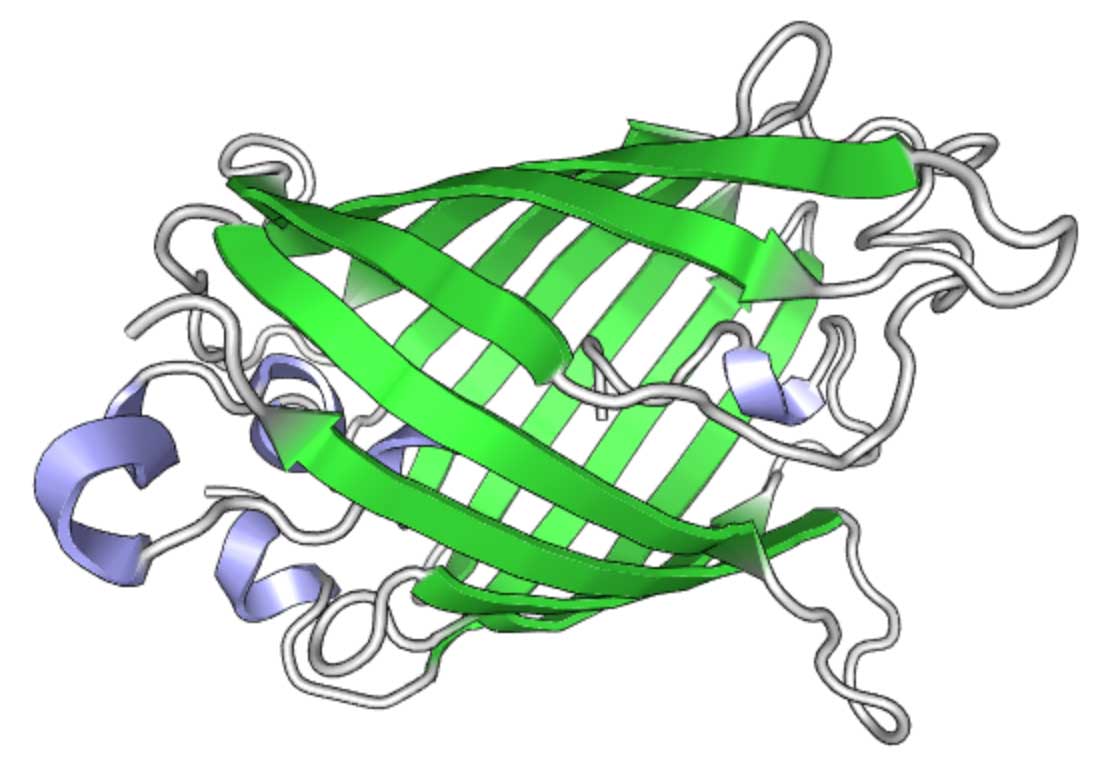 Struktur GFP (divisualisasikan menggunakan PV )
Struktur GFP (divisualisasikan menggunakan PV )Sintesis GFP dalam Twist
Saya cukup beruntung bisa masuk ke program pengujian alpha Twist, jadi saya menggunakan layanan sintesis DNA mereka (mereka dengan ramah memesan pesanan kecil saya - terima kasih, Twist!). Ini adalah perusahaan baru di bidang kami, dengan proses sintesis baru yang disederhanakan. Harga mereka sekitar
$ 0,10 per basis atau lebih rendah , tetapi mereka
masih dalam versi beta , dan program alpha tempat saya berpartisipasi ditutup. Twist mengumpulkan sekitar $ 150 juta, sehingga teknologi mereka hidup.
Saya mengirim urutan DNA saya ke Twist sebagai lembar kerja Excel (belum ada API, tapi saya kira itu akan segera), dan mereka mengirim DNA yang disintesis langsung ke kotak saya di laboratorium Transkriptik (saya juga menggunakan IDT untuk sintesis, tetapi mereka tidak mengirim IDT untuk sintesis, tetapi mereka tidak mengirim DNA tepat di Transkriptik, yang sedikit merusak kesenangan).
Jelas, proses ini belum menjadi kasus penggunaan yang khas dan memerlukan beberapa dukungan, tetapi berhasil, sehingga seluruh pipa tetap virtual. Tanpa ini, saya mungkin perlu akses ke laboratorium - banyak perusahaan tidak akan mengirim DNA atau reagen ke alamat rumah mereka.
 GFP tidak berbahaya, jadi apa pun yang disorot
GFP tidak berbahaya, jadi apa pun yang disorotVektor plasmid
Untuk mengekspresikan protein ini dalam bakteri, gen perlu hidup di suatu tempat, jika tidak, DNA sintetis yang mengkode gen hanya terdegradasi secara instan. Sebagai aturan, dalam biologi molekuler kami menggunakan plasmid, sepotong DNA bundar yang hidup di luar genom bakteri dan mengekspresikan protein. Plasmid adalah cara yang mudah bagi bakteri untuk berbagi modul fungsional yang berguna dan berdiri sendiri, seperti resistensi antibiotik. Mungkin ada ratusan plasmid di dalam sel.
Terminologi yang banyak digunakan adalah bahwa plasmid adalah
vektor , dan DNA sintetis adalah insersi (insersi). Jadi, di sini kita mencoba mengkloning penyisipan ke dalam vektor, dan kemudian mengubah bakteri menggunakan vektor.
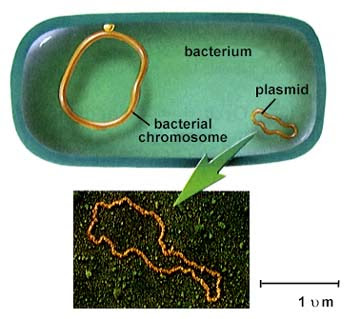 Genom bakteri dan plasmid (tidak untuk skala!) ( Wikipedia )
Genom bakteri dan plasmid (tidak untuk skala!) ( Wikipedia )pUC19
Saya memilih plasmid yang cukup standar di seri
pUC19 . Plasmid ini sangat sering digunakan, dan karena tersedia sebagai bagian dari inventaris Transkriptik standar, kita tidak perlu mengirim apa pun kepada mereka.
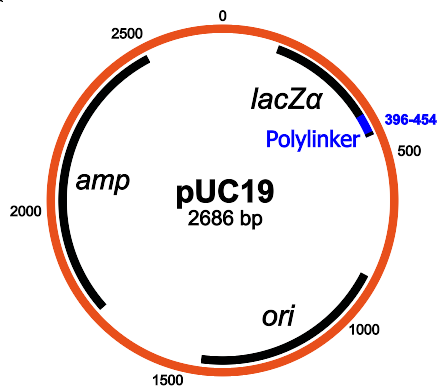 Struktur pUC19: komponen utamanya adalah gen resistensi ampisilin, lacZα, MCS / polylinker, dan asal replikasi (Wikipedia)
Struktur pUC19: komponen utamanya adalah gen resistensi ampisilin, lacZα, MCS / polylinker, dan asal replikasi (Wikipedia)PUC19 memiliki fungsi yang bagus: karena mengandung gen lacZα, Anda dapat menggunakan metode
seleksi biru-putih di atasnya dan melihat di mana koloni penyisipan berhasil. Dibutuhkan dua bahan kimia:
IPTG dan
X-gal , dan sirkuit berfungsi sebagai berikut:
- IPTG menginduksi ekspresi lacZα.
- Jika lacZα dinonaktifkan melalui DNA yang dimasukkan di beberapa situs kloning ( MCS / polylinker ) di lacZα, maka plasmid tidak dapat menghidrolisis X-gal dan koloni ini akan berwarna putih alih-alih biru.
- Oleh karena itu, penyisipan yang berhasil menghasilkan koloni putih, dan penyisipan yang gagal menghasilkan koloni biru.
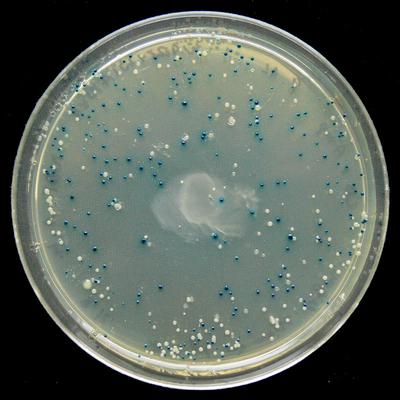 Pilihan biru dan putih menunjukkan di mana ekspresi lacZα dinonaktifkan ( Wikipedia )Dokumentasi openwetware
Pilihan biru dan putih menunjukkan di mana ekspresi lacZα dinonaktifkan ( Wikipedia )Dokumentasi openwetware mengatakan:
E. coli DH5α tidak memerlukan IPTG untuk menginduksi ekspresi dari promotor lac, bahkan jika represor Lac diekspresikan dalam strain. Jumlah salinan sebagian besar plasmid melebihi jumlah represor dalam sel. Jika Anda membutuhkan ekspresi maksimum, tambahkan IPTG ke konsentrasi akhir 1 mM.
Urutan DNA Sintetis
Urutan DNA SfGFP
Mudah untuk mendapatkan urutan DNA untuk sfGFP dengan mengambil
urutan protein dan
mengkodekannya dengan kodon yang cocok untuk organisme inang (di sini,
E. coli ). Ini adalah protein berukuran sedang dengan 236 asam amino, sehingga pada 10 sen biaya sintesis DNA sekitar
$ 70 per basa.
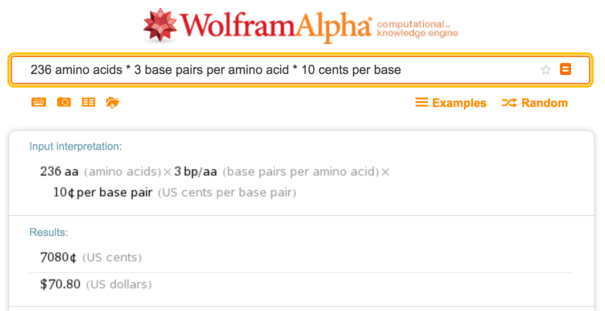 Wolfram Alpha, perhitungan biaya sintesis
Wolfram Alpha, perhitungan biaya sintesis12 basis pertama dari sfGFP kami adalah urutan
Shine-Delgarno , yang saya tambahkan sendiri, yang secara teoritis harus meningkatkan ekspresi (AGGAGGACAGCT, lalu ATG (
start kodon ) meluncurkan protein). Menurut alat komputasi yang dikembangkan oleh
Salis Lab (
slide kuliah ), kita dapat mengharapkan ekspresi protein kita sedang hingga tinggi (terjemahan tingkat inisiasi 10.000 "unit sewenang-wenang").
sfGFP_plus_SD = clean_seq(""" AGGAGGACAGCTATGTCGAAAGGAGAAGAACTGTTTACCGGTGTGGTTCCGATTCTGGTAGAACTGGA TGGGGACGTGAACGGCCATAAATTTAGCGTCCGTGGTGAGGGTGAAGGGGATGCCACAAATGGCAAAC TTACCCTTAAATTCATTTGCACTACCGGCAAGCTGCCGGTCCCTTGGCCGACCTTGGTCACCACACTG ACGTACGGGGTTCAGTGTTTTTCGCGTTATCCAGATCACATGAAACGCCATGACTTCTTCAAAAGCGC CATGCCCGAGGGCTATGTGCAGGAACGTACGATTAGCTTTAAAGATGACGGGACCTACAAAACCCGGG CAGAAGTGAAATTCGAGGGTGATACCCTGGTTAATCGCATTGAACTGAAGGGTATTGATTTCAAGGAA GATGGTAACATTCTCGGTCACAAATTAGAATACAACTTTAACAGTCATAACGTTTATATCACCGCCGA CAAACAGAAAAACGGTATCAAGGCGAATTTCAAAATCCGGCACAACGTGGAGGACGGGAGTGTACAAC TGGCCGACCATTACCAGCAGAACACACCGATCGGCGACGGCCCGGTGCTGCTCCCGGATAATCACTAT TTAAGCACCCAGTCAGTGCTGAGCAAAGATCCGAACGAAAAACGTGACCATATGGTGCTGCTGGAGTT CGTGACCGCCGCGGGCATTACCCATGGAATGGATGAACTGTATAAA""") print("Read in sfGFP plus Shine-Dalgarno: {} bases long".format(len(sfGFP_plus_SD))) sfGFP_aas = clean_aas("""MSKGEELFTGVVPILVELDGDVNGHKFSVRGEGEGDATNGKLTLKFICTTGKLPVPWPTLVTTLTYG VQCFSRYPDHMKRHDFFKSAMPEGYVQERTISFKDDGTYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNFNSHNVYITADKQKN GIKANFKIRHNVEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSVLSKDPNEKRDHMVLLEFVTAAGITHGMDELYK""") assert sfGFP_plus_SD[12:].translate() == sfGFP_aas print("Translation matches protein with accession 532528641")
Baca dalam sfGFP plus Shine-Dalgarno: 726 pangkalan
Terjemahan cocok dengan protein dengan aksesi 532528641
Urutan DNA PUC19
Pertama, saya memeriksa bahwa
urutan pUC19 yang saya unduh dari NEB memiliki panjang yang benar dan itu termasuk
polylinker yang diharapkan.
pUC19_fasta = !cat puc19fsa.txt pUC19_fwd = clean_seq(''.join(pUC19_fasta[1:])) pUC19_rev = pUC19_fwd.reverse_complement() assert all(nt in "ACGT" for nt in pUC19_fwd) assert len(pUC19_fwd) == 2686 pUC19_MCS = clean_seq("GAATTCGAGCTCGGTACCCGGGGATCCTCTAGAGTCGACCTGCAGGCATGCAAGCTT") print("Read in pUC19: {} bases long".format(len(pUC19_fwd))) assert pUC19_MCS in pUC19_fwd print("Found MCS/polylinker")
Baca dalam pUC19: 2686 pangkalan lama
Ditemukan MCS / polylinker
Kami melakukan beberapa QC dasar untuk memastikan bahwa EcoRI dan BamHI hadir dalam pUC19 hanya sekali (enzim restriksi berikut tersedia dalam persediaan Transkriptik default:
PstI ,
PvuII ,
EcoRI ,
BamHI ,
BbsI ,
BsmBI ).
REs = {"EcoRI":"GAATTC", "BamHI":"GGATTC"} for rename, res in REs.items(): assert (pUC19_fwd.find(res) == pUC19_fwd.rfind(res) and pUC19_rev.find(res) == pUC19_rev.rfind(res)) assert (pUC19_fwd.find(res) == -1 or pUC19_rev.find(res) == -1 or pUC19_fwd.find(res) == len(pUC19_fwd) - pUC19_rev.find(res) - len(res)) print("Asserted restriction enzyme sites present only once: {}".format(REs.keys()))
Sekarang kita melihat urutan lacZα dan memverifikasi bahwa tidak ada yang tidak terduga. Misalnya, harus dimulai dengan Met dan diakhiri dengan kodon stop. Juga mudah untuk mengkonfirmasi bahwa ini adalah ORF 324bp lacZα penuh dengan memuat urutan pUC19 ke
penampil snapgene gratis.
lacZ = pUC19_rev[2217:2541] print("lacZα sequence:\t{}".format(lacZ)) print("r_MCS sequence:\t{}".format(pUC19_MCS.reverse_complement())) lacZ_p = lacZ.translate() assert lacZ_p[0] == "M" and not "*" in lacZ_p[:-1] and lacZ_p[-1] == "*" assert pUC19_MCS.reverse_complement() in lacZ assert pUC19_MCS.reverse_complement() == pUC19_rev[2234:2291] print("Found MCS once in lacZ sequence")
lacZ berurutan: ATGACCATGATTACGCCAAGCTTGCATGCCTGCAGGTCGACTCTAGAGGATCCCCGGGTACCGAGCTCGAATTCACTGGCCGTCGTTTTACAACGTCGTGACTGGGAAAACCCTGGCGTTACCCAACTTAATCGCCTTGCAGCACATCCCCCTTTCGCCAGCTGGCGTAATAGCGAAGAGGCCCGCACCGATCGCCCTTCCCAACAGTTGCGCAGCCTGAATGGCGAATGGCGCCTGATGCGGTATTTTCTCCTTACGCATCTGTGCGGTATTTCACACCGCATATGGTGCACTCTCAGTACAATCTGCTCTGATGCCGCATAG
Urutan r_MCS: AAGCTTGCATGCCTGCAGGTCGACTCTAGAGGATCCCCGGGGTACCGAGCTCGAATTC
Ditemukan MCS sekali dalam urutan lacZ
Perakitan Gibson
Merakit DNA berarti mengaitkan fragmen. Biasanya Anda mengumpulkan beberapa fragmen DNA ke dalam segmen yang lebih panjang, dan kemudian mengkloningnya menjadi plasmid atau genom. Dalam percobaan ini, saya ingin mengkloning satu segmen DNA ke dalam plasmid pUC19 di bawah promotor lac untuk ekspresi dalam
E.coli .
Ada banyak metode kloning (mis.
NEB ,
openwetware ,
addgene ). Di sini saya akan menggunakan perakitan Gibson (
dikembangkan oleh Daniel Gibson dalam Synthetic Genomics pada 2009), yang belum tentu metode termurah, tetapi sederhana dan fleksibel. Anda hanya perlu meletakkan DNA yang ingin Anda kumpulkan (dengan tumpang tindih yang sesuai) dalam tabung reaksi dengan Campuran Master Majelis Gibson, dan itu akan berkumpul sendiri!
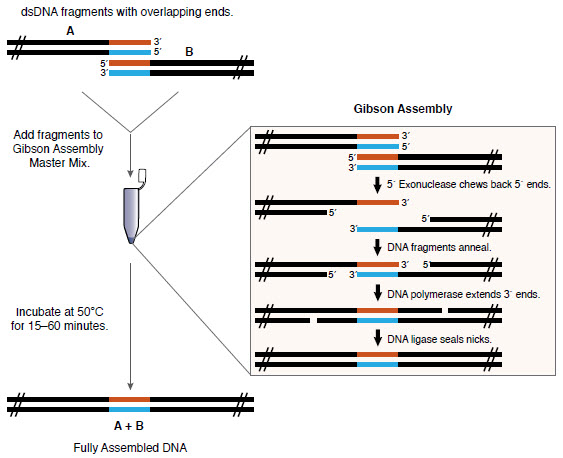 Gibson Assembly Review ( NEB )
Gibson Assembly Review ( NEB )Bahan sumber
Kami mulai dengan 100 ng DNA sintetis dalam 10 μl cairan. Ini sama dengan 0,21 picomoles DNA atau konsentrasi 10 ng / μl.
pmol_sfgfp = convert_ug_to_pmol(0.1, len(sfGFP_plus_SD)) print("Insert: 100ng of DNA of length {:4d} equals {:.2f} pmol".format(len(sfGFP_plus_SD), pmol_sfgfp))
Masukkan: 100ng DNA dengan panjang 726 sama dengan 0,21 pmol
Menurut
protokol perakitan NEB , ini adalah sumber materi yang cukup:
NEB merekomendasikan total 0,02-0,5 picomoles fragmen DNA ketika 1 atau 2 fragmen berkumpul ke dalam vektor, atau 0,2-1,0 picomoles fragmen DNA ketika 4-6 fragmen dikumpulkan.
0,02-0,5 pmol * X μl
* Efisiensi kloning yang dioptimalkan adalah 50-100 ng vektor dengan 2-3 kali lipat penyisipan. Gunakan sisipan 5 kali lebih banyak jika ukurannya kurang dari 200 bps. Total volume fragmen PCR tanpa filter dalam reaksi perakitan Gibson tidak boleh melebihi 20%.
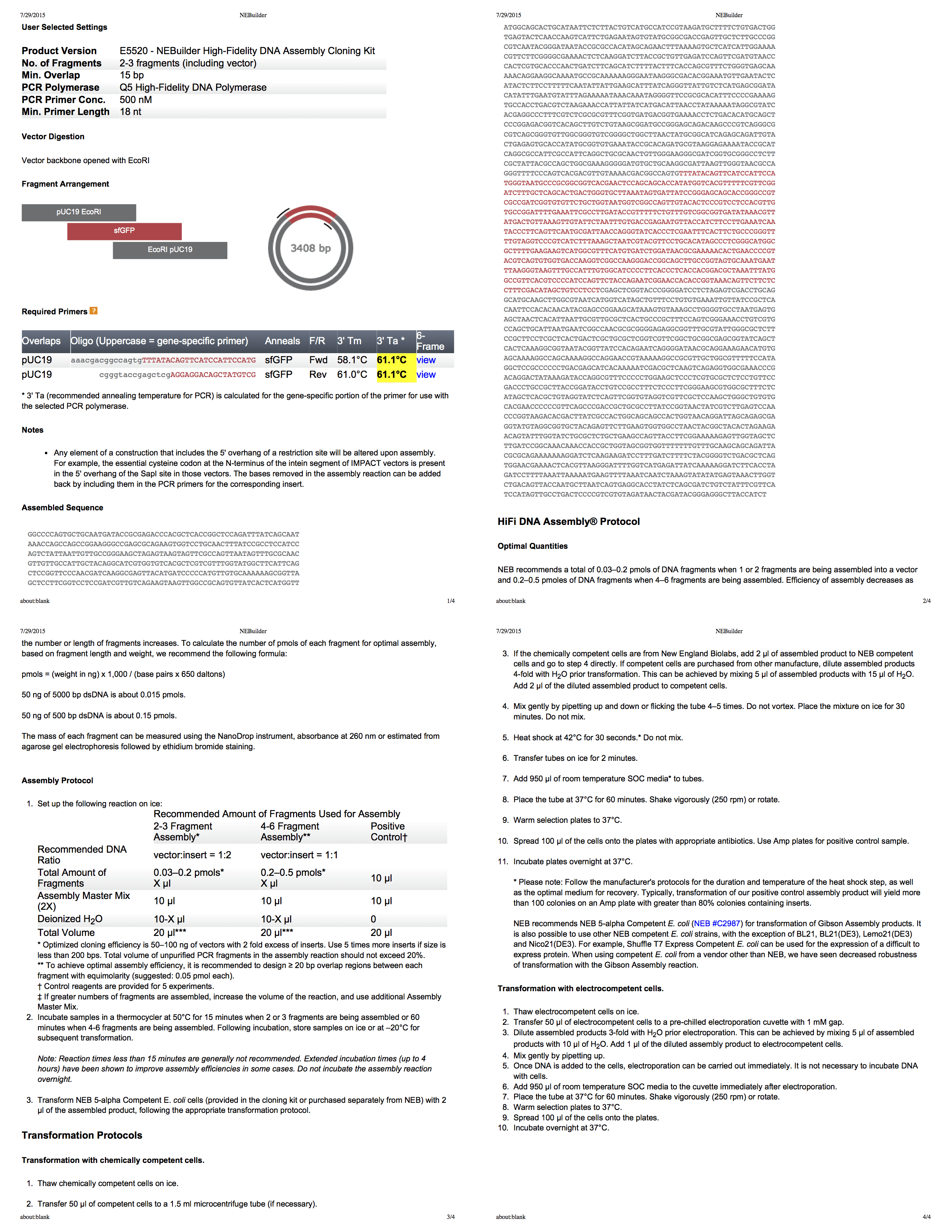
NEBuilder untuk perakitan Gibson
NEBuilder Biolab adalah alat yang sangat hebat untuk membuat protokol pembangunan Gibson. Bahkan menghasilkan Anda PDF empat halaman yang komprehensif dengan semua informasi. Dengan menggunakan alat ini, kami mengembangkan protokol untuk memotong pUC19 dengan EcoRI, dan kemudian menggunakan PCR [PCR, reaksi rantai polimerase memungkinkan untuk mencapai peningkatan signifikan dalam konsentrasi kecil dari fragmen DNA tertentu dalam bahan biologis - kira-kira. per.] untuk menambahkan fragmen dengan ukuran yang sesuai ke sisipan.
Bagian dua: percobaan
Eksperimen terdiri dari empat tahap:
- Reaksi penyisipan rantai polimerase untuk menambahkan bahan dengan urutan mengapit.
- Memotong plasmid untuk mengakomodasi penyisipan.
- Perakitan dengan penyisipan Gibson dan plasmid.
- Transformasi bakteri menggunakan plasmid rakitan.
Langkah 1. Penyisipan PCR
Perakitan Gibson tergantung pada urutan DNA yang Anda kumpulkan, memiliki beberapa urutan yang tumpang tindih (lihat protokol NEB dengan instruksi terperinci di atas). Selain amplifikasi sederhana, PCR juga memungkinkan Anda untuk menambahkan urutan DNA mengapit dengan hanya memasukkan urutan tambahan dalam primer (juga dapat dikloning
hanya menggunakan OE-PCR ).
Kami mensintesis primer berdasarkan protokol NEB di atas. Saya mencoba
protokol Quickstart di situs Transkriptik, tetapi masih
ada perintah auto- protokol . Transkriptik itu sendiri tidak mensintesis oligonukleotida, jadi setelah 1-2 hari menunggu, primer-primer ini secara ajaib muncul dalam inventaris saya (perhatikan bahwa bagian spesifik gen dari primer ditunjukkan dalam huruf besar di bawah, tetapi ini hanyalah hal-hal kosmetik).
insert_primers = ["aaacgacggccagtgTTTATACAGTTCATCCATTCCATG", "cgggtaccgagctcgAGGAGGACAGCTATGTCG"]
Analisis Primer
Anda dapat menganalisis properti dari primer ini menggunakan
IDT OligoAnalyzer .
ketika men-debug eksperimen PCR, penting untuk mengetahui titik lebur dan kemungkinan efek samping dari dimer primer , meskipun protokol NEB hampir pasti akan memilih primer dengan sifat yang baik. Bagian panggul khusus gen (huruf besar)
Suhu leleh: 51C, 53.5C
Urutan penuh
Temperatur leleh: 64.5C, 68.5C
Jepit rambut: -.4dG, -5dG
Peredup diri otomatis: -9dG, -16dG
Heterodimer: -6dG
Saya telah melalui banyak iterasi PCR sebelum mendapatkan hasil yang memuaskan, termasuk percobaan dengan beberapa merek campuran PCR. Karena setiap iterasi ini dapat memakan waktu beberapa hari (tergantung pada panjang antrian ke laboratorium), ada baiknya menghabiskan waktu untuk debugging di muka: ini menghemat banyak waktu dalam jangka panjang. Ketika kekuatan lab cloud meningkat, masalah ini seharusnya menjadi kurang akut. Namun, sepertinya protokol pertama Anda tidak akan berhasil - ada terlalu banyak variabel.Kode """ PCR overlap extension of sfGFP according to NEB protocol. v5: Use 3/10ths as much primer as the v4 protocol. v6: more complex touchdown pcr procedure. The Q5 temperature was probably too hot v7: more time at low temperature to allow gene-specific part to anneal v8: correct dNTP concentration, real touchdown """ p = Protocol()
PERINGATAN: root: Volume rendah untuk sumur sfGFP 1 / sfGFP 1: 2.0: mikroliter
sfGFP 1 / sfGFP 1 2.0: microliter {'dilution': '0.25ng / ul'}
sfgfp_pcroe_v5_puc19_primer1_10uM 75.0: microliter {}
sfgfp_pcroe_v5_puc19_primer2_10uM 75.0: microliter {}
Volume konsolidasi 52.0: mikroliter
Protocol 1. Amplify the insert (oligos previously synthesized)
---------------------------------------------------------------
✓ Protocol analyzed
11 instructions
8 containers
Total Cost: $32.18
Workcell Time: $4.32
Reagents & Consumables: $27.86 : PCR
Dalam gel, Anda dapat mengevaluasi ukuran produk yang benar setelah meningkatkan konsentrasi (posisi strip dalam gel) dan jumlah yang benar (strip gelap). Gel memiliki tangga yang sesuai dengan berbagai panjang dan jumlah DNA yang dapat digunakan untuk perbandingan.Dalam foto gel di bawah ini, pita D1, E1, F1 masing-masing mengandung 2 μl, 4 μl, dan 8 μl dari produk yang diamplifikasi. Saya dapat memperkirakan jumlah DNA di setiap jalur dibandingkan dengan DNA di tangga (50 ng DNA per jalur di tangga). Saya pikir hasilnya terlihat sangat bersih.Saya mencoba menggunakan GelEval untuk analisis gambar dan estimasi konsentrasi, dan cukup berhasil, walaupun saya tidak yakin apakah ini jauh lebih akurat daripada metode yang lebih naif. Namun, perubahan kecil pada lokasi dan ukuran pita menyebabkan perubahan besar dalam estimasi jumlah DNA. Perkiraan terbaik saya tentang jumlah DNA dalam produk teramplifikasi saya menggunakan GelEval adalah 40 ng / μl.Dengan asumsi kita dibatasi oleh jumlah primer dalam campuran dan bukan oleh jumlah dNTP atau enzim, karena saya memiliki 12,5 pmol dari masing-masing primer, ini berarti maksimum teoritis 6 μg DNA 740bp dalam 25 μl. Karena perkiraan saya tentang jumlah total DNA menggunakan GelEval adalah 40 ng x 25 μl (1 μg atau 2 pmol), hasil ini sangat masuk akal dan dekat dengan apa yang seharusnya saya harapkan dalam kondisi ideal.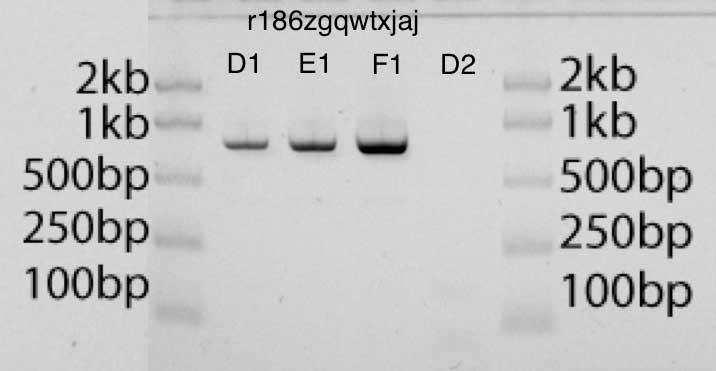 - EcoRI- pUC19, (D1, E1, F1), (D2)
- EcoRI- pUC19, (D1, E1, F1), (D2)PCR
Transkriptik baru-baru ini mulai menyediakan data diagnostik yang menarik dan berguna dari robotnya. Pada saat penulisan, mereka tidak tersedia untuk diunduh, jadi untuk saat ini saya hanya memiliki gambar suhu selama siklus termal.Data terlihat bagus, tanpa puncak atau palung yang tidak terduga. Sebanyak 35 siklus PCR, tetapi beberapa siklus ini dilakukan pada suhu yang sangat tinggi sebagai bagian dari touchdown PCR . Dalam upaya saya sebelumnya untuk memperkuat segmen ini - yang ada beberapa! - ada masalah dengan hibridisasi primer, jadi di sini PCR bekerja untuk banyak waktu pada suhu tinggi, yang seharusnya meningkatkan akurasi. Diagnostik termosiklik untuk touchdown PCR: suhu blok, sampel dan penutup selama 35 siklus dan 42 menit
Diagnostik termosiklik untuk touchdown PCR: suhu blok, sampel dan penutup selama 35 siklus dan 42 menitLangkah 2. Memotong plasmid
Untuk memasukkan DNA sfGFP kami ke pUC19, Anda harus terlebih dahulu memotong plasmid. Mengikuti protokol NEB, saya melakukan ini menggunakan enzim restriksi EcoRI . Dalam inventaris Transkriptik standar ada reagen yang saya butuhkan: ini adalah NEB EcoRI dan buffer CutSmart 10x , serta NEB pUC19 plasmid .Sebagai informasi, di bawah ini adalah harga dari inventaris mereka. Bahkan, saya hanya membayar sebagian dari harga, karena Transkriptik mengambil pembayaran untuk jumlah yang sebenarnya dikonsumsi: Item ID Jumlah Harga Konsentrasi
------------ ------ ------------- ----------------- - ----
CutSmart 10x B7204S 5 ml 10 X $ 19,00
EcoRI R3101L 50.000 unit 20.000 unit / ml $ 225,00
pUC19 N3041L 250 μg 1.000 μg / ml $ 268.00
Saya mengikuti protokol NEB sebanyak mungkin:. 10X dH2O 1X. , , , , . 50 5 10x NEBuffer , dH2O.
, 1 λ 1 37°C 50 . , 5-10 10-20 1- .
1 50 .
Kode """Protocol for cutting pUC19 with EcoRI.""" p = Protocol() experiment_name = "puc19_ecori_v3" options = {} inv = { 'water': "rs17gmh5wafm5p",
Volumes: re_tube:135.0:microliter water_tube:383.0:microliter EcoRI:30.0:microliter
Consolidated volume: 78.0:microliter
✓ Protocol analyzed
12 instructions
5 containers
Total Cost: $30.72
Waktu Kerja: $ 3,38
Reagen & Habis: $ 27,34
Hasil: pemotongan plasmid
Saya melakukan percobaan ini dua kali dalam kondisi yang sedikit berbeda dan dengan ukuran gel yang berbeda, tetapi hasilnya hampir sama. Saya suka kedua gel.Awalnya, saya tidak mengalokasikan cukup ruang untuk volume "mati" (dalam tabung reaksi 1,5 ml, volume mati adalah 15 μl!). Saya pikir ini menjelaskan perbedaan antara D1 dan E1 (kedua band harus identik). Masalah volume mati mudah untuk dipecahkan dengan menciptakan pasokan kerja yang sesuai dari EcoRI yang diencerkan pada awal protokol.Terlepas dari kesalahan ini, di kedua gel, pita D1 dan E1 memiliki pita kuat di posisi yang benar dari 2.6kb. Pada pita D2, plasmid yang tidak dipotong: seperti yang diharapkan, tidak terlihat dalam satu gel dan nyaris tidak terlihat pada yang lain.Dua foto gel terlihat sangat berbeda. Ini sebagian disebabkan oleh fakta bahwa langkah Transkriptik ini belum terotomatisasi.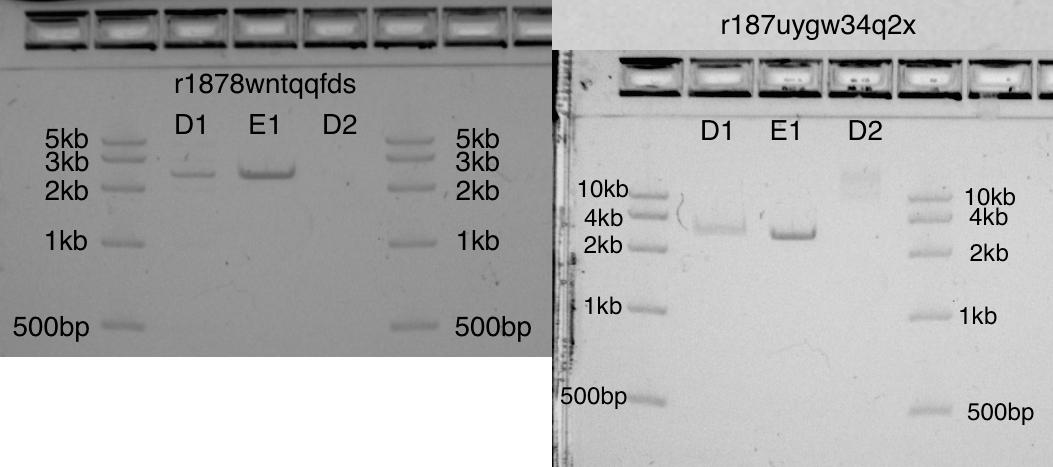 Dua gel menunjukkan cut pUC19 (2.6kb) di band D1 dan E1, dan pUC19 yang belum dipotong di D2
Dua gel menunjukkan cut pUC19 (2.6kb) di band D1 dan E1, dan pUC19 yang belum dipotong di D2Langkah 3. Perakitan Gibson
Cara termudah untuk memeriksa apakah perakitan saya bekerja menggunakan metode Gibson adalah untuk mengumpulkan penyisipan dan plasmid, kemudian menggunakan primer M13 standar (yang mengapit penyisipan) untuk memperkuat bagian dari plasmid dan memasukkan DNA dan menjalankan qPCR dan gel untuk memastikan bahwa amplifikasi bekerja. Anda juga dapat menjalankan reaksi sekuensing untuk mengonfirmasi bahwa semuanya dimasukkan seperti yang diharapkan, tetapi saya memutuskan untuk membiarkannya nanti.Jika perakitan Gibson tidak bekerja, maka amplifikasi M13 tidak bekerja karena plasmid dipotong antara dua sekuens M13.Kode """Debugging transformation protocol: Gibson assembly followed by qPCR and a gel v2: include v3 Gibson assembly""" p = Protocol() options = {} experiment_name = "debug_sfgfp_puc19_gibson_seq_v2" inv = { "water" : "rs17gmh5wafm5p",
PERINGATAN: root: Volume rendah untuk sumur sfgfp_puc19_gibson_v1_clone / sfgfp_puc19_gibson_v1_clone: 11.0: microliter
✓ Protokol dianalisis
11 instruksi
6 kontainer
Total Biaya: $ 32,09
Waktu Kerja: $ 6,98
Reagen & Habis: $ 25,11
Hasil: qPCR untuk perakitan Gibson
Saya bisa mengakses data qPCR dalam format JSON melalui Transcriptic API. Fitur ini tidak didokumentasikan dengan baik , tetapi bisa sangat berguna. API bahkan memberi Anda akses ke beberapa data diagnostik dari robot, yang dapat membantu dengan debugging.Pertama, kami meminta data peluncuran: project_id, run_id = "p16x6gna8f5e9", "r18mj3cz3fku7" api_url = "https://secure.transcriptic.com/hgbrian/{}/runs/{}/data.json".format(project_id, run_id) data_response = requests.get(api_url, headers=tsc_headers) data = data_response.json()
Kemudian kami menentukan id ini untuk mendapatkan data "pasca pemrosesan" qPCR: qpcr_id = data['debug_sfgfp_puc19_gibson_seq_v1_qpcr']['id'] pp_api_url = "https://secure.transcriptic.com/data/{}.json?key=postprocessed_data".format(qpcr_id) data_response = requests.get(pp_api_url, headers=tsc_headers) pp_data = data_response.json()
Berikut adalah nilai Ct (cycle threshold) untuk setiap tabung. Ct hanyalah titik di mana fluoresensi melebihi nilai tertentu. Dia mengatakan kira-kira berapa banyak DNA yang ada saat ini (dan karena itu, kira-kira di mana kita mulai).
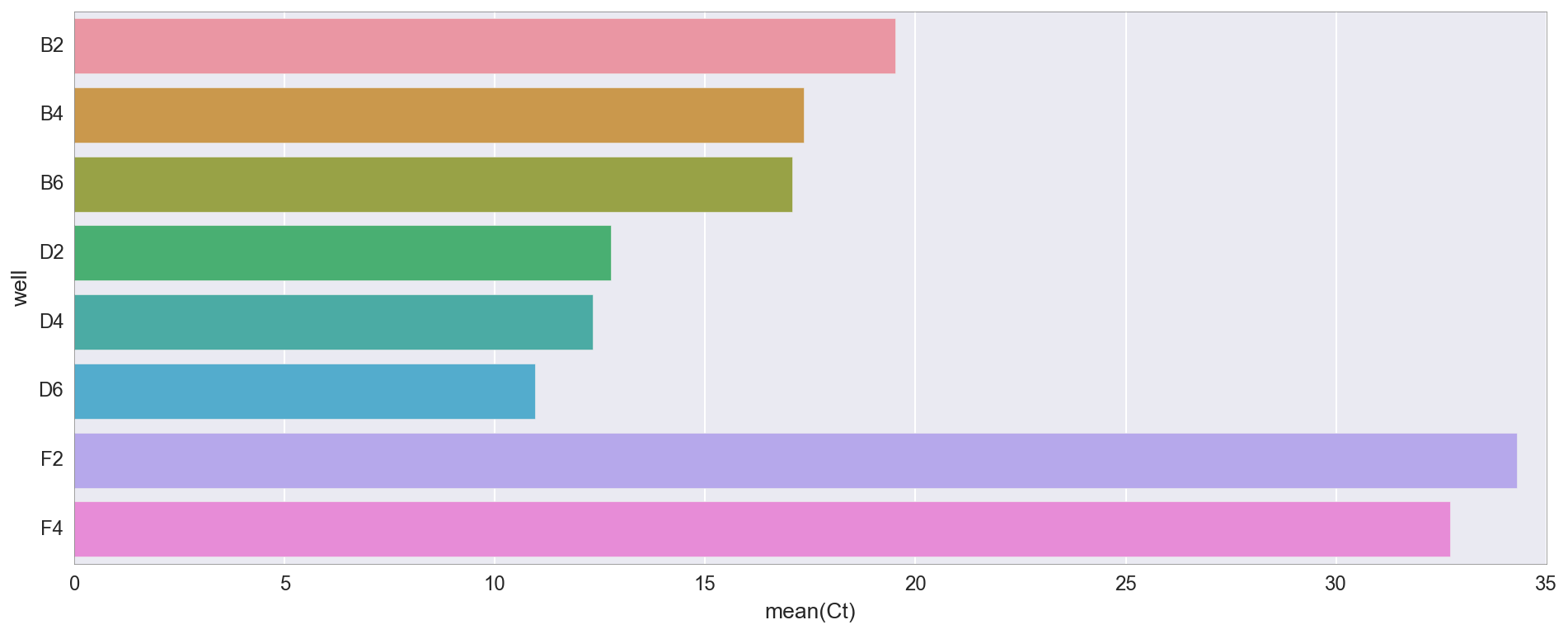 Seperti yang Anda lihat, amplifikasi terutama terjadi pada tabung uji D2 / 4/6 (di mana DNA dari rakitan Gibson saya adalah "v3"), kemudian B2 / 4/6 (rakitan Gibson adalah "v1"). Perbedaan antara v1 dan v3 pada dasarnya bahwa DNA v3 diencerkan 4X sesuai dengan protokol NEB, tetapi kedua opsi harus bekerja. Ada beberapa amplifikasi setelah siklus 30 dalam tabung kontrol (F2, F4) yang tidak memiliki template DNA, tetapi ini tidak jarang, karena mereka memasukkan banyak DNA primer.Saya juga bisa menggambar kurva amplifikasi qPCR untuk melihat dinamika amplifikasi.
Seperti yang Anda lihat, amplifikasi terutama terjadi pada tabung uji D2 / 4/6 (di mana DNA dari rakitan Gibson saya adalah "v3"), kemudian B2 / 4/6 (rakitan Gibson adalah "v1"). Perbedaan antara v1 dan v3 pada dasarnya bahwa DNA v3 diencerkan 4X sesuai dengan protokol NEB, tetapi kedua opsi harus bekerja. Ada beberapa amplifikasi setelah siklus 30 dalam tabung kontrol (F2, F4) yang tidak memiliki template DNA, tetapi ini tidak jarang, karena mereka memasukkan banyak DNA primer.Saya juga bisa menggambar kurva amplifikasi qPCR untuk melihat dinamika amplifikasi. f, ax = plt.subplots(figsize=(16,6)) ax.set_color_cycle(['#fb6a4a', '#de2d26', '#a50f15', '#74c476', '#31a354', '#006d2c', '#08519c', '#6baed6']) amp0 = pp_data['amp0']['SYBR']['baseline_subtracted'] _ = [plt.plot(amp0[w_n[well]], label=well) for well in ['B2', 'B4', 'B6', 'D2', 'D4', 'D6', 'F2', 'F4']] _ = ax.set_ylim(0,) _ = plt.title("qPCR (reds=Gibson v1, greens=Gibson v3, blues=control)") _ = plt.legend(bbox_to_anchor=(1, .75), bbox_transform=plt.gcf().transFigure)
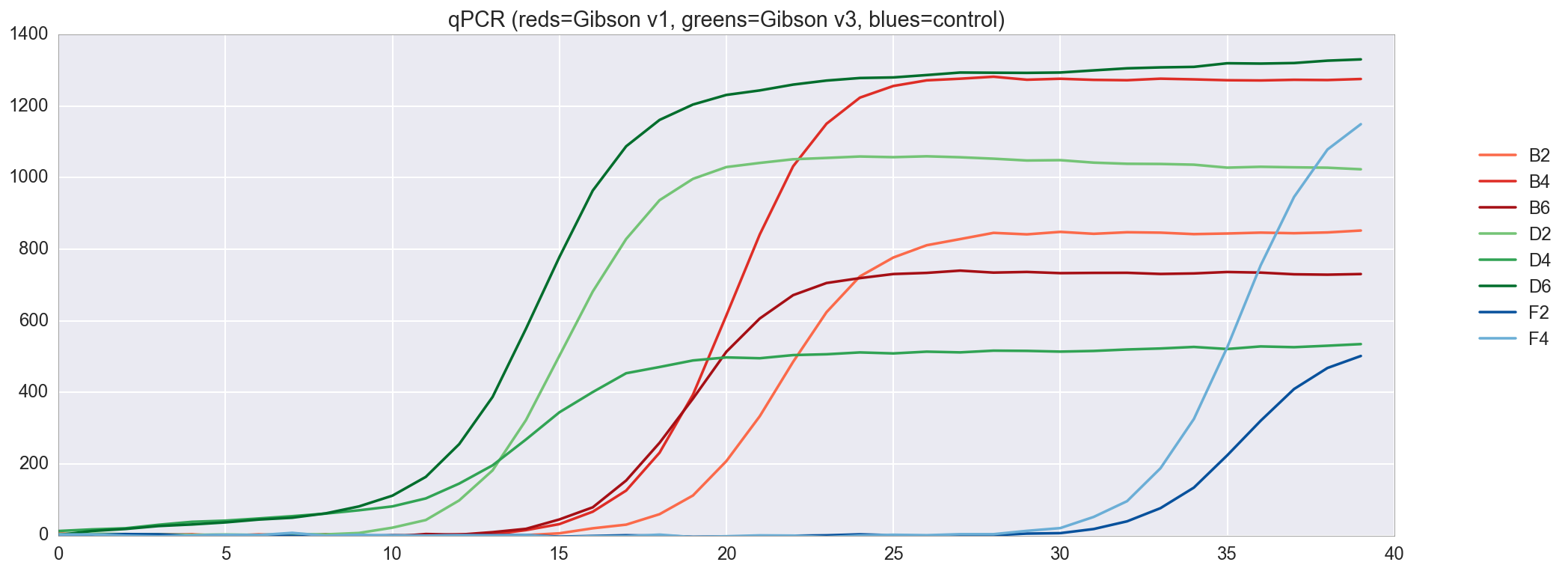 Secara umum, hasil qPCR terlihat hebat, dengan amplifikasi yang baik dari kedua versi perakitan Gibson saya dan tanpa amplifikasi nyata pada kelompok kontrol. Karena assembly v3 menunjukkan hasil yang sedikit lebih baik daripada v1, sekarang kita akan menggunakannya.
Secara umum, hasil qPCR terlihat hebat, dengan amplifikasi yang baik dari kedua versi perakitan Gibson saya dan tanpa amplifikasi nyata pada kelompok kontrol. Karena assembly v3 menunjukkan hasil yang sedikit lebih baik daripada v1, sekarang kita akan menggunakannya.Hasil: Perakitan Gibson dalam gel
Gel ini juga sangat bersih, menunjukkan pita kuat tepat di bawah 1kb pada pita B2, B4, B6, D2, D4, D6: ini adalah ukuran yang kami harapkan (penyisipan sekitar 740bp, dan primer M13 sekitar 40bp atas dan ke bawah). Jalur kedua sesuai dengan primer. Anda dapat yakin akan hal ini, karena pita F2 dan F4 hanya mengandung DNA primer.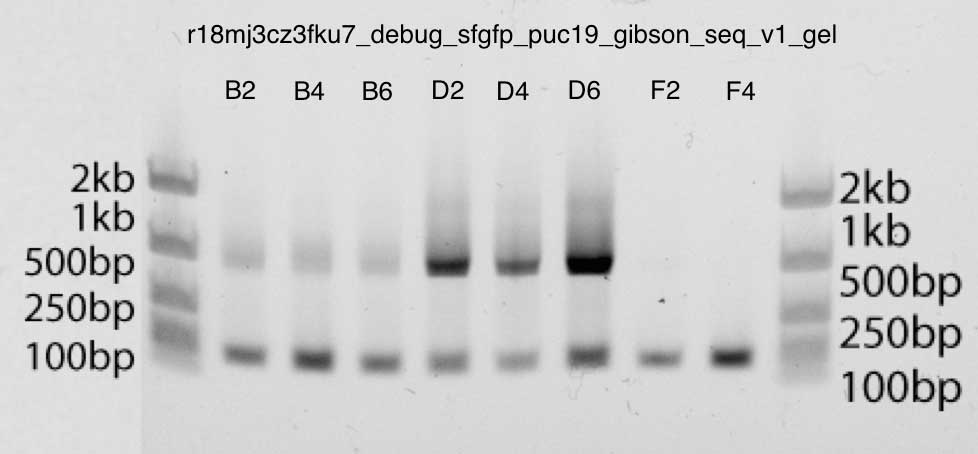 Elektroforesis gel poliakrilamid: c Gibson assembly v3 menunjukkan pita yang lebih kuat (D2, D4, D6), menurut data qPCR di atas
Elektroforesis gel poliakrilamid: c Gibson assembly v3 menunjukkan pita yang lebih kuat (D2, D4, D6), menurut data qPCR di atasLangkah 4. Transformasi
Transformasi adalah proses mengubah tubuh dengan menambahkan DNA. Dalam percobaan ini, kami mengubah E. coli menggunakan plasmid pUC19 yang mengekspresikan sfGFP.Kami menggunakan strain Zymo DH5α Mix & Go yang mudah digunakan dan protokol yang direkomendasikan . Jenis ini adalah bagian dari inventaris Transkriptik standar. Secara umum, transformasi bisa kompleks karena sel yang kompeten agak rapuh, sehingga protokol yang lebih sederhana dan lebih dapat diandalkan, semakin baik. Di laboratorium biologi molekuler biasa, sel-sel kompeten ini mungkin akan terlalu mahal untuk penggunaan umum. Zymo Mix & Go Cells dengan Simple Protocol
Zymo Mix & Go Cells dengan Simple ProtocolMasalah dengan robot
Protokol ini adalah contoh yang baik tentang betapa sulitnya untuk menyesuaikan protokol manusia untuk digunakan oleh robot dan bagaimana hal itu bisa gagal secara tak terduga. Protokol kadang-kadang secara mengejutkan tidak jelas ("ayunkan tabung dari sisi ke sisi") berdasarkan konteks umum dari ahli biologi molekuler, atau mereka mungkin tiba-tiba meminta pemrosesan gambar tingkat lanjut ("pastikan pelet dicampur"). Orang tidak keberatan dengan tugas seperti itu, tetapi robot membutuhkan instruksi yang lebih jelas.Transformasi ini menunjukkan masalah waktu yang menarik. Protokol transformasi menyarankan bahwa sel-sel tidak boleh tetap pada suhu kamar selama lebih dari beberapa detik dan pelat dengan tabung harus dipanaskan terlebih dahulu hingga 37 ° C. Secara teoritis, Anda ingin memulai pemanasan sehingga berakhir bersamaan dengan transformasi, tetapi tidak jelas bagaimana robot Transkriptik akan mengatasi situasi ini - sejauh yang saya tahu, tidak ada cara untuk menyinkronkan langkah protokol secara tepat. Kurangnya kontrol yang akurat dari waktu ke waktu tampaknya menjadi masalah umum dengan protokol robot karena tidak fleksibelnya lengan robot, konflik penjadwalan, dll. Kita harus menyesuaikan protokol sesuai dengan itu.Biasanya ada solusi yang masuk akal: kadang-kadang Anda hanya perlu menggunakan reagen yang berbeda (misalnya, sel yang lebih keras, seperti Mix & Go di atas); terkadang Anda hanya menggadaikan aksi dengan margin (misalnya, kocok sepuluh kali, bukan tiga); terkadang Anda perlu membuat trik khusus untuk robot (misalnya, gunakan mesin PCR untuk heat stroke).Tentu saja, keuntungan besar adalah sekali protokol telah bekerja sekali, Anda biasanya dapat mengandalkannya lagi dan lagi. Anda bahkan dapat mengukur seberapa andal protokol itu dan memperbaikinya seiring waktu!Tes transformasi
Sebelum memulai transformasi dengan plasmid yang dirakit penuh, saya melakukan percobaan sederhana untuk memastikan bahwa transformasi menggunakan pUC19 biasa (mis. Tanpa perakitan Gibson dan tanpa penyisipan sfGFP DNA) berfungsi. pUC19 mengandung gen resistensi ampisilin, sehingga transformasi yang berhasil seharusnya memungkinkan bakteri untuk tumbuh di piring yang mengandung antibiotik ini.Saya mentransfer bakteri langsung ke tablet ("6-flat" dalam terminologi Transkriptik), di mana ada ampisilin atau tidak. Saya berharap bahwa bakteri yang diubah mengandung gen resistensi ampisilin dan karenanya akan tumbuh. Bakteri non-transformasi tidak boleh tumbuh.Kode """Simple transformation protocol: transformation with unaltered pUC19""" p = Protocol() experiment_name = "debug_sfgfp_puc19_gibson_v1" inv = { "water" : "rs17gmh5wafm5p",
✓ Protokol dianalisis
43 instruksi
3 kontainer
$ 45,43
Hasil: uji transformasi
Dalam foto-foto berikut, kita melihat bahwa tanpa antibiotik (piring di sebelah kiri), pertumbuhan diamati pada semua enam piring, meskipun pada tingkat yang bervariasi, yang menyebabkan kekhawatiran. Tampaknya robot Transkriptik tidak benar-benar mengatasi distribusi seragam, yang memerlukan ketangkasan.Di hadapan antibiotik (piring di sebelah kanan), ada juga pertumbuhan, meskipun sekali lagi tidak konsisten. Dua lempeng pertama dengan antibiotik terlihat aneh, dengan pertumbuhan besar, yang mungkin merupakan hasil dari menambahkan 55 μl ke piring-piring ini dibandingkan dengan 10 μl di piring tanpa antibiotik. Ada beberapa koloni di piring ketiga, dan pada dasarnya inilah yang saya harapkan untuk dilihat di semua piring. Seharusnya ada beberapa pertumbuhan pada tiga lempeng terakhir, tetapi itu tidak terjadi. Satu-satunya penjelasan saya untuk hasil aneh ini adalah bahwa saya tidak mencampur sel dan medianya cukup, sehingga hampir semua sel masuk ke dalam dua lempeng pertama.(Saya masih harus membuat kontrol positif pada ampisilin dengan bakteri yang tidak berubah, tetapi saya sudah melakukan ini dalam percobaan sebelumnya, jadi saya tahu bahwa stok ampisilin harus membunuh jenis ini.E. coli . Pertumbuhan jauh lebih lemah pada pelat ampisilin, meskipun ada lebih banyak bakteri di sana, seperti yang diharapkan).Secara keseluruhan, transformasi berjalan cukup baik untuk melanjutkan, meskipun ada beberapa kekurangan. Piring sel diubah dengan pUC19 setelah 18 jam: tanpa antibiotik (kiri) dan dengan antibiotik (kanan)
Piring sel diubah dengan pUC19 setelah 18 jam: tanpa antibiotik (kiri) dan dengan antibiotik (kanan)Transformasi produk setelah perakitan
Karena perakitan Gibson dan transformasi pUC19 sederhana tampaknya berhasil, Anda sekarang dapat mencoba transformasi dengan sfGFP yang mengekspresikan plasmid yang dirakit lengkap.Selain penyisipan yang dikumpulkan, saya juga akan menambahkan sedikit IPTG dan X-gal ke piring untuk melihat konversi yang berhasil menggunakan metode seleksi biru dan putih . Informasi tambahan ini berguna, karena jika transformasi terjadi dengan pUC19 biasa, yang tidak mengandung sfGFP, itu masih akan memberikan resistensi antibiotik.Penyerapan dan fluoresensi
Menurut tabel ini , sfGFP bersinar terbaik pada panjang gelombang eksitasi 485 nm / 510 nm. Saya menemukan bahwa dalam Transkriptik, 485/535 berfungsi lebih baik. Saya kira karena 485 dan 510 terlalu mirip. Saya mengukur pertumbuhan bakteri pada 600 nm ( OD600 ).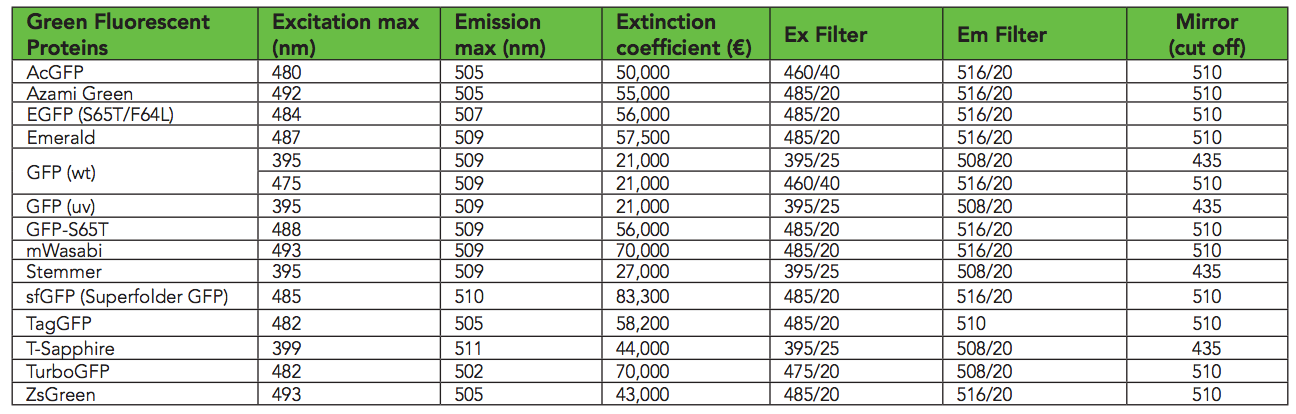 Ragam GFP ( biotek )
Ragam GFP ( biotek )IPTG dan X-gal
IPTG saya berada pada konsentrasi 1M dan harus diencerkan 1: 1000. Pada gilirannya, X-gal pada konsentrasi 20 mg / ml juga harus diencerkan 1: 1000 (20 mg / μl). Oleh karena itu, pada 2000μl LB saya tambahkan 2 μl masing-masing.Menurut satu protokol, Anda harus terlebih dahulu mengambil 40 μl X-gal pada konsentrasi 20 mg / ml dan 40 μl konsentrasi IPTG 0,1 mM (atau 4 μl IPTG per 1M), dan menginkubasi selama 30 menit. Prosedur ini tidak berhasil untuk saya, jadi saya hanya mencampur IPTG, X-gal dan sel-sel yang sesuai dan menggunakan campuran ini secara langsung.Kode """Full Gibson assembly and transformation protocol for sfGFP and pUC19 v1: Spread IPTG and X-gal onto plates, then spread cells v2: Mix IPTG, X-gal and cells; spread the mixture v3: exclude X-gal so I can do colony picking better v4: repeat v3 to try other excitation/emission wavelengths""" p = Protocol() options = { "gibson" : False,
Persediaan: IPTG / IPTG / IPTG / IPTG / IPTG / IPTG 832.0: mikroliter {}
Inventaris: sfgfp_puc19_gibson_v3_clone / sfgfp_puc19_gibson_v3_clone / sfgfp_puc19_gibson_v3_clone / sfgfp_puc19_gibson_v3_clone / sfgfp_pone}
✓ Protokol dianalisis
40 instruksi
8 kontainer
Total Biaya: $ 53,20
Waktu Kerja: $ 17,35
Reagen & Habis: $ 35,86 Koleksi koloni
Ketika koloni tumbuh di piring ampisilin, saya bisa "mengumpulkan" koloni individu dan menanamnya di piring 96-tabung. Untuk ini, ada perintah khusus ( autopick ) dalam protokol otomatis .Kode """Pick colonies from plates and grow in amp media and check for fluorescence. v2: try again with a new plate (no blue colonies) v3: repeat with different emission and excitation wavelengths""" p = Protocol() options = {} for k, v in list(options.items()): if v is False: del options[k] experiment_name = "sfgfp_puc19_gibson_pick_v3" def plate_expid(val): """refer to the previous plating experiment's outputs""" plate_exp = "sfgfp_puc19_gibson_plates_v4" return "{}_{}".format(plate_exp, val)
✓ Protokol dianalisis
62 instruksi
8 kontainer
Total Biaya: $ 66,38
Waktu Kerja: $ 57,59
Reagen & Habis: $ 8,78
Hasil: panen koloni
Layar biru-putih sempurna menunjukkan, terutama, koloni putih di piring dengan antibiotik (1-4) dan hanya biru di piring tanpa antibiotik (5-6). Ini persis seperti yang saya harapkan, dan saya senang melihatnya, terutama karena saya menggunakan IPTG dan X-gal saya sendiri, yang saya kirim ke Transcriptic.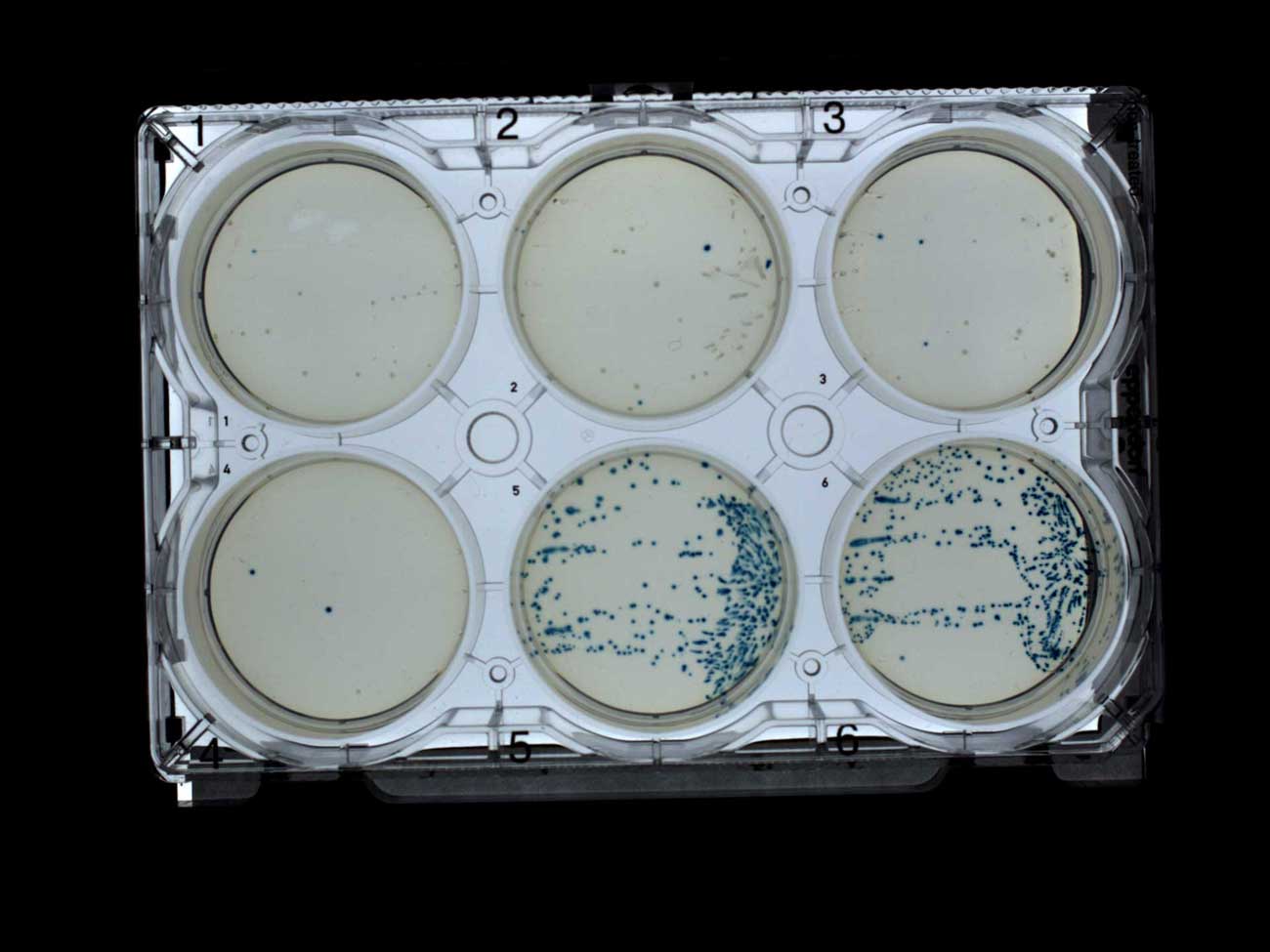 Penapisan dengan pemilihan pelat putih-biru dengan ampisilin (1-4) dan tanpa antibiotik (5-6)Namun, robot pengumpul koloni tidak bekerja dengan baik dengan koloni putih-biru ini. Gambar di bawah ini dibuat dengan mengurangi foto berturut-turut piring setelah setiap putaran pemilihan piring dan meningkatkan kontras perbedaan (dalam GraphicsMagick ). Dengan cara ini, saya dapat memvisualisasikan koloni mana yang dikumpulkan (walaupun tidak idealnya, karena koloni yang dikumpulkan tidak sepenuhnya dihapus).Saya juga menandatangani gambar dengan jumlah koloni yang dikumpulkan oleh robot Transkriptik. Diasumsikan bahwa ia akan mengumpulkan maksimal 10 koloni dari lima lempeng pertama. Namun, secara umum, beberapa koloni dikumpulkan, dan ini biasanya koloni biru. Robot hanya berhasil menemukan sepuluh koloni pada pelat kontrol dengan hanya koloni biru. Teori kerja saya adalah bahwa robot pengumpul koloni lebih disukai mengumpulkan koloni biru karena lebih kontras.
Penapisan dengan pemilihan pelat putih-biru dengan ampisilin (1-4) dan tanpa antibiotik (5-6)Namun, robot pengumpul koloni tidak bekerja dengan baik dengan koloni putih-biru ini. Gambar di bawah ini dibuat dengan mengurangi foto berturut-turut piring setelah setiap putaran pemilihan piring dan meningkatkan kontras perbedaan (dalam GraphicsMagick ). Dengan cara ini, saya dapat memvisualisasikan koloni mana yang dikumpulkan (walaupun tidak idealnya, karena koloni yang dikumpulkan tidak sepenuhnya dihapus).Saya juga menandatangani gambar dengan jumlah koloni yang dikumpulkan oleh robot Transkriptik. Diasumsikan bahwa ia akan mengumpulkan maksimal 10 koloni dari lima lempeng pertama. Namun, secara umum, beberapa koloni dikumpulkan, dan ini biasanya koloni biru. Robot hanya berhasil menemukan sepuluh koloni pada pelat kontrol dengan hanya koloni biru. Teori kerja saya adalah bahwa robot pengumpul koloni lebih disukai mengumpulkan koloni biru karena lebih kontras. Skrining piring untuk seleksi biru-putih dengan ampisilin (1-4) dan tanpa antibiotik (5-6), menunjukkan jumlah koloni yang dikumpulkanSkrining biru dan putih melayani tujuan tertentu. Dia menunjukkan bahwa sebagian besar koloni berubah dengan benar. Setidaknya ada sisipan. Namun, untuk pengumpulan koloni yang lebih baik, saya mengulangi percobaan tanpa X-gal.Hanya dengan koloni putih kolektor robot berhasil mengumpulkan sepuluh koloni dari masing-masing lima lempeng pertama. Dapat diasumsikan bahwa di sebagian besar koloni yang terkoleksi ada penyisipan yang berhasil.
Skrining piring untuk seleksi biru-putih dengan ampisilin (1-4) dan tanpa antibiotik (5-6), menunjukkan jumlah koloni yang dikumpulkanSkrining biru dan putih melayani tujuan tertentu. Dia menunjukkan bahwa sebagian besar koloni berubah dengan benar. Setidaknya ada sisipan. Namun, untuk pengumpulan koloni yang lebih baik, saya mengulangi percobaan tanpa X-gal.Hanya dengan koloni putih kolektor robot berhasil mengumpulkan sepuluh koloni dari masing-masing lima lempeng pertama. Dapat diasumsikan bahwa di sebagian besar koloni yang terkoleksi ada penyisipan yang berhasil. Koloni tumbuh di piring dengan ampisilin (1-4) dan tanpa antibiotik (5-6)
Koloni tumbuh di piring dengan ampisilin (1-4) dan tanpa antibiotik (5-6)Hasil: transformasi dengan produk rakitan
Setelah menumbuhkan 50 koloni yang dipilih di piring 96-tabung selama 20 jam, saya mengukur fluoresensi untuk memeriksa ekspresi sfGFP. Transkriptik menggunakan pembaca Tecan Infinite untuk mengukur fluoresensi dan penyerapan (dan luminesensi, jika Anda suka) .Secara teori, di koloni mana pun, sebuah plasmid harus dikumpulkan dengan pertumbuhan, karena ia membutuhkan resistensi antibiotik untuk tumbuh, dan setiap plasmid yang dikumpulkan menyatakan sfGFP. Sebenarnya ada banyak alasan mengapa hal ini mungkin tidak terjadi, paling tidak karena Anda dapat kehilangan gen sfGFP dari plasmid tanpa kehilangan resistensi ampisilin. Bakteri yang kehilangan gen sfGFP memiliki keunggulan dalam pemilihan dibandingkan pesaingnya, karena tidak menghabiskan energi ekstra, dan dengan mempertimbangkan jumlah generasi pertumbuhan yang cukup, ini pasti akan terjadi.Saya mengumpulkan data penyerapan (OD600) dan fluoresensi setiap empat jam selama 20 jam (sekitar 60 generasi). for t in [0,4,8,12,16,20]: abs_data = pd.read_csv("glow/sfgfp_puc19_gibson_pick_v3_abs_{}.csv".format(t), index_col="Well") flr_data = pd.read_csv("glow/sfgfp_puc19_gibson_pick_v3_fl2_{}.csv".format(t), index_col="Well") if t == 0: new_data = abs_data.join(flr_data) else: new_data = new_data.join(abs_data, rsuffix='_{}'.format(t)) new_data = new_data.join(flr_data, rsuffix='_{}'.format(t)) new_data.columns = ["OD 600:nanometer_0", "Fluorescence_0"] + list(new_data.columns[2:])
Kami menempatkan pada grafik data jam ke-20 dan jejak pengukuran sebelumnya. Bahkan, saya hanya tertarik pada data terbaru, karena saat itulah puncak fluoresensi harus diamati. svg = [] W, H = 800, 500 min_x, max_x = 0, 0.8 min_y, max_y = 0, 50000 def _toxy(x, y): return W*(x-min_x)/(max_x-min_x), HH*(y-min_y)/(max_y-min_y) def _topt(x, y): return ','.join(map(str,_toxy(x,y))) ab_fls = [[row[0]] + [list(row[1])] for row in new_data.iterrows()]
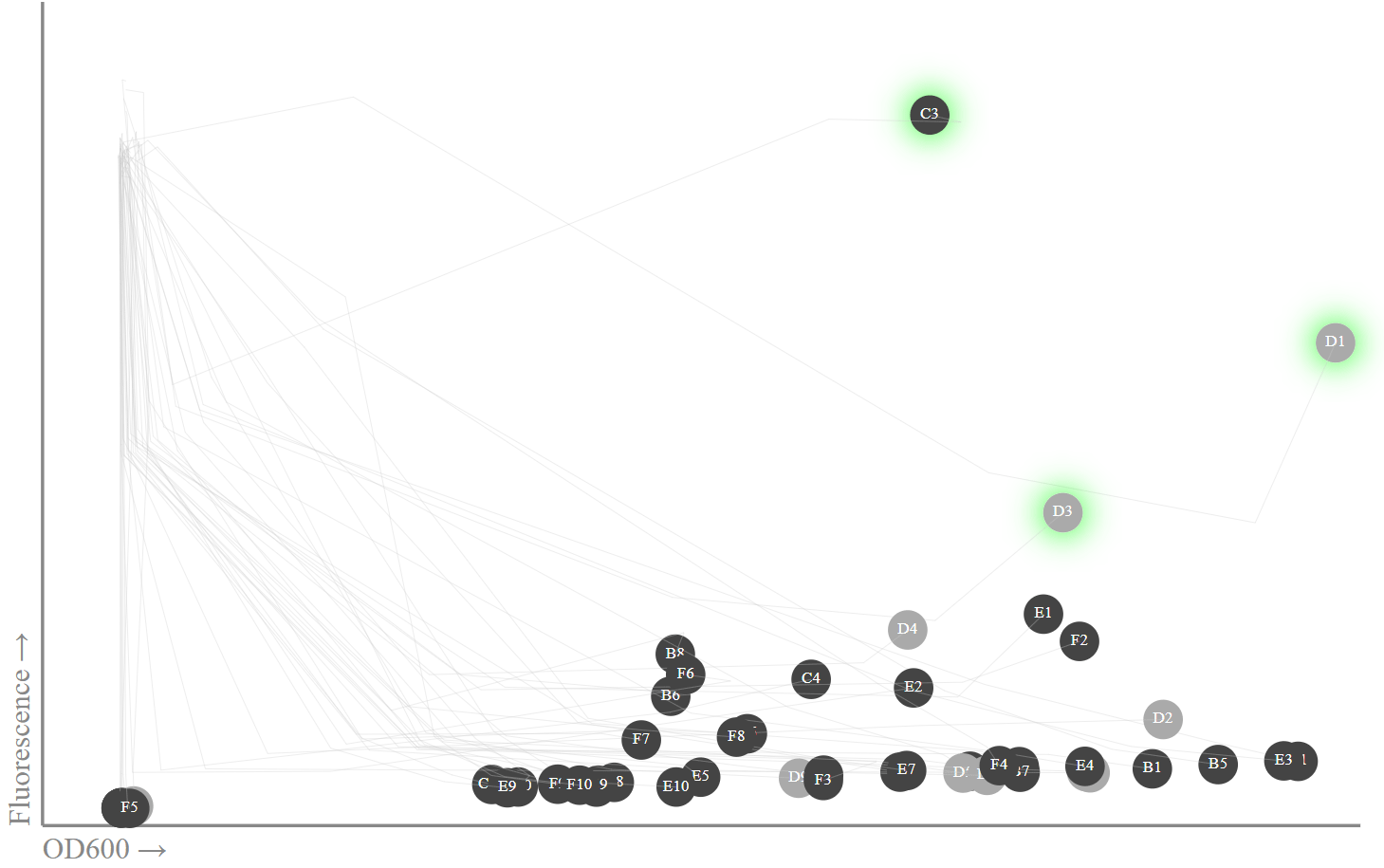 Fluoresensi dan OD600: koloni dengan ampisilin berwarna hitam, koloni kontrol tanpa ampisilin berwarna abu-abu. Koloni disorot dalam warna hijau, di mana saya mengkonfirmasi urutan protein sfGFP benar.Jalankan miniprep untuk mengekstraksi DNA plasmid, lalu sekuensing Sanger menggunakan primer M13. Sayangnya, untuk beberapa alasan, miniprep saat ini hanya tersedia melalui protokol web Transkriptik, dan bukan melalui protokol otomatis. Saya akan mengurutkan tiga tabung dengan pembacaan fluoresensi tertinggi (C1, D1, D3) dan tiga lainnya (B1, B3, E1), menyelaraskan urutan dengan sfGFP menggunakan otot .Dalam tabung C1, D3, dan D3, pasangan yang sempurna untuk urutan sfGFP asli saya, sedangkan di B1, B3, dan E1, mutasi atau penyelarasan kasar hanya gagal.
Fluoresensi dan OD600: koloni dengan ampisilin berwarna hitam, koloni kontrol tanpa ampisilin berwarna abu-abu. Koloni disorot dalam warna hijau, di mana saya mengkonfirmasi urutan protein sfGFP benar.Jalankan miniprep untuk mengekstraksi DNA plasmid, lalu sekuensing Sanger menggunakan primer M13. Sayangnya, untuk beberapa alasan, miniprep saat ini hanya tersedia melalui protokol web Transkriptik, dan bukan melalui protokol otomatis. Saya akan mengurutkan tiga tabung dengan pembacaan fluoresensi tertinggi (C1, D1, D3) dan tiga lainnya (B1, B3, E1), menyelaraskan urutan dengan sfGFP menggunakan otot .Dalam tabung C1, D3, dan D3, pasangan yang sempurna untuk urutan sfGFP asli saya, sedangkan di B1, B3, dan E1, mutasi atau penyelarasan kasar hanya gagal.Tiga koloni bercahaya
Hasilnya bagus, meskipun beberapa aspek mengejutkan. Misalnya, pembaca fluoresensi, tanpa alasan yang jelas, dimulai pada waktu 0 dengan nilai sangat tinggi (40.000 blok). Pada jam ke-20, ia telah tenang ke pola yang lebih masuk akal dengan korelasi yang jelas antara OD600 dan fluoresensi (saya kira karena sedikit tumpang tindih dalam spektrum), ditambah beberapa emisi dengan fluoresensi tinggi. Anehnya, itu bisa satu, tiga atau, mungkin, emisi 11-15.Beberapa tabung dengan nilai fluoresensi tinggi terletak di tabung kontrol (mis., Tanpa ampisilin, warnanya berwarna abu-abu), yang mengejutkan karena tidak ada tekanan seleksi dalam tabung ini, oleh karena itu, kehilangan plasmid dapat diharapkan).Berdasarkan data fluoresensi dan hasil sekuensing, tampaknya hanya tiga dari 50 koloni yang menghasilkan sfGFP dan fluoresce. Ini tidak sebanyak yang saya harapkan. Namun, karena ada tiga tahap pertumbuhan yang terpisah (di atas piring, in vitro, untuk miniprep), sekitar 200 generasi pertumbuhan telah mengalami tahap sel ini, sehingga ada cukup banyak peluang untuk terjadinya mutasi.Harus ada cara untuk membuat proses lebih efisien, terutama karena saya jauh dari ahli protokol ini. Namun, kami berhasil menghasilkan sel yang ditransformasi dengan ekspresi GFP yang direkayasa hanya menggunakan kode Python!Bagian Tiga: Kesimpulan
Harga
Bergantung pada cara mengukur, biaya percobaan ini adalah sekitar $ 360, tidak termasuk uang untuk debugging:- $ 70 untuk sintesis DNA
- $32 PCR
- $31
- $32
- $53
- $67
- $75 3 miniprep'
Saya pikir biaya dapat dikurangi menjadi $ 250-300 dengan beberapa perbaikan. Misalnya, koleksi robot 50 koloni mencurigakan mahal dan mungkin bisa ditinggalkan.Dalam pengalaman saya, harga ini tampaknya tinggi untuk sebagian (ahli biologi molekuler) dan rendah untuk yang lain (orang TI). Karena Transkriptik pada dasarnya hanya membebani reagen daftar harga, perbedaan utama dalam biaya adalah tenaga kerja. Robot sudah cukup murah dalam satu jam, dan dia tidak keberatan bangun di tengah malam untuk memotret piring. Setelah protokol disetujui, sulit untuk membayangkan bahwa bahkan seorang mahasiswa pascasarjana akan lebih murah, terutama mengingat biaya peluang.Untuk kejelasan, saya hanya berbicara tentang mengganti protokol rutin. Tentu saja, pengembangan protokol tingkat lanjut akan terus dilakukan oleh ahli biologi molekuler yang berkualitas, tetapi banyak bidang ilmu yang menarik menggunakan protokol rutin yang membosankan. Sampai baru-baru ini, banyak laboratorium memproduksi oligonukleotida mereka sendiri, tetapi sekarang hanya sedikit orang yang mengkhawatirkan hal ini: tidak ada biaya bagi siapa pun, bahkan mahasiswa pascasarjana, ketika IDT mengirimkannya kepada Anda dalam beberapa hari.Lab Robotik: Pro dan Kontra
Jelas, saya benar-benar percaya akan masa depan laboratorium robot. Ada beberapa hal yang benar-benar lucu dan berguna dalam bereksperimen dengan robot, terutama jika Anda terutama terlibat dalam komputasi dan alergi terhadap sarung tangan lateks dan tenaga kerja manual:- ! , . autoprotocol, .
- . 100 , .
- , , PCR. , , ? / ? , , , « 2-3 ». ?
- . . , .
- . .
- Ekspresivitas . Anda dapat menggunakan sintaks pemrograman untuk menyandikan langkah berulang atau logika bercabang. Misalnya, jika Anda ingin mengeluarkan 1 hingga 96 μl reagen dan (96 - x) μl air dalam plat 96-tabung, ini dapat ditulis secara singkat.
- Data yang dapat dibaca mesin . Data dengan hasil hampir selalu dikembalikan dalam format csv atau format lain yang cocok untuk pemrosesan mesin.
- Abstraksi . Idealnya, Anda dapat menjalankan seluruh protokol terlepas dari reagen yang digunakan atau gaya kloning dan mengganti sesuatu jika perlu, jika berfungsi lebih baik.
Tentu saja, ada beberapa kelemahan, terutama karena alat-alatnya baru saja mulai berkembang. Jika kita bandingkan dengan Internet, maka kita ada di wilayah 1994:- Transcriptic — . , , , . , , .
- — Transcriptic.
- , . Transcriptic ( , , ).
- Untuk banyak laboratorium, mungkin lebih mahal untuk menggunakan lab cloud daripada hanya mengambil mahasiswa pascasarjana untuk melakukan pekerjaan (biaya marjinal per jam: ~ $ 0). Itu tergantung pada apakah laboratorium membutuhkan tangan mahasiswa pascasarjana atau kecerdasannya.
- Transkriptik belum bereksperimen pada akhir pekan. Anda dapat memahaminya, tetapi ini bisa merepotkan, bahkan jika Anda memiliki proyek kecil.
Perangkat lunak untuk membuat protein
Meskipun ada banyak kode dan cukup banyak proses debug, saya pikir mungkin untuk membuat beberapa jenis perangkat lunak yang mengambil urutan protein sebagai input dan membuat bakteri dengan ekspresi protein ini pada output.Agar ini berfungsi, beberapa hal harus terjadi:- Integrasi Twist / IDT / Gen9 yang sebenarnya dengan Transcriptic (mungkin akan lambat karena permintaan saat ini rendah).
- , , , , . .
- ( NEB, IDT) (, primer3 ).
Dalam banyak aplikasi, Anda juga ingin memurnikan protein Anda (melalui kolom ), atau mungkin hanya membuat bakteri mengeluarkannya . Misalkan kita dapat segera melakukan ini di lab cloud, atau bahwa kita dapat melakukan eksperimen in vivo (mis., Di dalam sel bakteri).Ada banyak kemungkinan agar protokol benar-benar berfungsi lebih baik daripada manusia, misalnya: desain promotor dan RBS untuk mengoptimalkan ekspresi yang spesifik untuk urutan Anda; statistik probabilitas keberhasilan percobaan berdasarkan percobaan yang dapat dibandingkan; analisis gel otomatis.Kenapa semua ini?
Setelah semua ini, mungkin tidak sepenuhnya jelas mengapa membuat protein seperti itu. Berikut ini beberapa ide:- - //, .
- , , .
- in vivo split-GFP .
- scFv . scFvs - .
- BiTE , ( , ).
- Buat vaksin lokal yang masuk ke tubuh melalui folikel rambut (saya tidak menyarankan mencoba ini di rumah).
- Mutagenisasi protein Anda dengan ratusan cara berbeda dan lihat apa yang terjadi. Lalu skala hingga 1000 atau 10.000 mutasi? Mungkin ciri mutasi GFP?
Untuk ide-ide baru tentang apa yang mungkin dengan desain protein, lihat ratusan proyek iGEM .Pada akhirnya, saya ingin berterima kasih kepada Transkriptik Ben Miles atas bantuannya dalam menyelesaikan proyek ini.