
Hai, habrozhiteli! “Pemodelan prediktif dalam praktik” mencakup semua aspek peramalan, dimulai dengan tahap-tahap utama pemrosesan data, pemisahan data dan prinsip-prinsip dasar penyetelan model. Semua tahapan pemodelan dipertimbangkan pada contoh-contoh praktis dari kehidupan nyata, dalam setiap bab diberikan kode terperinci dalam R.
Buku ini dapat digunakan sebagai pengantar model prediksi dan panduan untuk aplikasi mereka. Pembaca yang tidak memiliki pelatihan matematika akan menghargai penjelasan intuitif metode tertentu, dan perhatian yang diberikan untuk memecahkan masalah aktual dengan data nyata akan membantu spesialis yang ingin meningkatkan keterampilan mereka.
Para penulis mencoba menghindari formula yang rumit, memahami konsep statistik dasar, seperti korelasi dan analisis regresi linier, sudah cukup untuk menguasai materi dasar, tetapi pelatihan matematika diperlukan untuk mempelajari topik-topik lanjutan.
Kutipan. 7.5. Perhitungan
Bagian ini akan menggunakan fungsi dari paket R caret, earth, kernlab, dan nnet.
R memiliki banyak paket dan fungsi untuk membuat jaringan saraf. Ini termasuk paket nnet, neural, dan RSNNS. Fokus utama adalah pada paket nnet, yang mendukung model dasar jaringan saraf dengan satu tingkat variabel tersembunyi, pengurangan berat badan, dan ditandai dengan sintaksis yang relatif sederhana. RSNNS mendukung berbagai jaringan saraf. Perhatikan bahwa Bergmeir dan Benitez (2012) memiliki deskripsi singkat tentang berbagai paket jaringan saraf di R. Ini juga menyediakan tutorial tentang RSNNS.
Jaringan saraf
Untuk memperkirakan model regresi, fungsi nnet dapat menerima formula model dan antarmuka matriks. Untuk regresi, hubungan linear antara variabel tersembunyi dan prakiraan digunakan dengan parameter linout = TRUE. Panggilan paling sederhana ke fungsi jaringan saraf akan terlihat seperti:
> nnetFit <- nnet(predictors, outcome, + size = 5, + decay = 0.01, + linout = TRUE, + ## + trace = FALSE, + ## + ## .. + maxit = 500, + ## , + MaxNWts = 5 * (ncol(predictors) + 1) + 5 + 1)
Panggilan ini menciptakan satu model dengan lima variabel tersembunyi. Diasumsikan bahwa data dalam prediktor distandarisasi pada skala tunggal.
Untuk rata-rata model, fungsi avNNet dari paket caret digunakan, yang memiliki sintaks yang sama:
> nnetAvg <- avNNet(predictors, outcome, + size = 5, + decay = 0.01, + ## + repeats = 5, + linout = TRUE, + ## + trace = FALSE, + ## + ## .. + maxit = 500, + ## , + MaxNWts = 5 * (ncol(predictors) + 1) + 5 + 1)
Poin data baru diproses oleh perintah
> predict(nnetFit, newData) > ## > predict(nnetAvg, newData)
Untuk mereproduksi metode yang disajikan sebelumnya memilih jumlah variabel tersembunyi dan jumlah pengurangan berat badan dengan pengambilan sampel berulang, kami menerapkan fungsi kereta dengan metode = "nnet" atau metode = "avNNet" parameter, pertama menghapus prediktor (sehingga korelasi pasangan absolut maksimum antara prediktor tidak melebihi 0,75):
> ## findCorrelation > ## , > ## > tooHigh <- findCorrelation(cor(solTrainXtrans), cutoff = .75) > trainXnnet <- solTrainXtrans[, -tooHigh] > testXnnet <- solTestXtrans[, -tooHigh] > ## - : > nnetGrid <- expand.grid(.decay = c(0, 0.01, .1), + .size = c(1:10), + ## — + ## (. ) + ## . + .bag = FALSE) > set.seed(100) > nnetTune <- train(solTrainXtrans, solTrainY, + method = "avNNet", + tuneGrid = nnetGrid, + trControl = ctrl, + ## + ## + preProc = c("center", "scale"), + linout = TRUE, + trace = FALSE, + MaxNWts = 10 * (ncol(trainXnnet) + 1) + 10 + 1, + maxit = 500)
Regresi adaptif multidimensi splines
Model MARS terkandung dalam beberapa paket, tetapi implementasi yang paling luas ada di paket earth. Model MARS menggunakan fase nominal direct pass dan pemotongan dapat dipanggil sebagai berikut:
> marsFit <- earth(solTrainXtrans, solTrainY) > marsFit Selected 38 of 47 terms, and 30 of 228 predictors Importance: NumNonHAtoms, MolWeight, SurfaceArea2, SurfaceArea1, FP142, ... Number of terms at each degree of interaction: 1 37 (additive model) GCV 0.3877448 RSS 312.877 GRSq 0.907529 RSq 0.9213739
Karena model ini dalam implementasi internal menggunakan metode GCV untuk memilih model, strukturnya agak berbeda dari model yang dijelaskan sebelumnya dalam bab ini. Metode ringkasan menghasilkan keluaran yang lebih luas:
> summary(marsFit) Call: earth(x=solTrainXtrans, y=solTrainY) coefficients (Intercept) -3.223749 FP002 0.517848 FP003 -0.228759 FP059 -0.582140 FP065 -0.273844 FP075 0.285520 FP083 -0.629746 FP085 -0.235622 FP099 0.325018 FP111 -0.403920 FP135 0.394901 FP142 0.407264 FP154 -0.620757 FP172 -0.514016 FP176 0.308482 FP188 0.425123 FP202 0.302688 FP204 -0.311739 FP207 0.457080 h(MolWeight-5.77508) -1.801853 h(5.94516-MolWeight) 0.813322 h(NumNonHAtoms-2.99573) -3.247622 h(2.99573-NumNonHAtoms) 2.520305 h(2.57858-NumNonHBonds) -0.564690 h(NumMultBonds-1.85275) -0.370480 h(NumRotBonds-2.19722) -2.753687 h(2.19722-NumRotBonds) 0.123978 h(NumAromaticBonds-2.48491) -1.453716 h(NumNitrogen-0.584815) 8.239716 h(0.584815-NumNitrogen) -1.542868 h(NumOxygen-1.38629) 3.304643 h(1.38629-NumOxygen) -0.620413 h(NumChlorine-0.46875) -50.431489 h(HydrophilicFactor- -0.816625) 0.237565 h(-0.816625-HydrophilicFactor) -0.370998 h(SurfaceArea1-1.9554) 0.149166 h(SurfaceArea2-4.66178) -0.169960 h(4.66178-SurfaceArea2) -0.157970 Selected 38 of 47 terms, and 30 of 228 predictors Importance: NumNonHAtoms, MolWeight, SurfaceArea2, SurfaceArea1, FP142, ... Number of terms at each degree of interaction: 1 37 (additive model) GCV 0.3877448 RSS 312.877 GRSq 0.907529 RSq 0.9213739
Dalam derivasi ini, h (·) adalah fungsi engsel. Dalam hasil yang disajikan, komponen h (MolWeight-5.77508) sama dengan nol untuk berat molekul kurang dari 5.77508 (seperti di bagian atas Gambar 7.3). Fungsi engsel yang dipantulkan memiliki bentuk h (5.77508 - MolWeight).
Fungsi plotmo dari paket bumi dapat digunakan untuk membuat diagram yang mirip dengan yang ditunjukkan pada gambar. 7.5. Anda dapat menggunakan kereta untuk mengkonfigurasi model menggunakan resampling eksternal. Kode berikut mereproduksi hasil yang ditunjukkan pada gambar. 7.4:
> # - > marsGrid <- expand.grid(.degree = 1:2, .nprune = 2:38) > # > set.seed(100) > marsTuned <- train(solTrainXtrans, solTrainY, + method = "earth", + # - + tuneGrid = marsGrid, + trControl = trainControl(method = "cv")) > marsTuned 951 samples 228 predictors No pre-processing Resampling: Cross-Validation (10-fold) Summary of sample sizes: 856, 857, 855, 856, 856, 855, ... Resampling results across tuning parameters: degree nprune RMSE Rsquared RMSE SD Rsquared SD 1 2 1.54 0.438 0.128 0.0802 1 3 1.12 0.7 0.0968 0.0647 1 4 1.06 0.73 0.0849 0.0594 1 5 1.02 0.75 0.102 0.0551 1 6 0.984 0.768 0.0733 0.042 1 7 0.919 0.796 0.0657 0.0432 1 8 0.862 0.821 0.0418 0.0237 : : : : : : 2 33 0.701 0.883 0.068 0.0307 2 34 0.702 0.883 0.0699 0.0307 2 35 0.696 0.885 0.0746 0.0315 2 36 0.687 0.887 0.0604 0.0281 2 37 0.696 0.885 0.0689 0.0291 2 38 0.686 0.887 0.0626 0.029 RMSE was used to select the optimal model using the smallest value. The final values used for the model were degree = 1 and nprune = 38. > head(predict(marsTuned, solTestXtrans)) [1] 0.3677522 -0.1503220 -0.5051844 0.5398116 -0.4792718 0.7377222
Dua fungsi digunakan untuk mengevaluasi pentingnya setiap prediktor dalam model MARS: evimp dari paket bumi dan varImp dari paket caret (yang kedua memanggil yang pertama):
> varImp(marsTuned) earth variable importance only 20 most important variables shown (out of 228) Overall MolWeight 100.00 NumNonHAtoms 89.96 SurfaceArea2 89.51 SurfaceArea1 57.34 FP142 44.31 FP002 39.23 NumMultBond s 39.23 FP204 37.10 FP172 34.96 NumOxygen 30.70 NumNitrogen 29.12 FP083 28.21 NumNonHBonds 26.58 FP059 24.76 FP135 23.51 FP154 21.20 FP207 19.05 FP202 17.92 NumRotBonds 16.94 FP085 16.02
Hasil ini diskalakan dalam kisaran dari 0 hingga 100, berbeda dari yang diberikan dalam tabel. 7.1 (model yang disajikan pada Tabel 7.1 tidak melalui proses pertumbuhan dan pemotongan yang lengkap). Perhatikan bahwa variabel mengikuti beberapa dari mereka kurang signifikan untuk model.
SVM, mendukung metode vektor
Implementasi model SVM terkandung dalam beberapa paket R. Chang dan Lin (Chang dan Lin, 2011) menggunakan fungsi svm dari paket e1071 untuk menggunakan antarmuka ke perpustakaan LIBSVM untuk regresi. Implementasi yang lebih lengkap dari model SVM untuk regresi Karatsogl (Karatzoglou et al., 2004) terkandung dalam paket kernlab, yang mencakup fungsi ksvm untuk model regresi dan sejumlah besar fungsi nuklir. Secara default, fungsi dasar radial digunakan. Jika nilai parameter biaya dan nuklir diketahui, maka perkiraan model dapat dilakukan sebagai berikut:
> svmFit <- ksvm(x = solTrainXtrans, y = solTrainY, + kernel ="rbfdot", kpar = "automatic", + C = 1, epsilon = 0.1)
Untuk memperkirakan σ, analisis otomatis digunakan. Karena y adalah vektor numerik, fungsi jelas mendekati model regresi (bukan model klasifikasi). Anda dapat menggunakan fungsi kernel lainnya, termasuk polinomial (kernel = "polydot") dan linear (kernel = "vanilladot").
Jika nilainya tidak diketahui, maka nilainya dapat diperkirakan dengan pengambilan sampel ulang. Dalam kereta, nilai "svmRadial", "svmLinear" atau "svmPoly" dari parameter metode pilih fungsi kernel yang berbeda:
> svmRTuned <- train(solTrainXtrans, solTrainY, + method = "svmRadial", + preProc = c("center", "scale"), + tuneLength = 14, + trControl = trainControl(method = "cv"))
Argumen tuneLength menggunakan 14 nilai default.

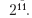
Estimasi default σ dilakukan melalui analisis otomatis.
> svmRTuned 951 samples 228 predictors Pre-processing: centered, scaled Resampling: Cross-Validation (10-fold) Summary of sample sizes: 855, 858, 856, 855, 855, 856, ... Resampling results across tuning parameters: C RMSE Rsquared RMSE SD Rsquared SD 0.25 0.793 0.87 0.105 0.0396 0.5 0.708 0.889 0.0936 0.0345 1 0.664 0.898 0.0834 0.0306 2 0.642 0.903 0.0725 0.0277 4 0.629 0.906 0.067 0.0253 8 0.621 0.908 0.0634 0.0238 16 0.617 0.909 0.0602 0.0232 32 0.613 0.91 0.06 0.0234 64 0.611 0.911 0.0586 0.0231 128 0.609 0.911 0.0561 0.0223 256 0.609 0.911 0.056 0.0224 512 0.61 0.911 0.0563 0.0226 1020 0.613 0.91 0.0563 0.023 2050 0.618 0.909 0.0541 0.023 Tuning parameter 'sigma' was held constant at a value of 0.00387 RMSE was used to select the optimal model using the smallest value. The final values used for the model were C = 256 and sigma = 0.00387.
Subobject finalModel berisi model yang dibuat oleh fungsi ksvm:
> svmRTuned$finalModel Support Vector Machine object of class "ksvm" SV type: eps-svr (regression) parameter : epsilon = 0.1 cost C = 256 Gaussian Radial Basis kernel function. Hyperparameter : sigma = 0.00387037424967707 Number of Support Vectors : 625 Objective Function Value : -1020.558 Training error : 0.009163
Sebagai vektor referensi, model ini menggunakan 625 titik data dari set pelatihan (mis. 66% dari set pelatihan).
Paket kernlab berisi implementasi model RVM untuk regresi dalam fungsi rvm. Sintaksnya sangat mirip dengan yang disajikan dalam contoh untuk ksvm.
Metode KNN
Paket caren knnreg mendekati model regresi KNN; fungsi kereta menetapkan model ke K:
> # . > knnDescr <- solTrainXtrans[, -nearZeroVar(solTrainXtrans)] > set.seed(100) > knnTune <- train(knnDescr, + solTrainY, + method = "knn", + # + # + preProc = c("center", "scale"), + tuneGrid = data.frame(.k = 1:20), + trControl = trainControl(method = "cv"))
Tentang penulis:
Max Kun adalah Kepala Departemen Penelitian dan Pengembangan Statistik Non-Klinis Pfizer Global. Dia telah bekerja dengan model prediktif selama lebih dari 15 tahun dan merupakan penulis beberapa paket khusus untuk bahasa R. Pemodelan prediktif dalam praktik mencakup semua aspek peramalan, dimulai dengan tahapan kunci preprocessing data, pemisahan data, dan prinsip-prinsip dasar
pengaturan model. Semua tahapan pemodelan dipertimbangkan pada contoh praktis.
dari kehidupan nyata, setiap bab memberikan kode terperinci dalam R.
Kjell Johnson telah bekerja di bidang statistik dan pemodelan prediktif untuk penelitian farmasi selama lebih dari sepuluh tahun. Salah satu pendiri Arbor Analytics, sebuah perusahaan yang berspesialisasi dalam pemodelan prediktif; sebelumnya mengepalai departemen penelitian dan pengembangan statistik di Pfizer Global. Karya ilmiahnya dikhususkan untuk aplikasi dan pengembangan metodologi statistik dan algoritma pembelajaran.
»Informasi lebih lanjut tentang buku ini dapat ditemukan di
situs web penerbit»
Isi»
KutipanKupon diskon 25% untuk pedagang asongan -
Applied Predictive ModellingSetelah pembayaran versi kertas buku, sebuah buku elektronik dikirim melalui email.