Montagem de circuitos a partir de interruptores biológicos
← Artigo anterior do cicloA lógica de incluir operons de diferentes vias metabólicas em E. coli está próxima dos elementos AND e OR. Portanto, com base neles, você pode criar uma construção de gene que implemente um esquema lógico arbitrário (não muito complicado). Existem programas, por exemplo, o Violoncelo, que, a partir da descrição da lógica em Verilog, faz uma sequência de uma construção de gene que implementa essa lógica.O problema inverso acaba sendo mais difícil: obter uma descrição da lógica de sua regulação a partir da sequência do DNA natural. Vamos começar com casos simples em que não há loops de feedback complexos e a integração de muitos sinais em um operon. Quase toda a regulação dos genes de E. coli satisfaz essas condições. Se tomarmos todas as conexões regulatórias conhecidas entre os genes de Escherichia coli e construirmos uma rede deles, poderemos calcular as estatísticas dos “motivos” dessa rede (métodos de conexão de elementos) e selecionar os mais usados.Dinâmica de comutação genética
No último artigo, modelamos a operação do operon lactose com um modelo simples com quatro parâmetros. O tempo não fazia parte desse modelo. Portanto, de fato, descreve apenas o estado de equilíbrio, quando os níveis do sinal de entrada permanecem constantes por um longo tempo. Para modelar processos rápidos ao ativar e desativar genes, fatores adicionais devem ser considerados.Em primeiro lugar, há um atraso na transcrição: do início do trabalho da RNA polimerase até o final da síntese do mRNA e, em seguida, das proteínas, passa um tempo notável, cerca de 10 minutos em Escherichia coli e de uma hora ou mais em humanos. Portanto, a taxa de aparecimento de novas moléculas de proteína não depende agora do estado do promotor de seu gene, mas do estado do promotor há alguns minutos atrás.Em segundo lugar, cada proteína não é apenas sintetizada, mas também decomposta. No caso mais simples, a degradação da proteína não é controlada e a cada minuto uma fração fixa das moléculas dessa proteína se decompõe na célula. Quanto mais eles são, mais eles se separam. Ou seja, a proteína tem meia-vida, durante a qual sua concentração cai pela metade.Mais detalhes para os mais curiosos— - . , — .
Para descrever a dinâmica de ativar e desativar o gene, precisamos escrever um diffur:
onde A é a atividade do gene (a mesma função de entrada do artigo anterior), tau é o tempo de atraso da transcrição ek é a taxa de quebra de proteínas. Se A é primeiro igual a zero e depois aumenta abruptamente e se mantém em um nível constante, as alterações na quantidade de proteína ao longo do tempo terão a seguinte aparência:Motivos nas redes de genes e suas funções
A característica mais simples do motivo de Escherichia coli - o loop de feedback - consiste em um gene que se regula. Na maioria das vezes, o gene é um repressor para si mesmo, ou seja, o feedback no loop é negativo. Se essa é a principal entrada regulatória, esse feedback geralmente serve para estabilizar o nível da proteína codificada pelo gene. Esse ciclo de feedback, por exemplo, mantém um nível constante de repressor de lactose na célula. Se, além do feedback negativo, houver outras entradas regulatórias, o feedback acelera a resposta do gene a um sinal externo.(linhas tracejadas são curvas teóricas, linhas sólidas são o resultado do experimento. Linhas verdes com feedback negativo, linhas azuis sem ele)O feedback positivo (uma proteína ativa seu próprio gene) geralmente leva a desencadear comportamentos. Esse gene tem dois estados estáveis: ou ele não funciona, ou funciona em sua capacidade total. Um sinal externo forte, mas curto, pode transferir um gene de um estado estável para outro no qual permanece até o próximo sinal. Tal motivo pode servir como um elemento de memória. Se um gene "capta" ruído em uma entrada reguladora, um feedback positivo levará a uma forte disseminação na atividade gênica entre células vizinhas, crescendo nas mesmas condições. Isso é usado para manter a heterogeneidade de bactérias que crescem juntas, para que o estresse repentino não mate todos de uma vez.Por exemplo, muitos antibióticos interferem na síntese de proteínas. Se a célula não crescer e não sintetizar proteínas, ela poderá sobreviver facilmente à adição de antibióticos ao meio e retornar ao crescimento mais tarde. Em E. coli, mesmo em condições ideais, até 0,01% das células estão em tal hibernação em caso de envenenamento repentino do ambiente. Essas células dormentes (chamadas de "persistentes") não diferem das demais no DNA, pois entram e saem da hibernação sob o controle de um gene com feedback positivo e entrada de ruído.Outro motivo comum das redes de genes é um pouco mais complicado. Ele é chamado de "loop feed-forward" e consiste em três genes: o gene X regula os genes Y e Z, o gene Y regula Z. Cada um dos três elos do loop pode ser ativação ou repressão, portanto, em princípio, são possíveis 8 tipos tais laços. Na realidade, geralmente são encontrados dois tipos de oito: os genes X e Y são ativadores (loop coerente tipo 1) ou X é o ativador Y e Z, Y é o repressor Z (loop incoerente tipo 1).O comportamento do loop com dois ativadores depende da lógica de interação das entradas em Z. Se o gene de saída funciona como AND no loop, então o loop funciona como um filtro passa-baixo que não passa pulsos curtos para a saída. Para que o sinal de saída apareça nesse loop, a entrada (X) deve receber um sinal por tempo suficiente para a proteína X se acumular e, em seguida, a proteína Y. Somente a ação combinada das proteínas X e Y ativará o gene Z.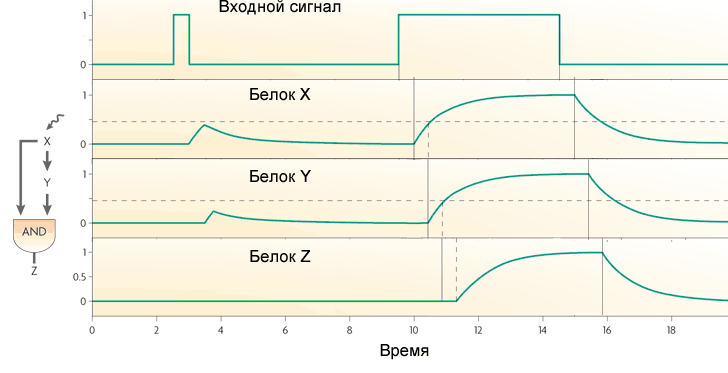 Se o gene Z funcionar como OR, a função do loop torna-se o oposto: passa todos os pulsos para a saída e se transforma em curto em longos. Mas ela não perde pausas curtas entre pulsos.O loop direto com o ativador X e o repressor Y atua como um filtro passa-alto. Produz pulsos de pequeno comprimento fixo (igual ao atraso transcricional do gene Y) na saída em resposta às bordas principais dos sinais de entrada.O terceiro motivo frequente nas redes de genes de Escherichia coli é um módulo de entrada única. É simplesmente um grupo de genes dirigidos pelo mesmo gene regulador. Normalmente, os genes desse grupo têm limiares de ativação diferentes, portanto, um aumento suave na concentração do fator de transcrição inclui genes em uma determinada ordem. Esse esquema é usado, por exemplo, no gerenciamento de respostas ao estresse.
Se o gene Z funcionar como OR, a função do loop torna-se o oposto: passa todos os pulsos para a saída e se transforma em curto em longos. Mas ela não perde pausas curtas entre pulsos.O loop direto com o ativador X e o repressor Y atua como um filtro passa-alto. Produz pulsos de pequeno comprimento fixo (igual ao atraso transcricional do gene Y) na saída em resposta às bordas principais dos sinais de entrada.O terceiro motivo frequente nas redes de genes de Escherichia coli é um módulo de entrada única. É simplesmente um grupo de genes dirigidos pelo mesmo gene regulador. Normalmente, os genes desse grupo têm limiares de ativação diferentes, portanto, um aumento suave na concentração do fator de transcrição inclui genes em uma determinada ordem. Esse esquema é usado, por exemplo, no gerenciamento de respostas ao estresse.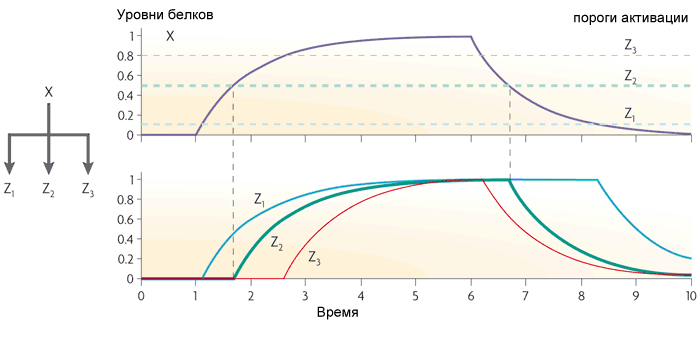 O quarto motivo mais complexo é o módulo de múltiplas entradas, ou o regulon denso e sobreposto. Consiste em vários genes reguladores que controlam vários genes de "saída".Dependendo da lógica de regulação de cada gene de saída, esse motivo pode funcionar de maneira muito diferente. Pode ser comparado com um conjunto de elementos lógicos que, dependendo das conexões, podem ser um decodificador, contador ou qualquer outra coisa. Ainda não existem descrições completas da lógica do trabalho de tais motivos.Com esses quatro motivos, a estrutura das redes de genes de Escherichia coli ou levedura está praticamente esgotada. Em sua base, vários esquemas artificiais já estão sendo feitos. Por exemplo, bactérias com um gerador de oscilação, que também sincroniza nas células vizinhas, e a saída de proteínas luminescentes- Um análogo completo de "piscar uma lâmpada" para microcontroladores e "Hello World" para outros programadores. Ao adicionar um "sentimento de quorum" a esse sistema (quando as bactérias percebem quantos primos os cercam) e mudar a saída de luminescência para toxinas, os cientistas coletaram bactérias que se arrastam para tumores cancerígenos e se acumulam em grandes quantidades, morrem juntas, liberando toxinas e matando tumor ( mais detalhes aqui ).Mas estas são bactérias e leveduras. As redes de genes animais são muito mais complexas. Na próxima parte, analisaremos uma das redes de genes animais bem estudadas e os problemas que surgem com seu estudo e modelagem.
O quarto motivo mais complexo é o módulo de múltiplas entradas, ou o regulon denso e sobreposto. Consiste em vários genes reguladores que controlam vários genes de "saída".Dependendo da lógica de regulação de cada gene de saída, esse motivo pode funcionar de maneira muito diferente. Pode ser comparado com um conjunto de elementos lógicos que, dependendo das conexões, podem ser um decodificador, contador ou qualquer outra coisa. Ainda não existem descrições completas da lógica do trabalho de tais motivos.Com esses quatro motivos, a estrutura das redes de genes de Escherichia coli ou levedura está praticamente esgotada. Em sua base, vários esquemas artificiais já estão sendo feitos. Por exemplo, bactérias com um gerador de oscilação, que também sincroniza nas células vizinhas, e a saída de proteínas luminescentes- Um análogo completo de "piscar uma lâmpada" para microcontroladores e "Hello World" para outros programadores. Ao adicionar um "sentimento de quorum" a esse sistema (quando as bactérias percebem quantos primos os cercam) e mudar a saída de luminescência para toxinas, os cientistas coletaram bactérias que se arrastam para tumores cancerígenos e se acumulam em grandes quantidades, morrem juntas, liberando toxinas e matando tumor ( mais detalhes aqui ).Mas estas são bactérias e leveduras. As redes de genes animais são muito mais complexas. Na próxima parte, analisaremos uma das redes de genes animais bem estudadas e os problemas que surgem com seu estudo e modelagem. Source: https://habr.com/ru/post/pt400899/
All Articles