
Apesar da existência de obstáculos óbvios e dificuldades que às vezes atrapalham o desenvolvimento e a implementação de produtos de engenharia genética (IG), o século XXI não pode ser imaginado sem os frutos dessa importante e diversificada tecnologia no arsenal de um biólogo moderno. O organismo mais comumente usado no GI é a bactéria.
O que é IG e por que precisamos? Por que as bactérias são tão populares entre os engenheiros genéticos? Qual é a maneira mais fácil de introduzir o gene desejado na bactéria? Que dificuldades podem ser encontradas ao trabalhar com esses organismos? O que aconteceu antes: a criação da primeira bactéria geneticamente modificada ou a descoberta da estrutura do DNA e do genoma? Leia sobre isso e muito mais sobre o gato.
0. Breve programa educacional em biologia
Este parágrafo fornece uma breve descrição do chamado
Dogma Central da biologia molecular . Se você possui conhecimentos básicos de biologia molecular, sinta-se à vontade para pular para a etapa 1.
 O dogma central da biologia molecular em uma imagem
O dogma central da biologia molecular em uma imagemEntão, vamos começar. Todas as informações sobre todos os estágios de desenvolvimento e as propriedades de qualquer organismo, sejam
procariontes (bactérias),
archaea ou
eucariotos (o restante é único e multicelular), são codificadas no DNA genômico, que é um complexo de duas cadeias polinucleotídicas complementares entre si, formando uma dupla hélice ( nucleotídeos de DNA complementares: AT e GC). Os cromossomos eucarióticos são moléculas lineares de DNA de fita dupla e os cromossomos procarióticos são em loop. Muitas vezes, os genes representam apenas uma pequena parte de todo o genoma (em humanos - cerca de 1,5%).
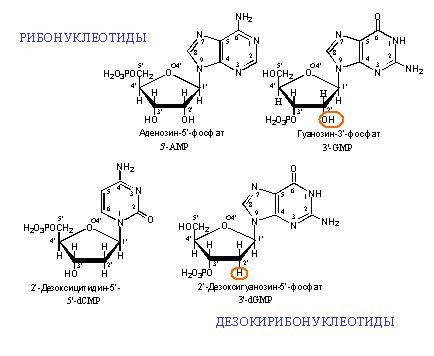 Exemplos de monômeros de DNA e RNA. "Desoxi" no nome do DNA significa a ausência de um átomo de oxigênio na posição 2 '(na figura, a posição 2' é circulada em vermelho).
Exemplos de monômeros de DNA e RNA. "Desoxi" no nome do DNA significa a ausência de um átomo de oxigênio na posição 2 '(na figura, a posição 2' é circulada em vermelho).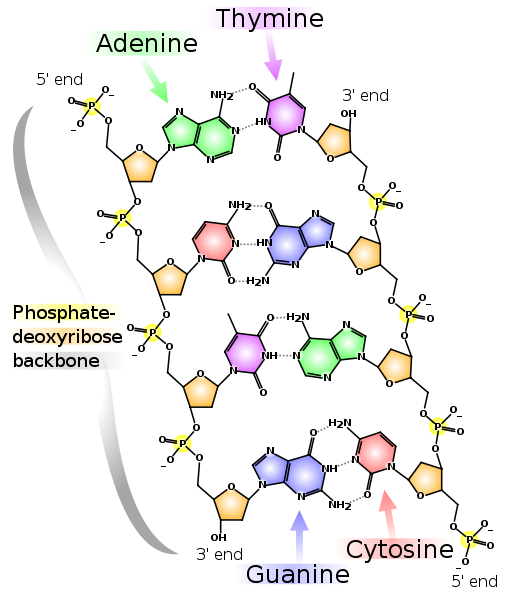 Duas cadeias complementares de DNA. As linhas tracejadas mostram as ligações de hidrogênio entre as bases. Como pode ser visto, a adenina e a timina formam duas ligações de hidrogênio entre si e a guanina e a citosina formam três. Portanto, a ligação GC é mais forte e as seções ricas em GC do DNA de fita dupla são mais difíceis de separar em duas cadeias.
Duas cadeias complementares de DNA. As linhas tracejadas mostram as ligações de hidrogênio entre as bases. Como pode ser visto, a adenina e a timina formam duas ligações de hidrogênio entre si e a guanina e a citosina formam três. Portanto, a ligação GC é mais forte e as seções ricas em GC do DNA de fita dupla são mais difíceis de separar em duas cadeias.Observe que cada uma das cadeias tem uma extremidade 5 'e uma extremidade 3'. Pode-se ver que perto da extremidade 5 'da cadeia esquerda está a extremidade 3'da direita e vice-versa, portanto, as cadeias são chamadas de "antiparalelas". O RNA também possui uma extremidade 5 'e 3'. As posições 5 'e 3' foram escolhidas para indicar o começo e o fim, porque é através delas que ligações covalentes são formadas nas cadeias de DNA e RNA.
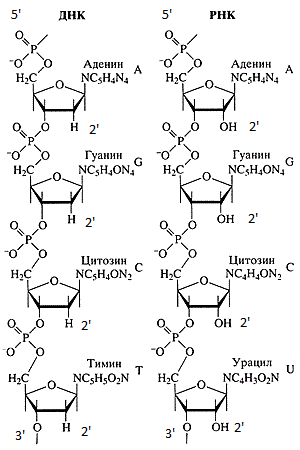 Cadeias de DNA e RNA.
Cadeias de DNA e RNA.As sequências de DNA e RNA são sempre registradas do extremo 5 'ao extremo 3'. Existem várias razões para isso:
- A síntese de novas cadeias de DNA e RNA começa na extremidade 5 '( DNA polimerase (enzimas que sintetizam uma cadeia de DNA complementar em uma matriz de DNA ou RNA) e RNA polimerase (enzimas que sintetizam uma cadeia de RNA complementar em uma matriz de DNA ou RNA) direção 3 '-> 5', então uma nova cadeia é sintetizada na direção 5 '-> 3');
- O ribossomo lê os códons, movendo-se ao longo do mRNA na direção de 5 '-> 3';
- A sequência de aminoácidos é escrita na cadeia de codificação do DNA na direção 5 '-> 3' (uma parte significativa do mRNA é uma cópia exata da região de codificação do DNA com a timina substituída pelo uracil e com um grupo hidroxila (-OH) em vez de hidrogênio na posição 2 ', é claro);
- Finalmente, é simplesmente conveniente ter uma regra de gravação geralmente aceita.
Um gene é uma porção do DNA genômico que define a sequência de nucleotídeos de uma molécula de RNA:
- RNA codificador: RNA mensageiro (mRNA), no qual a sequência de aminoácidos da proteína correspondente é codificada como códons. Você também pode encontrar o nome "RNA informativo" e a abreviação se parece com "mRNA";
- RNA não codificante: RNA de transporte, RNA ribossômico e outros.
O papel do RNAt é fornecer aminoácidos ao complexo RNAm-ribossomo. Além disso, é o tRNA responsável pelo reconhecimento dos códons de mRNA; para isso, cada tRNA inclui o chamado "anticódon" - um tripleto complementar ao códon de mRNA.
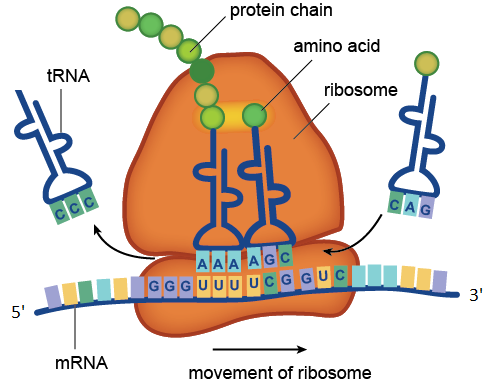 O processo de tradução catalisado pelo ribossomo. Na figura, os codões UUU e UCG contidos no mRNA são reconhecidos pelos anticódons AAA e AGC contidos nas moléculas de tRNA. O RNA de transporte com anticódon CCC já deu seu aminoácido à cadeia proteica crescente, e o tRNA com anticodonte CAG está esperando na fila. O gráfico da molécula de mRNA mostrado na figura consiste em quatro códons: GGGUUUUCGGUC. O codão GGG corresponde ao aminoácido glicina, UUU para fenilalanina, UCG para serina, GUC para valina. Portanto, esta região de mRNA codifica um fragmento de proteína com a sequência de aminoácidos glicina-fenilalanina-serina-valina.
O processo de tradução catalisado pelo ribossomo. Na figura, os codões UUU e UCG contidos no mRNA são reconhecidos pelos anticódons AAA e AGC contidos nas moléculas de tRNA. O RNA de transporte com anticódon CCC já deu seu aminoácido à cadeia proteica crescente, e o tRNA com anticodonte CAG está esperando na fila. O gráfico da molécula de mRNA mostrado na figura consiste em quatro códons: GGGUUUUCGGUC. O codão GGG corresponde ao aminoácido glicina, UUU para fenilalanina, UCG para serina, GUC para valina. Portanto, esta região de mRNA codifica um fragmento de proteína com a sequência de aminoácidos glicina-fenilalanina-serina-valina.Os RNAs ribossômicos são componentes indispensáveis do ribossomo. A principal função do rRNA é garantir o processo de tradução: ele está envolvido na leitura de informações do mRNA usando moléculas adaptadoras do tRNA e catalisa a formação de ligações peptídicas entre os aminoácidos ligados ao tRNA e a crescente cadeia de proteínas.
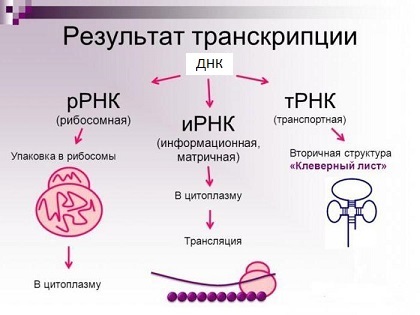 Os principais tipos de moléculas de RNA (de fato, existem muito mais).
Os principais tipos de moléculas de RNA (de fato, existem muito mais).Uma proteína, por outro lado, é uma cadeia de aminoácidos ligados covalentemente por meio de uma ligação peptídica (você pode ver um pouco mais a aparência do spoiler). Após a síntese, a cadeia de aminoácidos deve assumir uma certa estrutura espacial - “
conformação ” (
eles já me falaram sobre a estrutura espacial das proteínas
no Geektimes ). Além disso, muitas proteínas grandes na verdade consistem em várias proteínas combinadas por interações hidrofóbicas e ligações de hidrogênio em uma única estrutura estável. Nesse caso, cada uma das "proteínas de construção" é chamada de "subunidade" e a proteína grande resultante é chamada de "multisubunidade".
20 aminoácidos que compõem as proteínas  Complexo ribossômico. A foto foi tirada da publicação OlegKovalevskiy "Impressão 3D de modelos de moléculas de proteína" .
Complexo ribossômico. A foto foi tirada da publicação OlegKovalevskiy "Impressão 3D de modelos de moléculas de proteína" .No caso de genes que codificam uma proteína, o processo de decodificação de informações genéticas se parece com o seguinte:
- A RNA polimerase reconhece o promotor e se liga a ele (se estiver "aberto", discutiremos mais a regulamentação da atividade do promotor);
- Na matriz de DNA, a enzima RNA polimerase, de acordo com o princípio da complementaridade, sintetiza o "espaço em branco" do RNA da matriz (pré-mRNA, em eucariotos) ou o mRNA funcional final (em procariontes). Esse processo é chamado de "transcrição" ;
- (somente em eucariotos) A molécula pré-mRNA sofre modificações (“amadurece”) e se torna mRNA funcional;
- O mRNA é reconhecido pelo ribossomo , uma enzima que decodifica o código tripleto do mRNA e, com base nele, sintetiza um peptídeo / proteína. Os aminoácidos a partir dos quais o ribossomo constrói a proteína são entregues em complexo com RNA de transporte ( tRNA ). Esse processo é chamado de "transmissão" ;
- O peptídeo / proteína pode sofrer modificações pós-traducionais ("maturação" por analogia com o mRNA) e torna-se funcional. Um fator importante é que o sistema de modificação pós-traducional dos eucariotos é muito mais complexo e diversificado do que o dos procariontes, portanto, nem toda proteína eucariótica pode ser corretamente sintetizada por uma bactéria.
Além das regiões codificadoras, o genoma contém numerosos fragmentos que também participam da transcrição de uma maneira ou de outra. As parcelas localizadas próximas ao gene e chamadas promotoras são reconhecidas pelas RNA polimerases (eles dizem que o gene está sob o controle desse promotor). Diferentes promotores são reconhecidos por diferentes RNA polimerases. Por exemplo, um gene sob o controle de um promotor de
bacteriófago não será transcrito para bactérias se a RNA polimerase do bacteriófago correspondente não for sintetizada nele.
Geralmente .
Cada gene também pode ter várias seqüências reguladoras, que podem ser localizadas diretamente perto do promotor (ou até se sobreporem a ele) ou a uma distância de dezenas de milhares de pares de nucleotídeos. Os elementos de aprimoramento da transcrição são chamados de
“aprimoradores”, os elementos supressores de transcrição são chamados silenciadores e as proteínas que interagem com eles são chamadas de
fatores de transcrição . Embora também seja habitual chamar fatores de transcrição os componentes necessários do complexo de iniciação da transcrição, sem os quais a transcrição é impossível em princípio. O fato é que apenas para iniciar a síntese da molécula de RNA na matriz de DNA em eucariotos e arquéias, é necessária a montagem de todo o complexo supramolecular. O complexo mais simples desse tipo inclui a holoenzima da RNA polimerase e seis chamados
"fatores comuns de transcrição" (TFIIA, TFIIB, TFIID, TFIIE, TFIIF e TFIIH). O complexo em si é chamado de
"Complexo de pré-iniciação de transcrição" (
vídeo , cada componente do complexo é destacado em uma cor ou outra).
O complexo de transcrição procariótica é completamente diferente, portanto, não faz sentido incorporar o gene eucariótico ao promotor eucariótico da bactéria. Um análogo procariótico dos fatores de transcrição comuns de eucariotos e arquéias pode ser chamado de uma proteína chamada
"fator sigma" .
 Complexo transcricional procariótico. As letras mostradas na figura são designações geralmente aceitas das subunidades correspondentes. σ70 - fator sigma dos genes domésticos de E. coli
Complexo transcricional procariótico. As letras mostradas na figura são designações geralmente aceitas das subunidades correspondentes. σ70 - fator sigma dos genes domésticos de E. coliOs genomas de procariontes e eucariotos têm muitas características em comum, e o Dogma Central da Biologia Molecular, mencionado anteriormente, é verdadeiro para os dois reinos. No entanto, também existem muitas diferenças significativas. Por exemplo, uma bactéria é caracterizada por um sistema de operons - genes agrupados que participam do mesmo processo e não são transcritos separadamente, mas como parte de um longo mRNA. Nos eucariotos, tudo é completamente diferente: os genes envolvidos em um processo estão espalhados por diferentes cromossomos, e os próprios genes são divididos em fragmentos codificadores
de exons por regiões não codificadoras
de íntrons . Nesse caso, a princípio o gene é completamente transcrito e, então, já no estágio do RNA, os íntrons são excisados e os exons se cruzam para formar o mRNA codificador. Esse processo é chamado de
emenda . Ao mesmo tempo, nem todos os exons disponíveis podem ser costurados no mRNA final, mas apenas uma parte deles, nesse caso, fala em
"emenda alternativa" . Assim, uma célula eucariótica pode sintetizar várias proteínas, enquanto transcreve o mesmo gene. Entre outras coisas, isso dá uma conseqüência muito importante: muitas vezes não faz sentido inserir o gene eucariótico na bactéria "como no cromossomo", uma vez que a bactéria simplesmente não é capaz de se unir.
Há outra diferença importante. Os procariontes são caracterizados pela presença de material genético baseado em DNA fora do anel "cromossomo", os chamados
"plasmídeos" - pequenas moléculas circulares de DNA de fita dupla. Além disso, os procariontes carecem de organelas, incluindo o núcleo: todos os componentes de uma célula bacteriana são livres para viajar pelo espaço intracelular. Os eucariotos, no entanto, não possuem plasmídeos, mas existem
plastídeos e
mitocôndrias no genoma dos quais plasmídeos estão incluídos (de acordo com a hipótese mais fundamentada, plastídeos e mitocôndrias são “descendentes” da arquitetura procariótica do genoma de cianobactérias e bactérias capturadas por proto-eucariotos unicelulares antigos). Além disso, a presença de um núcleo e outros compartimentos intracelulares cercados por sua própria membrana já é típica para eucariotos. Portanto, a engenharia genética de células eucarióticas requer abordagens diferentes da engenharia genética de bactérias.
O próprio código genético está estruturado da seguinte maneira. Cada gene / éxon consiste em um conjunto de trigêmeos / códons - seqüências de três nucleotídeos entre os quais não há lacunas. A organização tripla é válida tanto para genes no DNA quanto para a parte codificadora do mRNA. No processo de tradução, os RNAs de transporte (tRNAs) que transportam um aminoácido específico "reconhecem" seus trigêmeos de três letras correspondentes. O ribossomo desconecta o aminoácido do tRNA e o anexa à crescente cadeia de aminoácidos, que ao final da tradução se transforma imediatamente em uma proteína madura e totalmente funcional, ou antes de sofrer uma série de modificações. Nesse caso, apenas um aminoácido corresponde a cada trigêmeo, mas vários códons diferentes podem corresponder a um aminoácido. Isso é compreensível, porque no código genético padrão existem 61 códons codificadores e existem
apenas 20 aminoácidos proteinogênicos (códons totais, é claro, 4 * 4 * 4 = 64, mas três deles não são codificantes, em vez disso, servem como um sinal para interromper a tradução e são chamados " parar os códons ").
 Códons no código genético padrão. Graças à Wikipedia para a imagem.
Códons no código genético padrão. Graças à Wikipedia para a imagem.Assim, as proteínas são precisamente aqueles elementos que são o último elo da cadeia entre o DNA genômico e as propriedades do corpo, o chamado
“fenótipo” . Portanto, para alterar de alguma forma as características do organismo que são importantes para nós, precisamos alterar seu DNA de tal maneira que, como resultado, certas proteínas apareçam em suas células, o que nos fornecerá o resultado desejado. Essa é a idéia básica de toda engenharia genética.
1) Para que fins as bactérias são usadas na engenharia genética e por que elas são
Então, descobrimos como e por que a sequência do DNA genômico afeta as propriedades e características do corpo. Obviamente, será muito bom se a característica for completamente determinada por apenas um gene - inserir um pequeno fragmento não será mais um problema sério. Por exemplo, geralmente a resistência de uma planta a um herbicida ou praga é determinada por um único gene, portanto, criar variedades com a resistência desejada em tais casos não é difícil (ao contrário de trazer essa planta ao mercado). O mesmo se aplica a muitos antibióticos resistentes a bactérias (de fato, as bactérias têm muitos mecanismos de proteção contra antibióticos, mas funcionam de maneira independente). O exemplo oposto é, por exemplo, uma tentativa dos cientistas de ensinar as plantas a absorver nitrogênio da atmosfera. O fato é que a única fonte de nitrogênio para as plantas é o solo no qual os compostos contendo nitrogênio adequados para assimilação pela planta são sintetizados por microorganismos (ou introduzidos como fertilizantes por um jardineiro ou um cachorro que passa). Obviamente, criar uma planta com um mecanismo nutricional alternativo seria muito benéfico para a agricultura. Infelizmente, esse processo é tão complicado que o problema de sua "transferência" do microorganismo para a planta ainda não foi resolvido.
Finalmente, se nosso objetivo é obter proteína para qualquer finalidade específica (estudar a estrutura e as funções da proteína, criar preparações médicas ou reagentes de laboratório com base nela, etc.), então, obviamente, também estamos muito felizes com a integração de um único gene na célula, que neste caso, é costume chamar o "organismo produtor".
Uma bactéria na engenharia genética é um material fonte potencial para criar:
- um produtor da proteína de que precisamos em escala laboratorial ou industrial;
- um agente ativo em uma certa transformação química de um composto em outro, seja um processo de fermentação na indústria de alimentos, a criação de condições mais favoráveis para o crescimento das plantas, introduzindo um “produtor de fertilizante bacteriano” no solo ou utilizando sucata de aço;
- klonotek de genes (um tópico cuja boa descrição aumentará o tamanho do artigo para indecente);
- uma droga medicamente significativa, por exemplo, para restaurar a microflora do trato gastrointestinal;
- estirpes bacterianas de Agrobacterium tumefaciens para posterior modificação genética de plantas.
* Eu poderia esquecer alguma coisa, então acréscimos nos comentários são bem-vindos.
Um fato interessante é que as primeiras experiências bem-sucedidas no campo da engenharia genética de bactérias ocorreram muito antes do trabalho de referência de Watson e Crick. Além disso, com base nessas experiências, foi comprovado o fato de que a informação está contida no DNA, após o que os cientistas não puderam gastar seu tempo em hipóteses sobre RNA e proteínas.
Este trabalho, realizado em 1944, é conhecido como
Experimento de Avery, MacLeod e McCarthy , baseado no
trabalho de Frederick Griffith , durante o qual foi constatado que a infecção por cepas de pneumococos patogênicas mortas e não patogênicas vivas causa o desenvolvimento da doença, enquanto individualmente eles não causam sintomas significativos. A partir desse experimento, concluiu-se que uma bactéria morta é capaz de transmitir algo a um "colega" não patogênico, como resultado do qual se torna perigoso. Mas o que eles transmitem um ao outro? Em 1944, havia três candidatos principais: DNA, RNA e proteína. Para estabelecer o transportador, foi realizado um experimento elegante: na época, já estavam disponíveis enzimas capazes de destruir separadamente DNA (DNase), separadamente RNA (RNase) e separadamente proteínas (proteinase). Foi demonstrado que a transferência de propriedades patogênicas não ocorreu apenas nos casos em que a preparação de uma cepa patogênica morta foi tratada com DNase e não dependia do tratamento da droga com RNase e proteinase.
Assim, ficou provado que o DNA é o portador de informações sobre os sinais. Além disso, foi mostrado claramente que é possível a penetração espontânea de uma molécula de DNA estranha em uma célula bacteriana.
Por que as bactérias são tão populares com falhas óbvias (por exemplo, a falta de modificações pós-traducionais eucarióticas)? Tudo é simples.
Eles são despretensiosos em operação, fáceis de usar e não requerem meios nutritivos caros.2) Como é criado um construto genético que é introduzido na bactéria
A engenharia genética moderna de bactérias é principalmente a introdução de um vetor plasmídico (um plasmídeo bacteriano modificado contendo o gene alvo e um conjunto de outros elementos necessários, que serão discutidos abaixo). Alterar o cromossomo bacteriano é menos típico, mas esse procedimento também não é estranho: por exemplo, o gene da RNA polimerase do bacteriófago T7 foi introduzido no cromossomo E. coli usando um vetor baseado no profago λ durante a criação de uma das cepas populares na placa de laboratório. Há três razões pelas quais um pesquisador geralmente escolhe introduzir um gene em um vetor plasmídico:- primeiro, introduzir um vetor plasmídeo em uma bactéria é mais barato do que incorporar algo em um cromossomo;
- -, , ;
- -, .
Um vetor plasmídico típico para trabalhar com bactérias é uma pequena molécula de DNA circular de fita dupla que transporta o gene da proteína alvo sob o controle de um promotor específico e vários genes e elementos reguladores necessários, cuja presença garante uma quantidade constante de plasmídeo na célula ("controle de cópia"). Obviamente, mesmo no caso de síntese ultra-eficiente de mRNA, o vetor é de pouca utilidade se existir nas bactérias na quantidade de um par de pedaços: no processo de divisão, a probabilidade de formação de células filhas sem o plasmídeo necessário será banal.Além do gene e do promotor, os principais elementos do vetor plasmídeo são:- ori é a região de origem da replicação do plasmídeo. Necessário para manter uma quantidade constante de plasmídeo e sua herança pelas células filhas;
- — , , , . , , (« »). , , . , .
, β- (GUS). , . , . — (GFP) ( GUS GFP );
- , ( — , — );
- — , ( ). , «» .
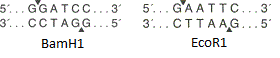 A figura mostra os locais de restrição de endonuclease BamH1 e EcoR1 . Ambas as enzimas reconhecem um local específico a partir de seis pares de bases e introduzem rupturas de fita simples em lugares diferentes (indicadas por setas triangulares). Nesse caso, os pontos de quebra da corrente não coincidem, o que significa que são formadas "extremidades adesivas" (se coincidem, são formadas "extremidades cegas").
A figura mostra os locais de restrição de endonuclease BamH1 e EcoR1 . Ambas as enzimas reconhecem um local específico a partir de seis pares de bases e introduzem rupturas de fita simples em lugares diferentes (indicadas por setas triangulares). Nesse caso, os pontos de quebra da corrente não coincidem, o que significa que são formadas "extremidades adesivas" (se coincidem, são formadas "extremidades cegas").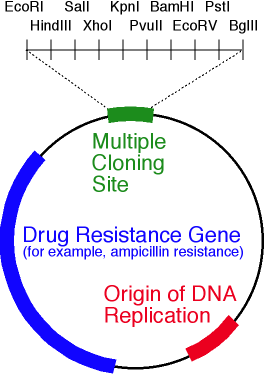 Um esquema simplificado de um vetor plasmídico. A figura mostra ori, o gene de resistência a antibióticos e o poliligante contendo 10 locais de endonuclease de restrição.Bem, o vetor está em nossas mãos. Como incorporar um gene nele? E de qualquer maneira, onde posso obter esse gene?Suponha que conheçamos a sequência nucleotídica do gene de que precisamos. Em seguida, proceda da seguinte maneira:
Um esquema simplificado de um vetor plasmídico. A figura mostra ori, o gene de resistência a antibióticos e o poliligante contendo 10 locais de endonuclease de restrição.Bem, o vetor está em nossas mãos. Como incorporar um gene nele? E de qualquer maneira, onde posso obter esse gene?Suponha que conheçamos a sequência nucleotídica do gene de que precisamos. Em seguida, proceda da seguinte maneira:- , ;
- .
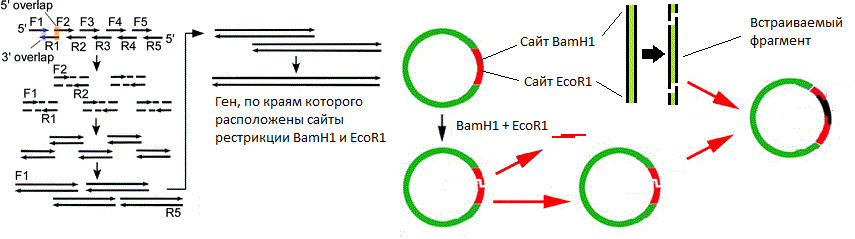 ( ). .
( ). ., , , . , (
BamH1 EcoR1 ) , , « » . , . ,
-, que elimina quebras de cadeia em moléculas de DNA de fita dupla.Outro fator importante na montagem de genes é o fato de que as frequências de vários códons são diferentes para diferentes organismos, enquanto nas células geralmente há mais desses tRNAs que correspondem a códons mais “populares”. Como muitos aminoácidos são codificados por vários códons, é altamente provável que, copiando um gene de um organismo para outro sem pensar, corremos o risco de sofrer um grande atraso no processo de tradução. De fato, se muitos códons desse gene são raros no novo organismo, o ribossomo aguardará mais tempo quando o tRNA desejado finalmente chegar. , , , , , . E. coli .Agora, algumas palavras sobre o promotor. A seleção de um promotor adequado é muito importante, pois o processo de transcrição depende em grande parte dele. Os promotores são condicionalmente divididos em forte, médio e fraco. A "força" do promotor é determinada pela forma como os genes são transcritos sob seu controle, e todas as outras coisas são iguais: quanto mais ativa a transcrição, mais forte o promotor. Obviamente, quando queremos criar um produtor de proteínas, devemos começar com fortes promotores. Em alguns casos, a transcrição excessivamente rápida (portanto, a tradução ativa) prejudica a célula; nesse caso, você pode tentar usar um promotor mais fraco. Embora, de fato, seja muito mais fácil influenciar a atividade de transcrição de um produtor existente do que criar uma nova.Outra coisa é importante. Freqüentemente, as proteínas codificadas pelo vetor têm um efeito extremamente negativo na viabilidade bacteriana. Não apenas a síntese dessas proteínas consome uma grande quantidade de recursos (e a quantidade da proteína alvo não deve ser inferior a 10% do peso seco total da célula), como também flutua para frente e para trás através do citoplasma ! Portanto, por enquanto, é melhor desativar completamente a expressão de um gene estranho à célula. Para esse fim, foram desenvolvidos sistemas de expressão controlada que permitem "ativar" a expressão do gene de que precisamos "sob comando". Os mais comuns são:
, , , , , . E. coli .Agora, algumas palavras sobre o promotor. A seleção de um promotor adequado é muito importante, pois o processo de transcrição depende em grande parte dele. Os promotores são condicionalmente divididos em forte, médio e fraco. A "força" do promotor é determinada pela forma como os genes são transcritos sob seu controle, e todas as outras coisas são iguais: quanto mais ativa a transcrição, mais forte o promotor. Obviamente, quando queremos criar um produtor de proteínas, devemos começar com fortes promotores. Em alguns casos, a transcrição excessivamente rápida (portanto, a tradução ativa) prejudica a célula; nesse caso, você pode tentar usar um promotor mais fraco. Embora, de fato, seja muito mais fácil influenciar a atividade de transcrição de um produtor existente do que criar uma nova.Outra coisa é importante. Freqüentemente, as proteínas codificadas pelo vetor têm um efeito extremamente negativo na viabilidade bacteriana. Não apenas a síntese dessas proteínas consome uma grande quantidade de recursos (e a quantidade da proteína alvo não deve ser inferior a 10% do peso seco total da célula), como também flutua para frente e para trás através do citoplasma ! Portanto, por enquanto, é melhor desativar completamente a expressão de um gene estranho à célula. Para esse fim, foram desenvolvidos sistemas de expressão controlada que permitem "ativar" a expressão do gene de que precisamos "sob comando". Os mais comuns são:- Um sistema baseado em elementos reguladores do lactose operon E. coli (lacperon) e um forte promotor.
O fato é que E. coli tem suas próprias regras nutricionais. Em primeiro lugar, existe um mecanismo para suprimir a atividade do lac- peron, que é ativado somente quando a lactose não entra na célula. Isso é lógico: por que desperdiçar energia na síntese do que não é útil? Mas assim que a lactose começa a entrar na célula em quantidades suficientes, esse mecanismo é desativado.
No entanto, existe um segundo mecanismo para suprimir a atividade do lac- peron. Se houver glicose no meio, a célula se alimenta exclusivamente de glicose, uma vez que ativa o segundo mecanismo de inibição da transcrição do lac- peron. Assim, o lac- peron é ativo apenas quando existe apenas lactose no espaço que circunda a célula. O menos do operon lactose é um promotor extremamente fraco, portanto, nas linhagens produtoras, ele é substituído por um forte. Promotores fortes geralmente são derivados de patógenos. Os promotores mais fortes mais amplamente utilizados na engenharia genética de procariontes são isolados de vírus bacterianos - bacteriófagos . Por exemplo, o promotor do fago T7 é amplamente utilizado.
A propósito, alguns fortes promotores da engenharia genética das plantas também são isolados de vírus, por exemplo, este é o promotor do vírus do mosaico da couve-flor.

Como mencionado acima, E. coli não possui uma RNA polimerase que reconhece promotores de bacteriófagos; portanto, o gene da RNA polimerase do bacteriófago correspondente é previamente inserido no produtor.
O popular sistema de síntese de proteínas à base de E. coli transporta o gene da RNA polimerase do fago T7 sob o controle do promotor da RNA polimerase bacteriana, regulado pelo mecanismo de laceron . Se essa cepa for transformada com um vetor que transporta o gene alvo sob o controle do complexo "fago T7 + regulação do complexo promotor tipo laceron ", surgirá um mecanismo de dois níveis de inibição da transcrição do gene alvo.
Ao usar esse design, glicose e lactose são adicionadas ao meio nutriente simultaneamente. Por algum tempo, as células se alimentam de glicose e se dividem silenciosamente, já que a síntese de uma proteína estranha é completamente suprimida. Quando a glicose termina e as células mudam para o metabolismo da lactose, já haverá biomassa suficiente na cultura, é o momento de iniciar a síntese da proteína de que precisamos. Este procedimento é chamado de "auto-indução".
Você pode fazer isso de outra maneira: não adicione glicose e lactose ao meio nutriente e, quando a cultura atingir a densidade desejada, adicione o que a célula precisará para a lactose, mas não pode metabolizá-la ou destruí-la. Agora, o IPTG é usado como um indutor.
- Um sistema baseado no mecanismo regulador do promotor pL do bacteriófago λ .
Este promotor é inativado pela proteína repressora cI. Nesse caso, uma forma termossensível dessa proteína chamada cI857 foi descoberta: esse fator de transcrição mantém a funcionalidade a uma temperatura de cerca de 30 ° C e a perde a 42 ° C. Portanto, ao usar esse sistema, a cultura bacteriana é primeiro cultivada até a densidade desejada a 30 ° C e, em seguida, a temperatura é aumentada para 42 ° C, iniciando assim a síntese da proteína alvo.
Bem, o vetor é projetado. Então, o mais pequeno é encontrar um método adequado para sua introdução na célula bacteriana. Mas esta é uma história completamente diferente.