Olá pessoal, meu nome é Alexander Sokolov e quero lhe contar como fiz um seqüenciador em casa - um dispositivo para decodificar o DNA. O preço de mercado desse dispositivo é de cerca de 10 milhões de rublos.

Uma breve excursão à genética. Se você se lembra de repente, em 2003, foi feita uma declaração sensacional: os cientistas finalmente decifraram o genoma humano. O genoma é construído a partir do DNA, e o DNA é o código fonte do corpo. O DNA é uma cadeia dupla composta por 4 tipos de nucleotídeos, que são repetidos no genoma humano cerca de 3 bilhões de vezes. Assim como todas as informações do seu computador são criptografadas em bits, as instruções para reunir todas as proteínas do corpo humano são criptografadas em nucleotídeos. Ou seja, sabendo em que sequência os nucleotídeos do DNA estão localizados, teoricamente podemos coletar todas as proteínas necessárias e obter um modelo humano. Portanto, no sentido padrão, os cientistas não decifraram o DNA, mas simplesmente traduziram a sequência química em um conjunto de zeros e zeros no computador. O que fazer a seguir é uma conversa em separado. Por exemplo, no momento, estamos claros sobre a função de apenas 5% de toda a matriz genômica (isso é codificação de proteínas). O que os 95% restantes fazem, só se pode especular.
Em 2003, o custo do seqüenciamento de DNA humano foi de cerca de US $ 100 milhões. Com o tempo, esse número diminuiu e agora está se aproximando de mil dólares. Você paga, seu DNA é sequenciado e recebe um disco rígido com 3 GB de informação - seu genoma em formato digital.
Existem três sequenciadores principais no mercado hoje. O mais produtivo, o Hiseq, e seu receptor NovaSeq, fornecem o seqüenciamento mais barato (fluorescente). Um de seus lançamentos dura vários dias e, durante esse período, os genomas de várias pessoas são processados ao mesmo tempo. No entanto, o próprio lançamento custa cerca de dez mil dólares. A propósito, o dispositivo em si custa cerca de US $ 1 milhão e, como se torna obsoleto em cerca de 3 anos, para ser recompensado, deve gerar US $ 1000 por dia.
O segundo dispositivo apareceu no mercado no último verão. É chamado Nanopore e é baseado em uma tecnologia muito interessante quando o DNA é sequenciado passando por um nanoporo. A variante mais barata da Nanopore está posicionada como um seqüenciador doméstico descartável e custa US $ 1000.
O terceiro dispositivo é o PGM, um sequenciador de semicondutores que custa US $ 50.000 em sua terra natal e cerca de 10 milhões de rublos (com entrega, desembaraço aduaneiro etc.) na Rússia. O processo de seqüenciamento leva cerca de várias horas.
Bem, eu não tinha dez milhões, mas queria o PGM. Eu tive que fazer isso sozinho. Primeiro, brevemente sobre como ocorre o seqüenciamento de semicondutores. Toda a cadeia de DNA é dividida em fragmentos de 300 a 400 nucleotídeos de comprimento, chamados de leituras. Em seguida, as leituras são anexadas a pequenas esferas e são copiadas muitas vezes - como resultado, um monte de fragmentos de DNA idênticos “paira” em cada esfera. É necessário copiar para amplificar o sinal de cada leitura específica. Um conjunto de áreas diferentes é chamado de biblioteca de DNA.
O coração do PGM é um chip único - uma matriz semelhante à matriz da câmera, mas em vez de pixels que respondem à luz, aqui estão os transistores de pH que respondem a mudanças no equilíbrio ácido-base. A biblioteca de DNA resultante é carregada em um chip contendo 10 milhões de poços; no fundo de cada um deles há um transistor de pH. Apenas uma esfera se encaixa em um buraco e, portanto, lê apenas um tipo (com uma sequência nucleotídica específica). Em seguida, os reagentes são alimentados no chip para que o DNA comece a se copiar. E é copiado linearmente, isto é, os nucleotídeos são ligados à cadeia recém-criada na ordem em que estão na cadeia mãe. Portanto, um tipo de nucleotídeo é alimentado ao chip - e a alteração do pH em alguns poços é registrada imediatamente (isso significa que o nucleotídeo foi anexado a eles). Então, um tipo diferente de nucleotídeo é alimentado e as mudanças de pH nos poços etc. são registradas.Portanto, aplicando ao chip todos os 4 tipos de nucleotídeos muitas vezes, podemos obter informações sobre a sequência de nucleotídeos em cada leitura. Então, por métodos matemáticos, os segmentos curtos de leitura são coletados em um computador em uma única cadeia. Para coletá-lo com mais ou menos confiança, cada leitura deve ser lida cerca de 100 vezes.
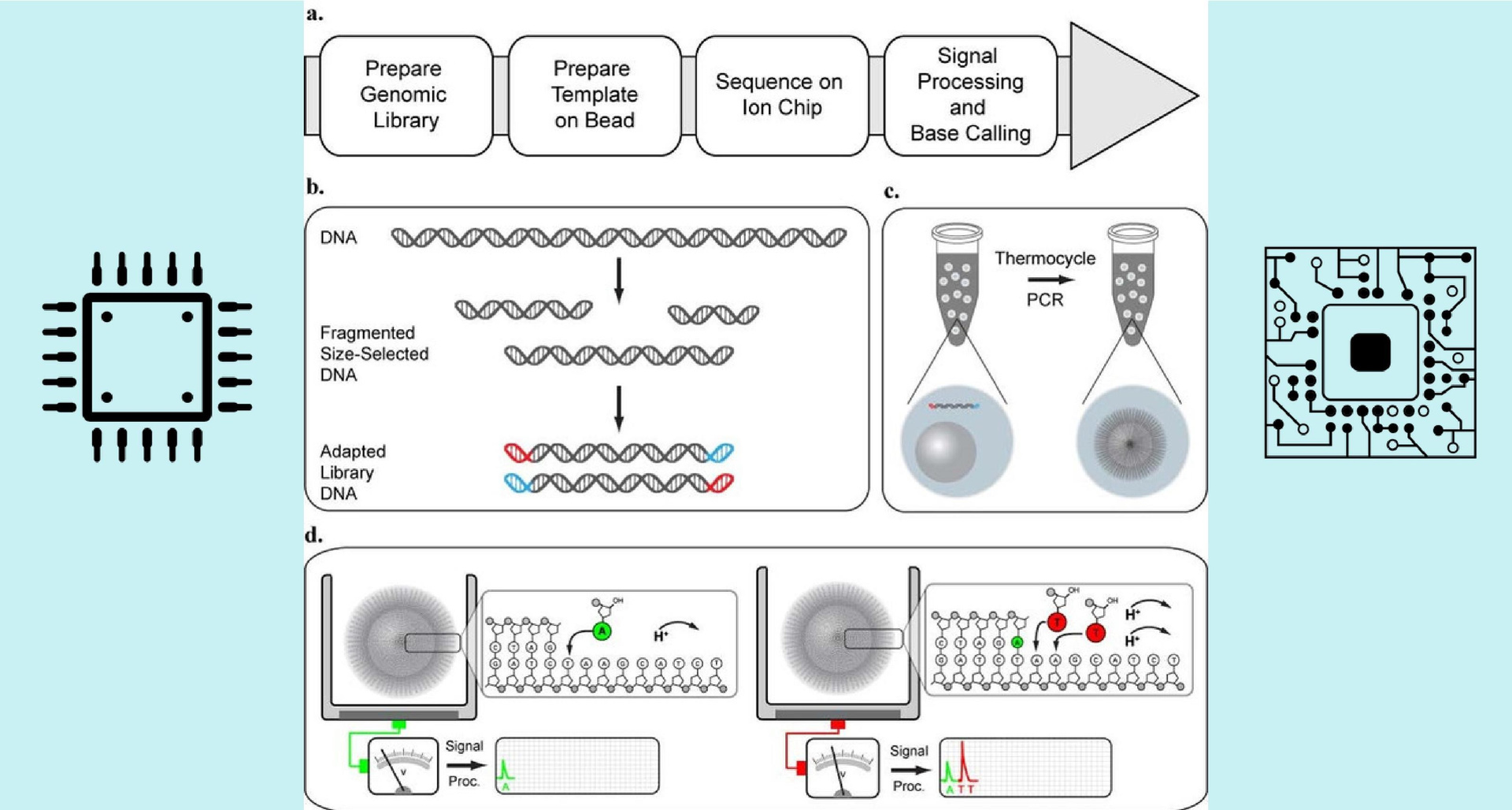 Fig. 1. Sequenciamento de semicondutores
Fig. 1. Sequenciamento de semicondutoresAgora vamos descobrir em que consiste o próprio dispositivo. Existe, como já sabemos, um chip, um sistema de suprimento de reagentes e uma placa-mãe. Todo o seqüenciamento é realizado precisamente no chip - o resto do dispositivo apenas transmite certos sinais para ele, entrega reagentes, lê sinais analógicos, digitaliza-os e direciona o fluxo de informações recebido para um computador, onde os dados são acumulados e processados.
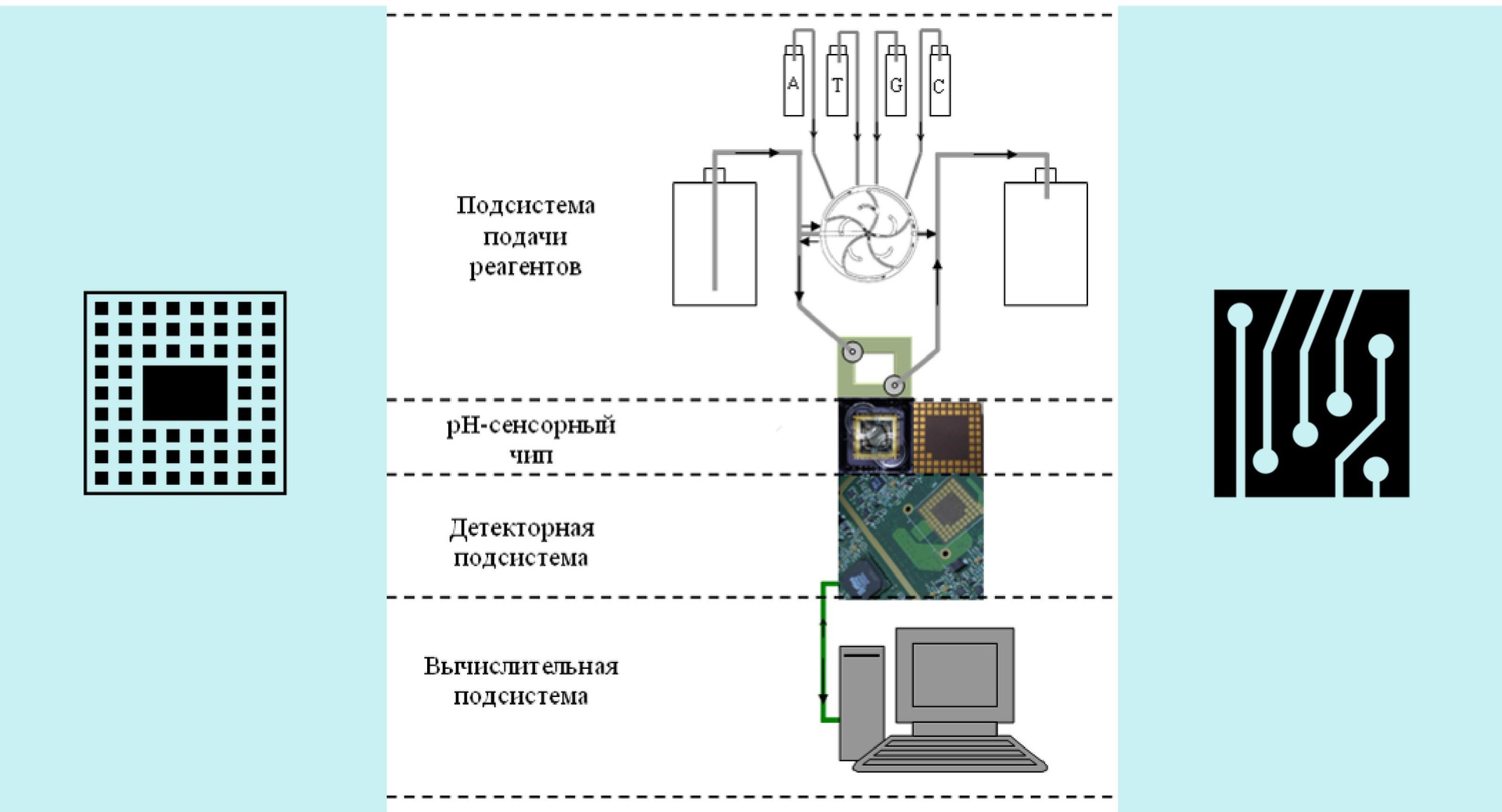 Fig. 2. Dispositivo sequenciador
Fig. 2. Dispositivo sequenciadorO chip é posicionado como único e é jogado fora após o uso. Por conseguinte, onde o PGM funciona, esses chips podem ser obtidos gratuitamente em qualquer quantidade. Por que pegá-los, você pergunta? O fato é que eu já consegui usar o chip repetidamente. De fato, é eterno: enxágüe bem o suficiente - e pode ser usado repetidamente. Em precisão, não será diferente do novo. Minha idéia era criar um dispositivo para esse chip shareware.
Então, eu enfrentei a tarefa de fazer engenharia reversa do chip. Obviamente, não foi encontrada nenhuma documentação para o microcircuito cobiçado - o fabricante não iria compartilhar os segredos da produção, mas queria vender seus dispositivos em silêncio por US $ 50.000. Para começar, fiz o mais óbvio e simples: os contatos tocaram com um testador. Ficou claro onde estão localizadas as E / S digitais e analógicas, energia e muito mais. Algumas informações foram obtidas de patentes no chip. Mas tudo isso, é claro, não foi suficiente para criar um produto completo. Eu ainda brinquei com o chip, verifiquei minhas várias suposições, experimentei dar sinais, mas basicamente não avançou em lugar algum. Eu tive que pausar o projeto.
 Fig. 3. Chip
Fig. 3. ChipE de repente, em Habrahabr, me deparei
com um artigo do famoso blogueiro
BarsMonster sobre como ele faz engenharia reversa de chips! Ele se inspirou, escreveu para ele, escreveu para outros entusiastas, enviou um pedido a Kiev, onde estava envolvido em fotografar chips. Eles responderam em Kiev que não sabem polir em camadas, só podem filmar a camada superior e, como meu chip é de várias camadas, não ficará claro para onde vão as faixas dos contatos. Então, ele conheceu um americano que também está envolvido em engenharia reversa de chips, enviou seus microcircuitos, mas mesmo aqui, não foi além de fotografar a camada superior. Então me deparei com um artigo na Internet sobre aqueles que conseguiram reverter o chip Sony PlayStation etc. (“Glória aos heróis!”) E isso é tudo, se alguém souber). Decidi escrevê-las com perguntas, encontrei seus apelidos - e então percebi que um deles era familiar para mim. Recentemente, um amigo me levou a um amigo que também está “engajado em genética no nível amador”, conversamos com esse amigo no Skype e terminamos esse diálogo. E agora entendo que meu novo amigo é um mestre mega legal de chips de engenharia reversa. Escrevi para ele ali mesmo. Contudo, apesar de estar pronto para ajudar, ele não tinha um microscópio. Beco sem saída novamente.
Alguns meses depois, o microscópio necessário foi encontrado em um laboratório próximo! É verdade que a câmera embutida era terrível, tirei fotos em um telefone celular através de uma ocular e recebi fotos dessa qualidade:
 Fig. 4. O chip sob o microscópio
Fig. 4. O chip sob o microscópioEntão, no último ano novo, um excelente microscópio para 130 mil apareceu em meu trabalho (sou especialista em criptografia quântica). Sonhos se tornam realidade. Por fim, consegui fotografar normalmente o chip de cima.
 Fig. 5. Meu microscópio de trabalho
Fig. 5. Meu microscópio de trabalhoE então ... Então eu ainda tinha que dominar a técnica de polir eu mesma. A dificuldade do polimento é remover camadas de metal com uma espessura da ordem de 1 mícron - enquanto a largura do chip é de 1 centímetro. Para comparação, direi que é o mesmo que permitir um erro não superior a 10 cm por 1 km. Tentei muito. Os resultados do meu trabalho são apresentados na seguinte foto:
 Fig. 6. Engenharia reversa sob um microscópio óptico
Fig. 6. Engenharia reversa sob um microscópio ópticoA camada inferior de silício, a camada superior com transistores, a primeira, a segunda, a terceira e a quarta camadas de metal são claramente visíveis.
O chip consiste em zonas repetidas (como registros de turno) e, a partir dessas imagens, era muito conveniente analisá-lo: ficou imediatamente claro o que estava acontecendo em diferentes camadas. Eu "inverti" as seções mais "empalhadas" com uma abundância de lógica, que foram repetidas várias vezes. Mas o mais difícil era rastrear as trilhas que percorriam o chip inteiro, entender o que o contato externo se refere a quê. Desde o feriado de Ano Novo até o final de fevereiro, eu, armado com um novo microscópio bonito, debruçamos sobre essa tarefa - fiquei no trabalho até as dez da noite, "invertido", pensei. E então um novo milagre aconteceu: um amigo conseguiu organizar a fotografia gratuita do chip em camadas em um microscópio eletrônico no MIREA. Migração de "sessão de fotos" no 1º trimestre cm eram 50 GB de fotografias em preto e branco.
Agora todas essas fotografias individuais tinham que ser de alguma forma combinadas em uma imagem inteira. Quase no mesmo dia, escrevi um programa em python que gerava um arquivo HTML - quando o abri em um navegador, obtive o que precisava. (A propósito, a 10ª Ópera mais antiga fez o melhor, eu recomendo!) Então escrevi outro programa em javascript que permite comparar camadas, fazer uma transição suave entre elas, alinhar, selecionar a escala etc. Finalmente, nas minhas mãos havia todas as ferramentas para resolver os principais problemas. Rastreei os caminhos que perfuravam o chip e restaurei toda a estrutura até o último transistor.
Outra fotografia de uma fatia de chip tirada sob um raio-x (no MIREA):
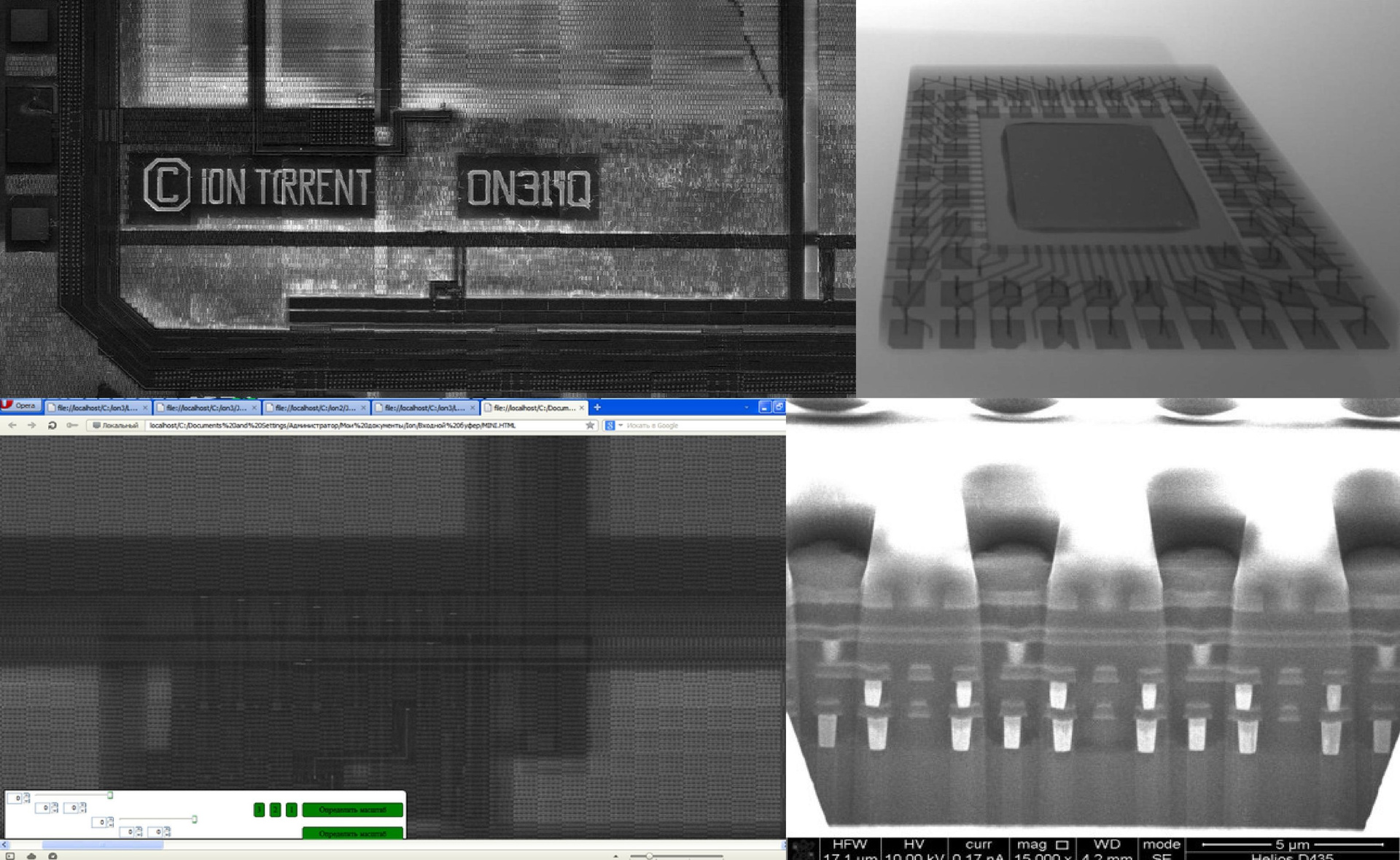 Fig. 7. microscopia eletrônica
Fig. 7. microscopia eletrônicaOs poços são visíveis onde as esferas com leituras caem. Abaixo estão três camadas de metal, e ainda mais baixas - uma camada com transistores.
O próximo estágio na luta por um futuro melhor foi a criação de uma placa-mãe para o chip. Projetou e enviou uma ordem de produção. Enquanto isso, o tribunal e o caso usaram a placa Mars Rover-2 com FPGA para trabalhar com o chip. (O FPGA é, grosso modo, uma matriz de 10.000 elementos lógicos universais; ao programar o FPGA, podemos obter qualquer lógica que processe facilmente fluxos de informações de gigabits.) Eu mesmo escrevi o firmware para o FPGA e, além disso, escrevi um software para controlar dinamicamente o sistema. que define toda a configuração do FPGA. Então, um intervalo de meio ano reapareceu (ele viajou a negócios para o lago Baikal, preparou uma planta em laboratório, o que foi demonstrado por Putin). Mas, no final, as estrelas se juntaram: eu tive tempo, as placas prontas chegaram - e montei meu sistema.
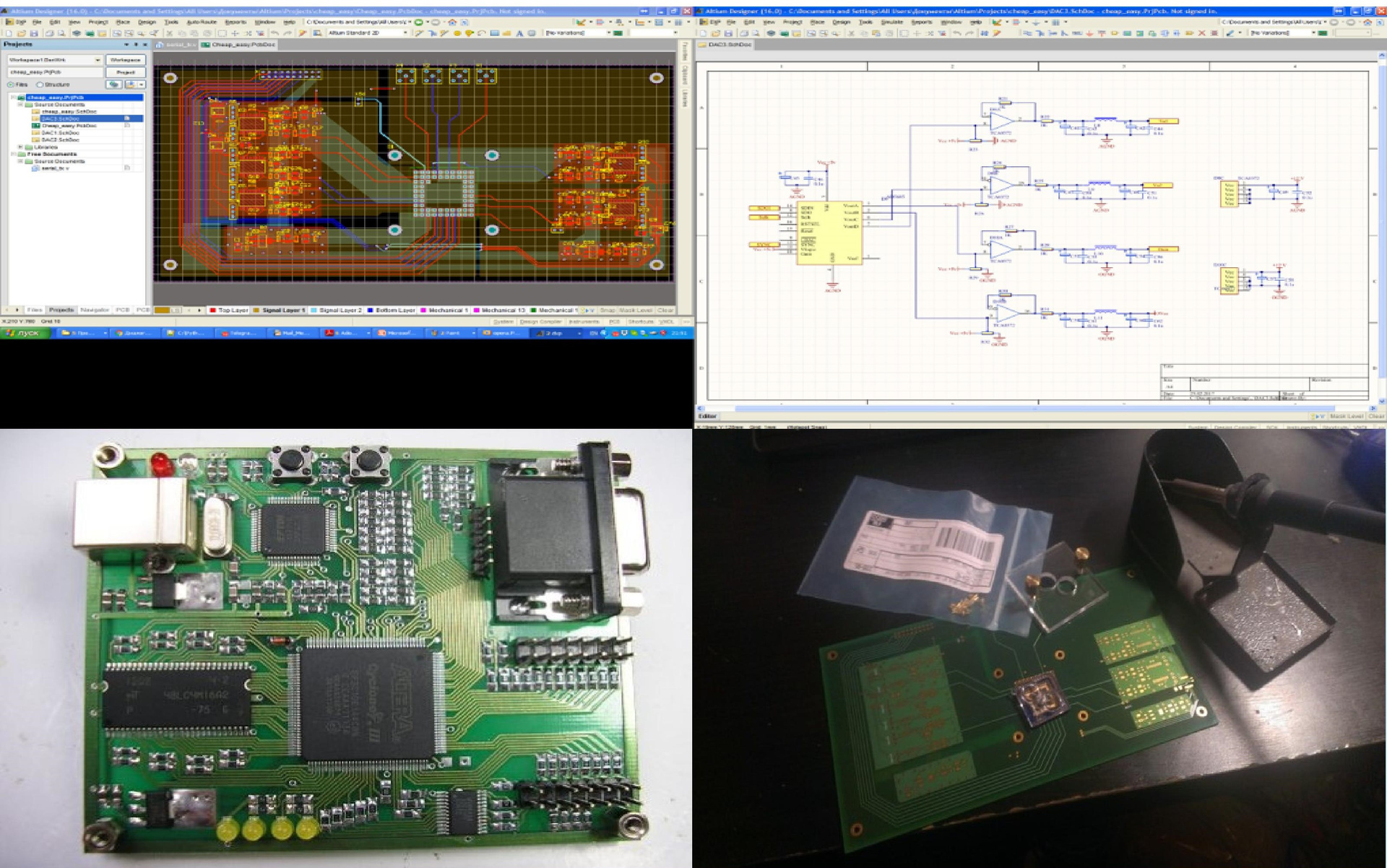 Fig. 8. A criação de "ferro"
Fig. 8. A criação de "ferro"Ele deu todos os sinais necessários e - oh, um milagre! Vi um sinal do chip no osciloscópio. (Uma vez comprei um osciloscópio por 6.000 rublos no eBay, outro firmware com custo de 1.000). Os pontos são claramente visíveis na imagem - gotas de algum reagente.
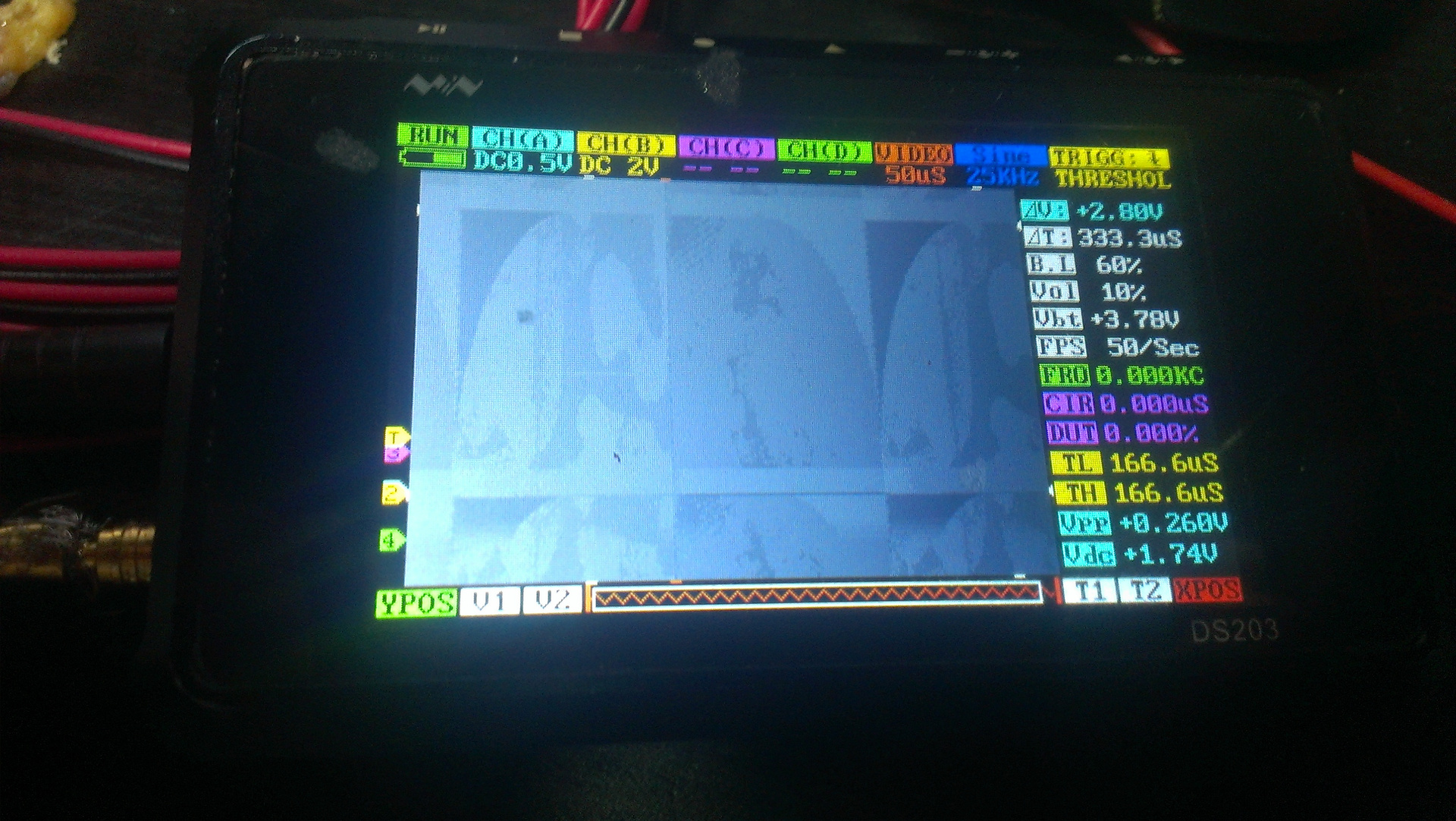 Fig. 9. O sinal do chip no osciloscópio
Fig. 9. O sinal do chip no osciloscópioAgora eu precisava descobrir como digitalizar essa imagem e transferi-la para um computador. Eu montei essa configuração:
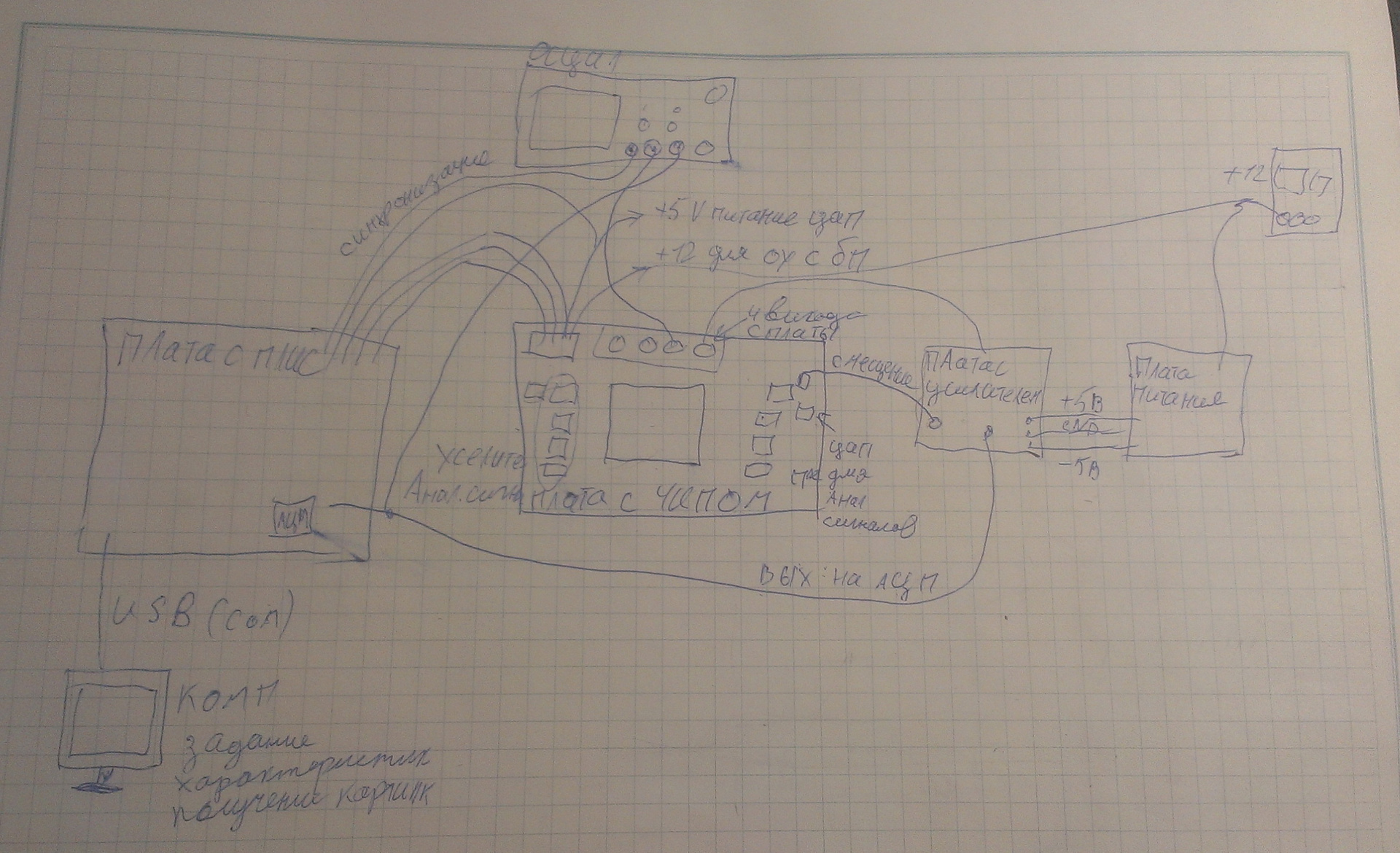 Fig. 10. Circuito de instrumento
Fig. 10. Circuito de instrumento Fig. 11. Instalação pronta
Fig. 11. Instalação prontaHá um computador que fornece dados de controle à placa com FPGA. A placa gera sinais digitais e os envia para o chip. O sinal do chip vai para o amplificador, depois para o ADC na placa, é digitalizado e transmitido através da porta COM para o computador. Em geral, a largura de banda da porta COM é pequena: 15 kilobits por segundo (já que existem de 1 a 10 milhões de "pixels" em um chip e a velocidade máxima de transmissão é de 115200 baud). No entanto, a imagem acaba chegando ao computador.
 Fig. 12. O sinal processado no computador.
Fig. 12. O sinal processado no computador.A foto acima mostra que, quando uma biblioteca de DNA é fornecida ao chip usado usado, o chip é preenchido de maneira desigual: nas bordas - em menor grau. Cores diferentes são devidas a diferentes voltagens nos transistores de pH. Ou seja, podemos distinguir claramente os buracos em que as esferas com leituras atingem - mais tarde, isso nos ajudará a controlar a liberação do chip.
Consequentemente, a próxima tarefa foi liberar o chip. Era necessário garantir que ele se tornasse novo. Felizmente, eu tinha um chip completamente novo como amostra de referência. Na Fig. E pode-se ver que na região ativa esse chip é quase da mesma cor (listras verticais de repetição são apenas ruído, interferência).
 Fig. 13. Lavando o chip
Fig. 13. Lavando o chipNa fig. 13 B O chip não foi bem lavado - é multicolorido. Na Figura 13, D é um chip usado mas bem lavado. É visto que o gradiente ao longo das bordas desapareceu. No entanto, valeria a pena provar que é realmente limpo e pode ser reutilizado.
Como as bibliotecas de DNA são conectadas ao revestimento de tântalo do chip em meio ácido e destacadas em alcalinas (isto é, em pH alto), o chip é lavado usando pipetas semi-automáticas especiais com soluções com pH diferente. Até o momento, consegui uma limpeza quase completa dos cavacos.
Eu estava interessado em saber por que, quando compreendi completamente a estrutura do chip, não pedi sua fabricação, mas preferi continuar a procurar e me acostumar, a mexer na lavagem, etc. Sim, porque o desenvolvimento do microcircuito custa muito dinheiro, milhões de dólares, e uma parte substancial desse valor é destinada à depuração física do produto resultante: ajustar, ajustar todos os parâmetros dos transistores etc. Ou seja, simplesmente copiar o circuito lógico não é suficiente. Portanto, tomo microcircuitos condicionalmente gratuitos, prontos - projetados, fabricados, depurados - e assim economizo dinheiro significativo, barateando seriamente o projeto.
Minha próxima tarefa foi montar um dispositivo mais avançado, o que permitiria uma transferência mais rápida de informações para um computador e, ao mesmo tempo, não consistiria em um grande número de placas separadas.
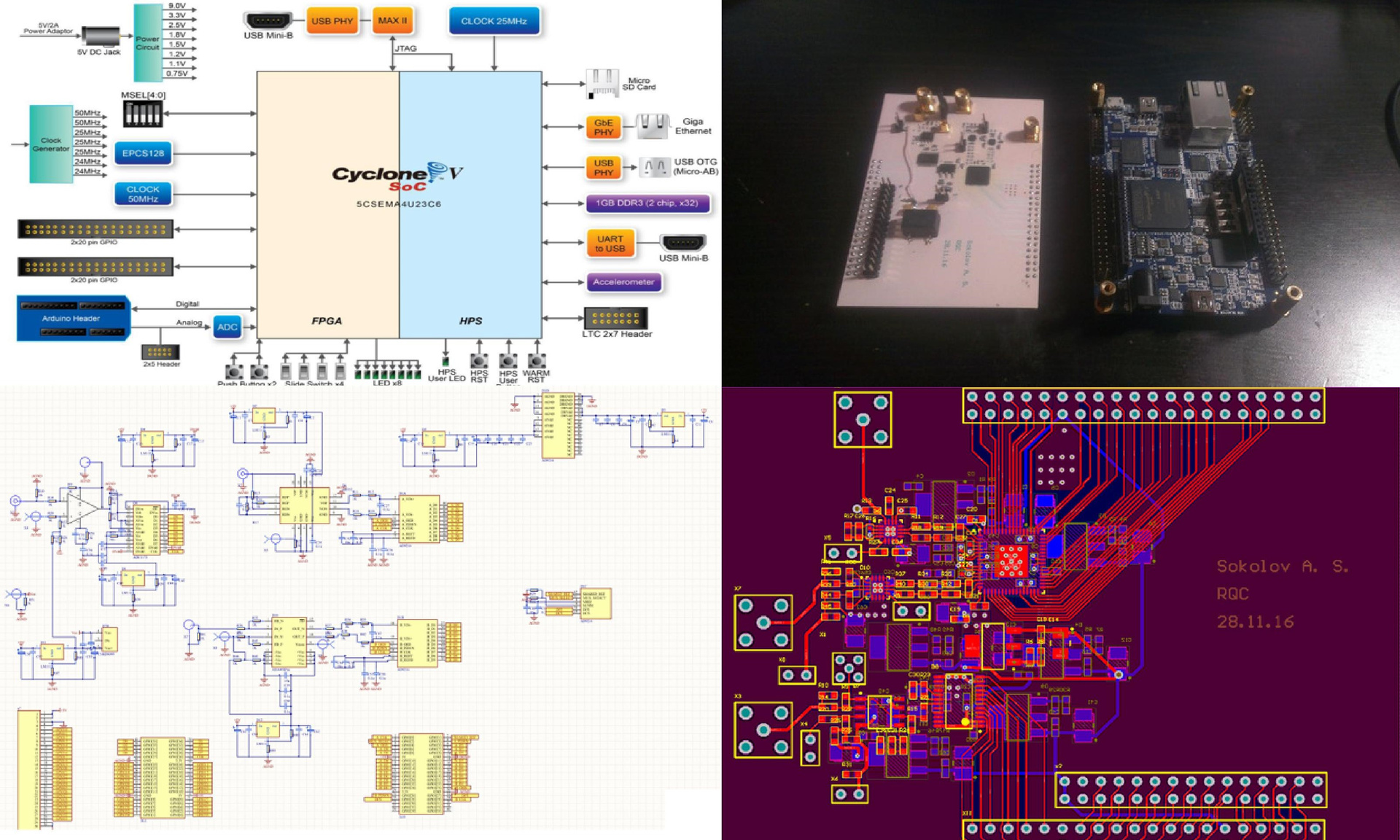 Fig. 14 Desenvolvimento da próxima versão do dispositivo
Fig. 14 Desenvolvimento da próxima versão do dispositivoPeguei uma nova placa-mãe com FPGA - no mesmo chip havia 2 núcleos ARM com Linux, Gigabit Ethernet e outros "brindes", mas, ao contrário da versão anterior, não havia ADC. Mais tarde, ele projetou outra placa, com ADCs de alta velocidade e todos os outros elementos necessários. Lançado - tudo funcionou.
O que resta a ser feito para a aparência do dispositivo final? Apenas três coisas.
O primeiro. Precisa de internet gigabit, transferência rápida de dados para um computador. Eu percebi isso ontem.
O segundo Sistema de alimentação de reagentes. O projeto de uma válvula especial já está em andamento.
O terceiro. Software para processamento de informações do chip. Ainda tenho dúvidas com o software, então convido os programadores a colaborar.
O dispositivo final custa 10 milhões de rublos. O custo do seqüenciamento é de vários milhares de dólares. Os chips custam de US $ 100 a US $ 1.000 - dependendo do número de "pixels" neles. (A propósito, a restauração de chips por si só pode ser uma boa receita, especialmente considerando que são necessários apenas alguns cliques para liberar a descarga.) Os reagentes também são comprados, mas no futuro serão criados.
Em geral, tudo isso é muito interessante, mas o principal é o futuro. Hoje, a biotecnologia ocupa o mesmo lugar no progresso científico e tecnológico do mundo que a informática nos anos 80. século passado. Além disso, o seqüenciamento é uma das áreas principais da biologia e da medicina modernas. E, claro, a biotecnologia é muito lucrativa.
Recentemente, o sequenciador de semicondutores S5 apareceu no mercado e, em um futuro próximo, pretendo mudar para ele.
Ficarei feliz em conversar com todos que desejam participar de uma maneira ou de outra no desenvolvimento deste projeto!
O projeto não teria sido possível sem o treinamento teórico de
Vladimir Zubov . Expresso minha gratidão a ele.
Obrigado pela atenção!