Muitas pessoas usam o termo DNA. Mas quase não há artigos descrevendo normalmente como funciona (não é compreensível para os biólogos). Já descrevi em termos gerais a
estrutura da célula e os próprios fundamentos de seus
processos energéticos . Agora vamos ao DNA.
O DNA armazena informações. Todo mundo sabe disso. Mas aqui está como ela faz isso?
Vamos começar com o local onde está armazenado na célula. Aproximadamente 98% são armazenados no núcleo. O resto está nas mitocôndrias e cloroplastos (a fotossíntese ocorre nesses indivíduos). O DNA é um enorme polímero composto de unidades de monômero. Parece algo assim.
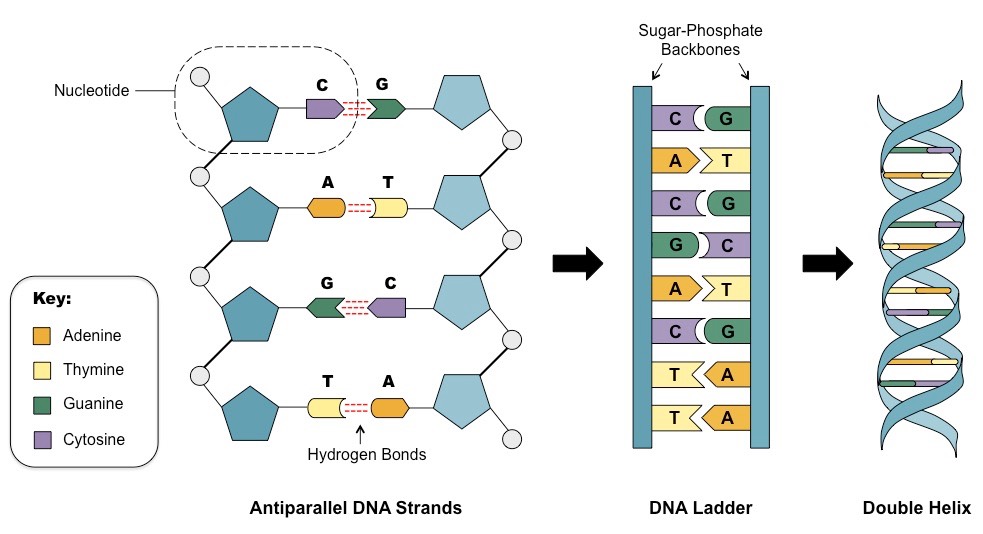
O que vemos aqui? Primeiro, o DNA é uma molécula de fita dupla. Por que é tão importante - um pouco mais tarde. Em seguida, vemos os pentágonos azuis. Estas são moléculas de
desoxi ribose (como açúcar, um pouco menos de glicose. Difere da ribose pela ausência de um grupo OH, que dá estabilidade à molécula de DNA, ao contrário do RNA, no qual a ribose é usada. Em seguida, por simplicidade, omitirei o prefixo de desoxi e direi apenas ribose, sim camaradas escrupulosos nos perdoarão). Pequenos círculos - os restos de ácido fosfórico. Bem, na verdade existem bases nitrogenadas. Existem cinco deles, mas eles são encontrados principalmente no DNA 4. São Adenina, Guanina, Timin e Citosina. Ou seja, existe uma ribose à qual uma base nitrogenada está associada. Juntos, eles formam os chamados nucleosídeos, que se ligam usando resíduos de ácido fosfórico. Assim, obtemos uma longa cadeia composta por monômeros. Agora olhe para a cadeia esquerda ampliada. Veja C e G conectados por três linhas tracejadas e T e A por dois. O que isso significa? Sim, o DNA tem duas vertentes, mas o que as mantém unidas? Existe uma ligação de hidrogênio. Parece algo assim. Uma carga negativa parcial é formada nos átomos de oxigênio (O) e nitrogênio (N) e uma carga positiva no hidrogênio (H). Isso leva à formação de ligações fracas.

As conexões são realmente muito fracas. Sua energia pode ser 200 vezes menor que a energia das ligações covalentes (elas são formadas devido à sobreposição de um par de nuvens de elétrons, por exemplo, uma ligação em uma molécula de CO2). No entanto, existem muitos desses laços. Em cada uma de nossas células, as cadeias de DNA estão ligadas por quase 16 bilhões de ligações fracas, não um pouco, concorda?
Mas voltando ao número de conexões entre as bases. A citosina e a guanina são conectadas por três ligações, e a adenina e a timin são duas. Isso leva ao fato de que G e C estão conectados muito mais fortemente que A e T. Alguns organismos precisam de estabilidade especial das ligações de DNA, por exemplo, vivendo a altas temperaturas. Quando aquecido, o DNA contendo mais pares de HC é mais estável. Então você quer viver em um gêiser - tenha muitos pares de HZ. Embora estudos recentes digam que não há uma conexão clara entre a composição do GC (% de pares de HC de todos os pares) e a temperatura do habitat. Vale dizer que ele varia muito. Portanto, no Candidatus Carsonella ruddii PV (endossimbionte intracelular), é de cerca de 16%, temos quase 41%, e no Anaeromyxobacter K (uma bactéria de tamanho médio) atinge 75%.
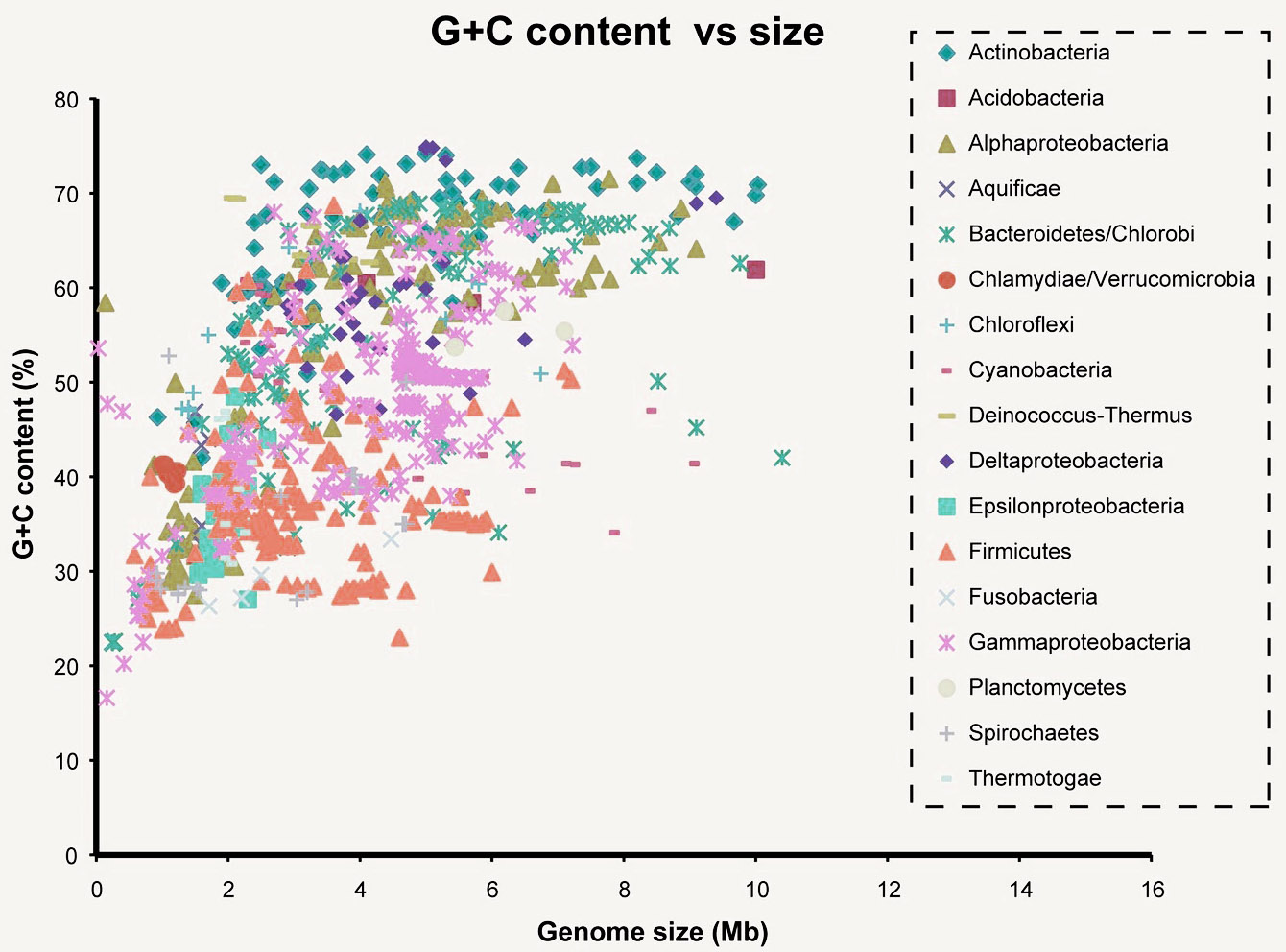
Aqui você pode ver a relação da composição do GC com o tamanho do genoma bacteriano. Mb é um milhão de pares de nucleotídeos. O indicador é bastante variável. A propósito, é frequentemente usado como um recurso ao ensinar vários tipos de classificadores. Recentemente, eu mesmo escrevi um classificador para reconhecer patógenos com base em dados brutos de seqüenciamento, e constatou-se que a composição do GC pode ser usada mesmo para um tipo.
Ainda não esqueci. Por que é importante que o DNA seja de fita dupla? Com base em uma cadeia, você pode restaurar outra. Se uma peça oposta à sequência Adenina-Adenina-Citosina é danificada em uma cadeia, sabemos com certeza que antes do dano havia Timin-Timin-Guanin. Assim, a presença do segundo circuito permite um armazenamento mais confiável de informações.
Legal! Agora, de volta à própria molécula de DNA. Esta é uma cadeia de 4 tipos de links. No entanto, quanto tempo? O Candidatus Carsonella ruddii PV possui apenas 160.000 nucleotídeos, como mencionado acima. Você e eu temos 3,2 bilhões (em uma célula haplóide, ou seja, com um conjunto de cromossomos. A maioria de nossas células possui dois). Parece muito, certo? Na verdade não. Em uma ameba unicelular (Amoeba dubia), possui cerca de 670 bilhões de pares de nucleotídeos. Parece ser uma cadeia infinitamente longa, então vamos traduzir o tamanho em nossos medidores favoritos. Se todos os nossos cromossomos (46 deles, não se esqueça; 23 duas cópias cada) são expandidos e esticados em uma linha, temos uma corrente de 2 metros. O DNA de uma ameba é suficiente para cercar um estádio de futebol. Mas o que eu estou levando? O núcleo em que o DNA é armazenado não é muito grande. Em média, temos um diâmetro de 6 mícrons. Não muito, se você deseja enrolar uma linha de 2 metros, embora muito fina. E você não precisa apenas empurrar a linha para dentro do núcleo. É necessário entrar em colapso para que a qualquer momento seja possível fornecer acesso a qualquer parte dele. A tarefa é difícil. E proteínas especializadas conseguem lidar com isso com sucesso. Eles criam uma série de espirais e loops que fornecem níveis cada vez mais altos de embalagem e não permitem que o DNA seja emaranhado no nó górdio. Vamos falar sobre como é empacotado.
Devo dizer que é empacotado de maneiras muito diferentes. Mas se você descartar o exótico, há duas maneiras. O primeiro é característico das bactérias, o segundo dos eucariotos (ou de outra forma nuclear).
Embalagem de DNA em bactérias
Vamos começar com nossos irmãos menores. As próprias bactérias não têm um genoma muito grande, em média de 1 a 5 milhões de pares de nucleotídeos. A diferença mais característica entre eles e nós é que eles não têm um núcleo e o DNA flutua na célula. Não sabe nadar, está parcialmente preso à membrana celular e também é dobrado, mas não tanto quanto o nosso.
O segundo O DNA bacteriano geralmente é circular. Portanto, é mais fácil copiar (não há fins que podem ser perdidos ao copiar e você não precisa criar mecanismos para salvar os fins). Normalmente, esse anel é um deles, mas algumas bactérias podem ter 2 ou 3. Existem ainda menos anéis (de alguns milhares a centenas de milhares de resíduos), com um nome de plasmídeo, e essa é outra história.
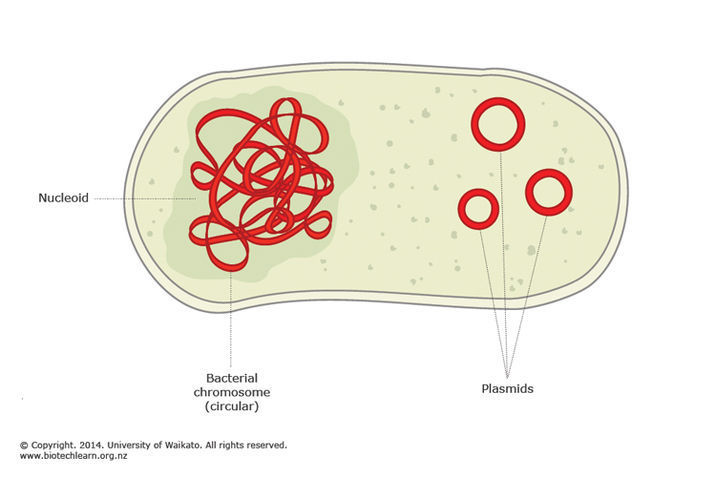
De volta ao pacote de DNA. O DNA é repleto de proteínas histonas (também existem proteínas semelhantes a histonas). DNA é ácido desoxirribonucléico.
Acid . Isso significa que é carregado negativamente (devido a resíduos de ácido fosfórico). Portanto, as proteínas que o ligam são carregadas positivamente. Dessa forma, eles podem se ligar ao DNA. O DNA bacteriano, juntamente com suas proteínas de embalagem, formam um nucleoide, com 80% de sua massa sendo DNA. Parece algo assim. Ou seja, o DNA do anel é dividido em domínios de 40 mil pares de nucleotídeos. Depois, há uma torção. A torção também ocorre dentro dos domínios, mas seu grau difere em domínios diferentes. Em média, o grau de empacotamento do DNA bacteriano varia de cem a mil vezes.
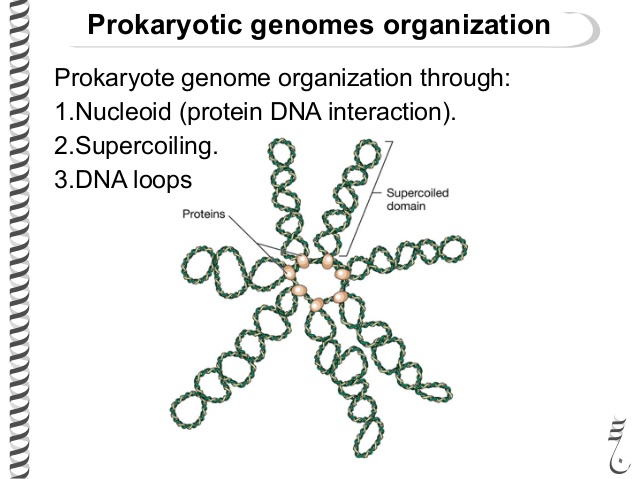
Ainda há um
vídeo legal .
Embalagem de DNA em eucariotos
Tudo é muito mais interessante aqui. Nosso DNA está bem compactado e escondido dentro do núcleo. E é muito mais eficiente que as bactérias. Durante a mitose (divisão celular), o tamanho do cromossomo 22 é de 2 mícrons. Se for desembaraçado e puxado, já terá 1,5 cm, o que corresponde ao grau de embalagem em 10.000 vezes. Trata-se do grau máximo de embalagem do nosso DNA. Durante a divisão, você precisa empacotar o DNA o máximo possível para poder dividi-lo efetivamente entre as células filhas. Na vida cotidiana, o grau de compactação é de aproximadamente 500 vezes. É difícil ler muito DNA.
Existem vários níveis de embalagem de DNA eucariótico
O primeiro é o nível do nucleossomo. As proteínas de 8 histonas formam uma partícula na qual o DNA é enrolado. Então outra proteína o corrige. Parece algo assim.
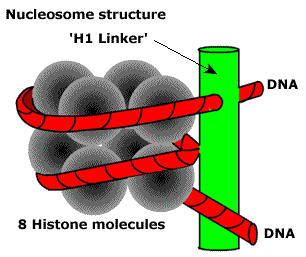

Acontece um tipo de contas. Devido a isso, a densidade da gaxeta aumenta de 7 a 10 vezes. Em seguida, os nucleossomos são compactados em fibrilas. Um pouco como um picles. Aqui, o grau total de embalagem pode chegar a 60 vezes.

O próximo estágio da compactação de DNA está associado à formação de estruturas semelhantes a alças chamadas cromômeros. A fibrila é dividida em seções de 10 - 80 mil pares de bases nitrogenadas. Nos locais de colapso existem glóbulos de proteínas não-histonas. As proteínas de ligação ao DNA reconhecem glóbulos de proteínas não-histonas e as juntam. A boca do laço é formada. O comprimento médio do loop inclui aproximadamente 50 mil bases. Essa estrutura é chamada cromoneme em interfase. E é nele que nosso DNA se localiza a maior parte do tempo. O nível de embalagem aqui atinge 500-1500 vezes.
Se necessário, a célula pode compactar ainda mais o material genético. A formação de alças maiores de fibrilas cromoméricas. Esses loops, por sua vez, formam novos loops (loops em loops ... e isso não é tricô). Que finalmente formam um cromossomo.
Em geral, o processo de embalagem pode ser descrito da seguinte maneira.

Como resultado, a partir de filamentos de DNA, obtemos, mediante divisão, estruturas superenroladas que podem ser vistas sob um microscópio. Nós os chamamos de cromossomos.
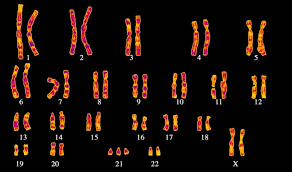
A substância dos próprios cromossomos é chamada cromatina. E o grau de sua embalagem varia de acordo com o local do cromossomo. Há eucromatina e heterocromatina. A euucromatina é uma região bastante limpa da cromatina, nela o DNA está no nível cromomérico (embalagem de 500 a 1000 vezes). Aqui está uma leitura ativa da informação. Por exemplo, se uma célula está sintetizando ativamente a proteína A agora, a região do DNA que a codifica estará em um estado de eucromatina, para que as enzimas que lêem o DNA possam alcançá-la. A heterocromatina contém a parte do DNA que a célula realmente não precisa agora. Ou seja, o DNA é compactado o mais firmemente possível para não ficar sob os seus pés. Dependendo das necessidades da célula, algumas regiões da cromatina podem se desfazer parcialmente, enquanto outras podem se entrelaçar. Assim, a regulação também é realizada (uma aproximação muito aproximada), porque não se pode alcançar uma região distorcida e, portanto, não pode ser lido.
Na verdade, é tudo por agora. Discutimos como o meio de armazenamento é armazenado. Vamos fazer uma pequena pausa e em alguns dias falaremos sobre a codificação de informações.