
Os cientistas realizaram uma festa no jogo da velha usando DNA. Parece engraçado, mas esta é apenas a ponta do iceberg, abalada por um toque de humor dos próprios cientistas. De fato, o estudo de hoje revela novos métodos para monitorar a cinética da interação de nanoestruturas de DNA, que juntas podem executar tarefas complexas. Usar o DNA como partes principais do sistema não é uma tarefa fácil, mas isso não impede os cientistas cujas mentes estão cheias de idéias, mas com corações de entusiasmo. E então, como eles conseguiram mover os blocos de DNA de acordo com um determinado padrão, como mostrou o estudo, e quais são as perspectivas para essa técnica no futuro? Para obter respostas, recorremos ao relatório do grupo de pesquisa. Vamos lá
Base de estudoNeste estudo, os cientistas demonstraram uma nova técnica para mover as peças de DNA, que se baseia nos princípios de junções de suporte e compensações de ramos. Esses princípios são semelhantes ao deslocamento padrão das cadeias de DNA, mas são direcionados a objetos maiores, mais precisamente às estruturas de DNA.
Essa técnica tornou possível ganhar controle sobre a cinética das telhas de DNA e realizar a cadeia de reações desejada dentro de sistemas de múltiplas estruturas. O jogo do jogo da velha não foi realizado por diversão, mas para demonstrar a possibilidade de mudar o DNA do ladrilho em qualquer ordem e posição.
Para entender os mecanismos dos sistemas biológicos, é necessário considerar seus principais componentes, dentre os quais as estruturas moleculares são uma das mais importantes. O DNA, como molécula transportadora, é considerado um dos melhores materiais para o estudo e criação de nanoestruturas auto-organizadas.
Entre as técnicas baseadas no DNA, a técnica do DNA-origami vem ganhando cada vez mais popularidade nos últimos anos, o que permite criar estruturas a partir do DNA de qualquer forma e configuração desejada.
Os componentes de um único origami de DNA podem ser torcidos e torcidos como você quiser, mas até agora não foi possível alcançar uma interação controlada entre vários origami de DNA como componentes de uma estrutura maior e, portanto, mais complexa. No momento, só era possível conectar / desconectar vários origami de DNA.
No estudo de hoje, os cientistas demonstraram uma nova técnica de interação com origami de DNA. Existe uma estrutura na qual o mosaico de DNA-origami é incorporado, deslocando outro mosaico da matriz geral e conectando-se à estrutura devido ao
domínio de ligação * na borda do mosaico (ponto de apoio / conexão).
Domínio de ligação / ligação * - um domínio proteico que faz parte da cadeia proteica, mas é capaz de funcionar separadamente.
Como exemplo, os cientistas criaram três opções de reconfiguração: competitiva, seqüencial e cooperativa. A totalidade desses "movimentos" (reconfiguração passo a passo) foi demonstrada como um jogo da velha, quando cada jogador tinha 9 origens de DNA exclusivos que podem ser movidos em qualquer ordem em um campo de 264x264 nm.
Formação de DNA Tile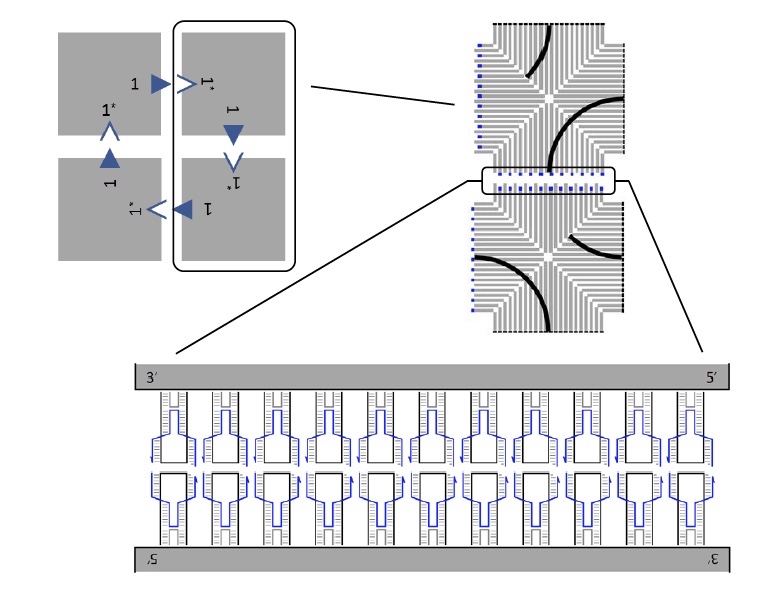 Estruturas de ladrilhos de DNA.
Estruturas de ladrilhos de DNA.Anteriormente, antes de conduzir este estudo, os cientistas desenvolveram uma técnica para criar blocos de DNA individuais. Cada bloco em duas arestas possui 11 grampos, cada um dos quais participa do empilhamento.
A imagem acima mostra uma matriz de 4 blocos (canto superior esquerdo), cada um dos quais é conectado ao adjacente através de colchetes.
Os pesquisadores consideraram várias opções possíveis para criar matrizes maiores com um grande número de peças. Para isso, em teoria, era necessário que a conexão entre os ladrilhos fosse fraca o suficiente. Isso permitiria que ela quebrasse e reconstruísse a matriz. No entanto, as observações mostraram que é possível criar matrizes de vários mosaicos reconfiguráveis, mesmo em temperaturas em que a conexão entre os mosaicos deve ser permanente.
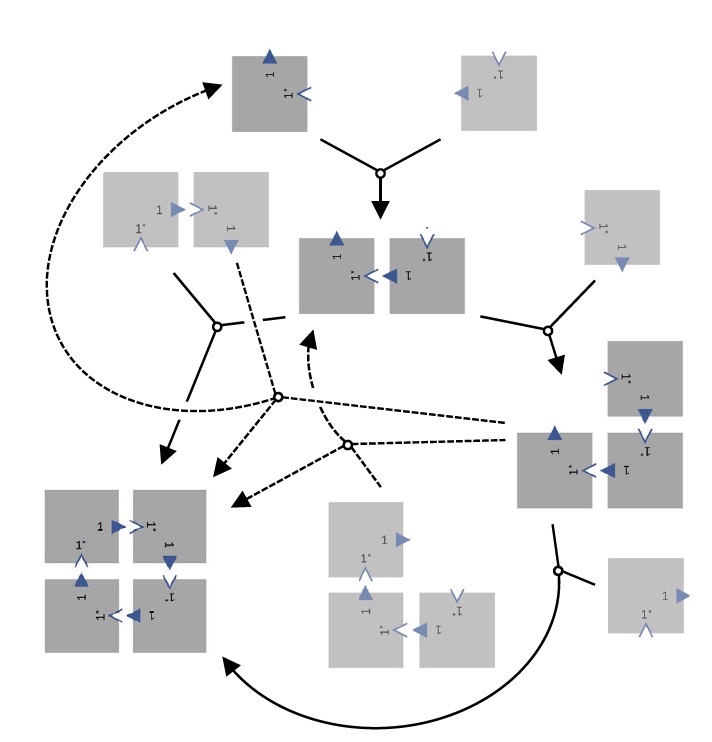 Reconfiguração de ladrilhos.
Reconfiguração de ladrilhos.Este fenômeno tem duas explicações possíveis. Primeiro, a formação da ligação ocorre a uma temperatura suficientemente alta, enquanto cai na faixa de temperatura da reversibilidade do processo. O segundo - o dímero e o trímero passam por uma reação de deslocamento; portanto, um arranjo 2x2 é formado quando o monômero é liberado. Também podem ser feitas 2 cópias dos trimers, formando uma matriz 2x2 liberando um dímero.
Se durante as reações de deslocamento não houver desconexão espontânea dos ladrilhos, não haverá armadilhas cinéticas, pelas quais os ladrilhos serão coletados em uma configuração 2x2.
Era necessário verificar se um origami de DNA deslocaria outro na estrutura geral, sem se ligar espontaneamente à própria estrutura. Em outras palavras, se o origami do DNA mostrará iniciativa ou não. Para isso, foram realizados dois experimentos.
 Experiência prática: 2 blocos de DNA (quadrados) + 1 triângulo de DNA, que devem formar uma estrutura comum.
Experiência prática: 2 blocos de DNA (quadrados) + 1 triângulo de DNA, que devem formar uma estrutura comum.No primeiro experimento, foram envolvidos 2 blocos de DNA e 1 triângulo de DNA; o experimento em si foi realizado a temperatura constante. Estruturalmente, um triângulo pode ter o mesmo domínio de conexão que os quadrados ou um domínio de conexão adicional complementando o quadrado adjacente.
Na primeira versão do experimento, dois quadrados permanecem conectados um ao outro, enquanto o triângulo se conecta a um dos quadrados. Na segunda versão, um triângulo substituiu um dos quadrados, deslocando-o, por assim dizer. Na prática, isso mostrou que é possível modificar origens complexos de DNA. Mas agora precisamos garantir que esse processo seja controlado pelos cientistas e que não ocorra por conta própria devido a forças naturais.
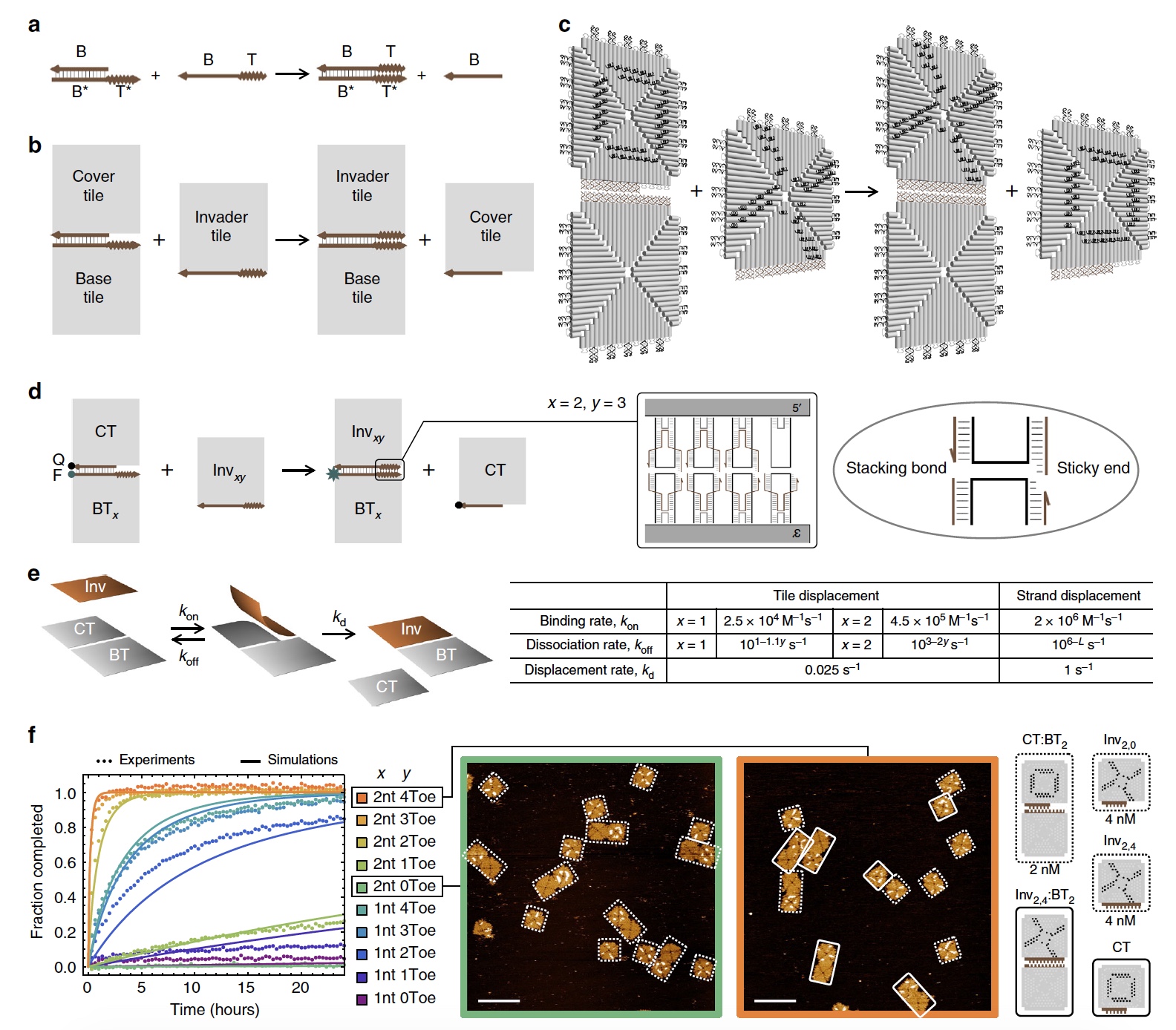 Imagem Nº 1
Imagem Nº 1Se falamos de reações de deslocamento nas cadeias de DNA, então aqui temos uma cadeia com um domínio de ligação que se conecta a um domínio complementar "livre". Portanto, obtemos uma estrutura de fita dupla na qual o processo de migração de ramificações ocorre quando as cadeias se movem através do ponto de conexão (
1a ). Um processo semelhante ocorre nas reações de deslocamento de DNA dos ladrilhos (
1b e
1c ).
A partir da imagem
1c, também podemos ver que cada bloco de DNA consiste em 4 triângulos isósceles conectados por aparelhos, dos quais já conhecemos.
Em relação ao jogo do jogo da velha: para uma visualização mais conveniente, o processo de deslocamento e reconfiguração dos participantes foi marcado com uma cruz (X) e um dedo do pé (O) por meio de colchetes em duas cores diferentes. Veja mais de perto a imagem nº 1, X (preto) e O (branco) são visíveis nos ladrilhos.
Cinética das reações de deslocamento do DNA da telhaEm palavras simples, obter controle sobre a cinética de um processo precisa tornar algumas reações mais rápidas e outras mais lentas. Dessa maneira, é possível unir, desconectar e reconfigurar os blocos. Mas você precisa determinar a faixa de cinética que permite que isso seja feito. Para fazer isso, os cientistas criaram pontos de conexão (entre ladrilhos) com diferentes forças, por assim dizer, de acoplamento.
O número de grampos no conector no domínio de migração da ramificação foi o mesmo para todos os blocos. Mas aqui o domínio do domínio dos pontos de referência (empate) variou de 0 a 4 (
1d ).
O experimento envolveu o bloco principal (todos os 4 pontos de comunicação estão "ativos") e os blocos acessórios (o número de pontos de conexão variou de 0 a 4). No final do domínio de migração de ramificação, 2 pares de grampos foram modificados com fluoróforo e extintor. Se a placa acessória permanecer em contato com a placa principal, o fluoróforo será extinto, o que levará a um sinal fluorescente baixo. Mas se a placa acessória (com o amortecedor) for desconectada, o sinal fluorescente será amplificado. Assim, será possível determinar como o processo prossegue.
Um dia depois, foi possível tirar as primeiras conclusões das observações. Os cientistas observam que as trajetórias fluorescentes das peças acessórias com 0 suportes de conexão permaneceram praticamente inalteradas. Também foi observado que ladrilhos com o mesmo comprimento da nervura de conexão, mas com um grande número de pontos de conexão, exibem cinética muito mais rápida. É curioso que os pontos de conexão com os grampos com 1 nucleotídeo e 2 nucleotídeos mostrem taxas muito diferentes de saturação (saturação).
A imagem
1e mostra o modelo matemático usado para a estimativa numérica da cinética. Os próprios cientistas dizem que o modelo é muito simples e é frequentemente usado para avaliar a cinética das reações de deslocamento da cadeia de DNA e, portanto, também é adequado para peças de DNA.
Uma análise comparativa de simulações (
1f ) (reações simuladas) e experimentos possibilitou determinar os parâmetros que afetam o processo de recombinação de placas de DNA.
Em primeiro lugar, os cientistas observam que a velocidade de união das peças é de 10 a 100 vezes (dependendo do comprimento da borda da peça de união) menor que a velocidade de união das fitas de DNA. Uma coisa comum entre os processos de deslocamento de ladrilhos e fios é que, com um aumento no número de nucleotídeos nos pontos de conexão, a taxa de decaimento diminui exponencialmente. Além disso, essa velocidade é 40 vezes menor para os ladrilhos do que para os filamentos de DNA. O deslocamento total máximo para placas de DNA (taxa de reação) foi de 4,5x10
5 M
-1 * s
-1 . Se a concentração for baixa (<50 nM), a taxa de ligação bimolecular limitará a taxa de deslocamento dos ladrilhos. Se a concentração for superior a 50 nanomoles, a taxa de deslocamento monomolecular limitará a taxa de deslocamento dos ladrilhos.
Além disso, os cientistas realizaram várias experiências descrevendo três tipos principais de reconfiguração: competitiva, seqüencial e cooperativa. E agora vamos ver os resultados dos experimentos para cada um deles com mais detalhes.
Reconfiguração competitivaPara alcançar a reconfiguração competitiva, os cientistas propuseram o uso de uma função sigmóide em resposta à concentração do sinal, obtendo assim um sistema de reconfiguração baseado em informações. Esta função é um elemento importante dos cálculos lógicos digitais dos processos de deslocamento das cadeias de DNA; portanto, também pode ser aplicada às peças de DNA.
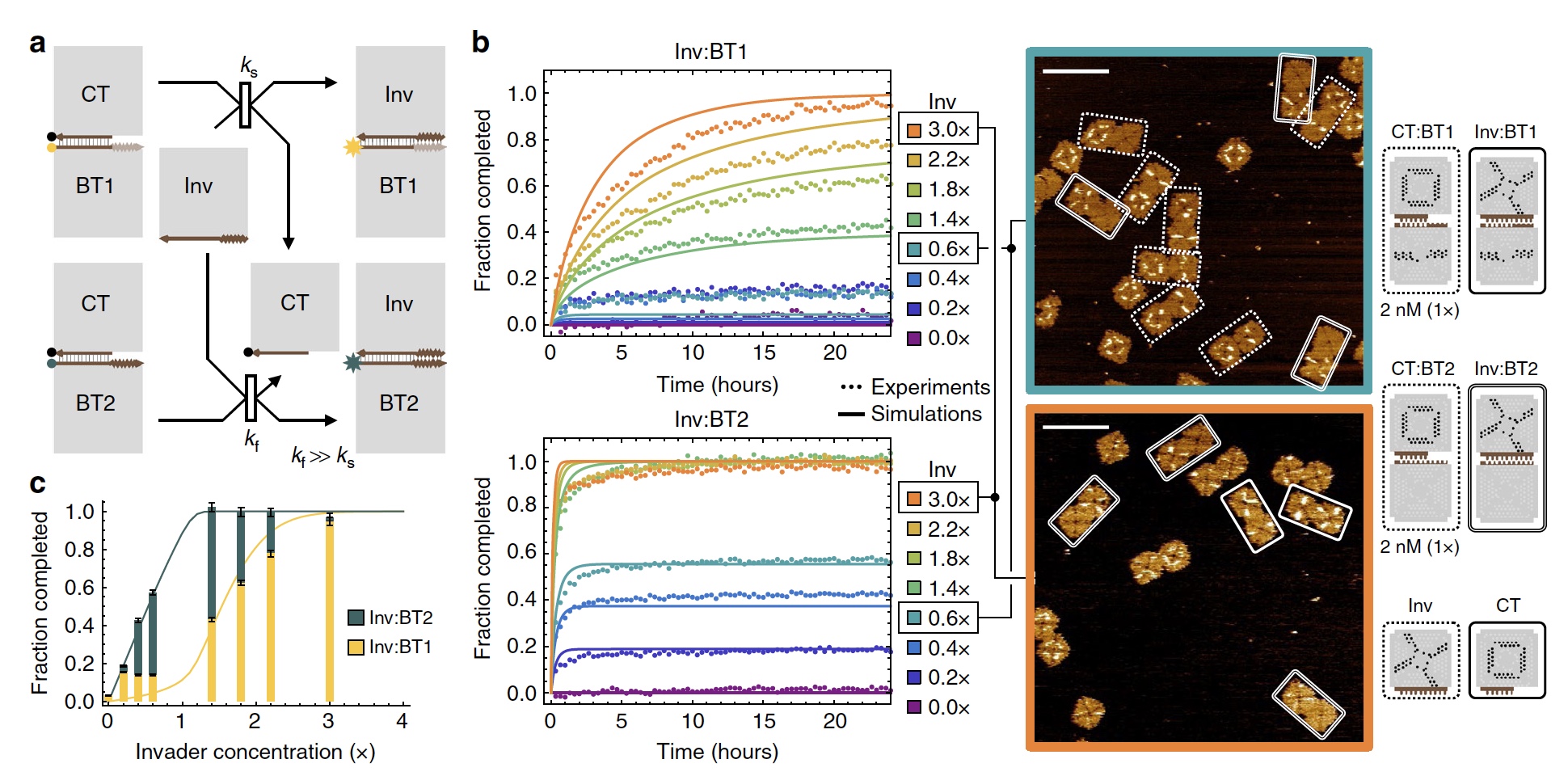 Imagem No. 2
Imagem No. 2Para demonstrar o "trabalho" da função sigmóide, os cientistas criaram 2 reações concorrentes de deslocamento de ladrilhos que são ativadas pelo mesmo ladrilho subordinado. A diferença em sua velocidade (
2a ).
Nos ladrilhos acessórios, o ponto de conexão é quatro grampos de 2 nucleotídeos. Espera-se que ele se conecte com um ponto de ligação adequado e com quatro grampos de 1 nucleotídeo. A taxa de reação dos ladrilhos acessórios difere em 18 vezes. Quando a concentração do ladrilho acessório foi menor que 2 nM, uma reação mais rápida (
2b ) foi desencadeada. Se a concentração fosse maior que 2 nM, uma reação mais lenta seria iniciada.
Após 24 horas, verificou-se que o resultado de uma reação mais rápida aumentou linearmente, e o resultado de uma reação mais lenta mostrou uma função sigmóide (
2c ).
Reconfiguração sequencialApós a reconfiguração competitiva foi consistente. Esse tipo é muito mais complicado, mas oferece mais controle sobre o processo, a menos que tudo dê certo.
Assim, os cientistas criaram uma matriz 2x2 na qual o primeiro sub-bloco desloca outro bloco, liberando o ponto de conexão usado anteriormente. Em seguida, a segunda estrutura subordinada de 2 peças desloca 2 peças da matriz. Uma espécie de deslocamento em cascata (
3a ).
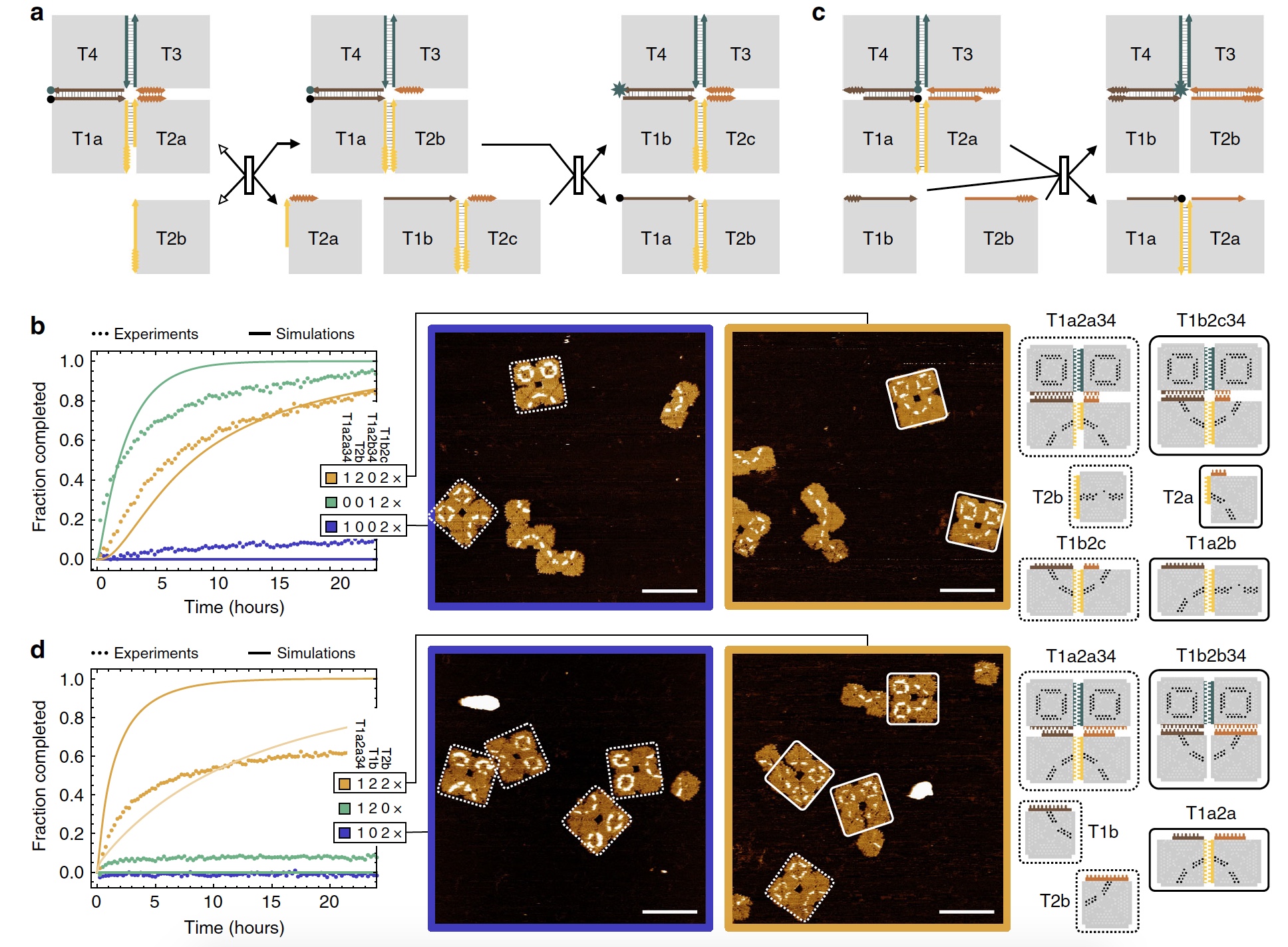 Imagem No. 3
Imagem No. 3Os cientistas notaram que o deslocamento nos cantos dos ladrilhos é lento, cerca de 100 vezes mais lento que no restante da borda do ladrilho. Portanto, esses estados intermediários de deslocamento devem ser levados em consideração para procedimentos de reconfiguração mais complexos.
Na imagem
3b , podemos ver resultados bem-sucedidos do experimento com reconfiguração sequencial (curva laranja).
E agora há um pouco mais de criatividade dos pesquisadores. Preste atenção nas imagens acima (
3b ): matrizes que reagiram ao primeiro bloco subordinado, mas não ao segundo, como resultado, parecendo uma “cara triste”. Mas as matrizes, nas quais a conexão com os dois blocos subordinados foi bem-sucedida, parecem um "rosto sorridente". E você diz que os cientistas não têm senso de humor.
Segundo os cientistas, quanto mais complexa a estrutura dos participantes da reação, mais complicado é o processo de reconfiguração, mas mais interessante é o resultado. O uso de dois tipos de participantes (um arranjo 2x2 e uma estrutura de dois ladrilhos) mostrou os seguintes resultados sobre a eficiência do deslocamento após 48 horas de observação: 83,3 ± 9,8% e 90,5 ± 6,1%. Em outras palavras, as reações de deslocamento são excelentes. Existem alguns erros, mas eles são insignificantes.
Os experimentos com reconfiguração seqüencial permitiram não apenas compreender os princípios básicos da sequência de pontos de comunicação e a "cascata" da reação, mas também demonstraram novos recursos. O sistema é capaz de responder a mais de 1 sinal indicando quais instruções requerem execução e o que está disponível para isso. Este sinal é apresentado no experimento na forma de dois tipos de estruturas subordinadas. Mesmo que a primeira estrutura subordinada entre em contato com a base anterior à segunda, a estrutura ainda mostra a reconfiguração esperada.
Esses processos, na maioria das vezes, prosseguem sozinhos. Os cientistas os controlam apenas levemente, mas não intervêm completamente. O próximo passo nos experimentos é aumentar o controle sobre o processo de reconfiguração e estudar a possibilidade de programar esse processo.
Reconfiguração cooperativaNesta experiência, havia 2 peças acessórias conectadas às duas arestas da matriz 2x2 (
3s ). Se apenas um bloco estiver presente, ele deverá se mover para o centro da matriz. Além disso, a ausência de um segundo bloco permite reverter o processo, desconectando repetidamente o bloco acessório da matriz. Na presença de ambos os blocos subordinados, dois processos de migração de ramificações surgem imediatamente, que convergem no centro da matriz e, como resultado, obtemos um deslocamento cooperativo.
A experiência com fluorescência mostrou que, com apenas um bloco subordinado, o sinal de fluorescência é muito fraco, mas amplificado se ambos os blocos estiverem presentes (
3d : curva azul e verde - sinal fraco, amarelo - sinal forte).
Como no experimento anterior, as matrizes que interagem apenas com 1 bloco subordinado foram marcadas como "rosto triste" e as matrizes e os dois blocos eram "rosto sorridente". Além disso, este último se formou em 68,0 ± 7,7% dos casos.
Tic Tac ToeE, finalmente, o experimento "jogo da velha". Nesta experiência, foi utilizada uma matriz de 3x3, que permite 9 reações únicas de deslocamento de ladrilhos em qualquer ordem (
4a ).
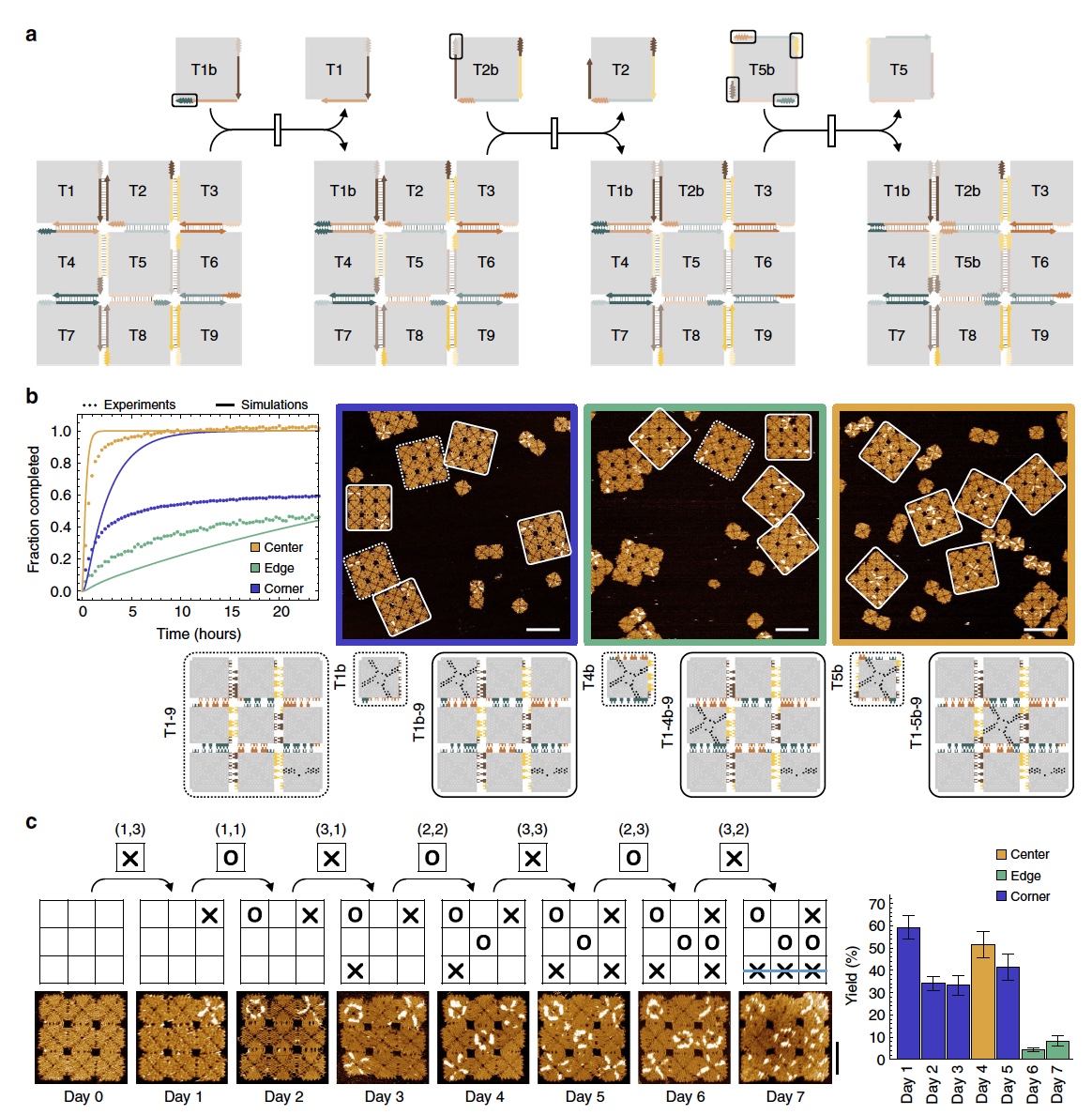 Imagem No. 4
Imagem No. 4Este experimento é uma combinação de vários tipos de reações ao mesmo tempo: o deslocamento do ladrilho de canto, o deslocamento do ladrilho de borda e o deslocamento do central. Ao longo do perímetro da matriz entre as peças adjacentes, havia um ponto de conexão, um total de 8 desses pontos. 4 deles participaram do deslocamento do ladrilho de canto, quando o ladrilho acessório com o ponto de conexão correspondente está em contato com o ladrilho de borda próximo ao canto. Nesse momento, a ramificação migra para dentro da borda do ladrilho e por um ângulo de 90 °, e o ladrilho de canto anteriormente conectado à matriz é liberado e integrado à própria matriz (dentro da matriz quando estava anteriormente na borda). Os outros 4 pontos de ligação foram usados para ativar o deslocamento dos ladrilhos de borda.
A coisa mais difícil neste experimento foi iniciar um deslocamento do ladrilho central, devido ao número limitado de pontos de comunicação disponíveis. Portanto, foram adicionados 4 pontos adicionais entre o ladrilho central e todos os ladrilhos vizinhos (
4a ).
As observações mostraram excelentes resultados. Todos os sub-mosaicos foram incorporados na matriz, que foi reconstruída como os cientistas haviam planejado. A porcentagem de conversão dentro da matriz foi a seguinte: 78,4 ± 6,0% para o deslocamento dos ladrilhos de canto, 52,8 ± 6,0% para o deslocamento dos ladrilhos de borda e 100% dos centrais.
E agora nos voltamos diretamente para o próprio jogo, que combina todos os tipos de reações na forma de uma cascata. O tabuleiro de jogo era uma matriz de 3x3, todas as peças estavam "limpas", ou seja, não estavam marcadas de forma alguma.
Cada um dos jogadores, dos quais havia 2 naturalmente, recebeu à sua disposição 9 peças subordinadas marcadas com “X” e “O”. Fazer uma jogada neste jogo está adicionando esse bloco à câmara de teste com um intervalo de 24 horas (um longo lote de jogo da velha, não é?).
Demonstração em vídeo de como jogar tic-tac-toe usando blocos de DNA.As observações (
4b e
4c ) mostraram que o campo de jogo reagiu a todos os movimentos, ou seja, a reconfiguração ocorreu de acordo com as instruções dos jogadores (adição de peças marcadas).
O problema todo era que, ao final do jogo em campo, havia muitas peças acessórias, respectivamente, a probabilidade de seu deslocamento espontâneo aumentou. Assim, a precisão da reconfiguração também foi reduzida. No final do jogo, esse indicador era de apenas 8,3 ± 2,3%. No entanto, o resultado ainda foi alcançado, como podemos ver nas imagens acima.
Para um conhecimento mais detalhado das nuances de pesquisas e experimentos, recomendo fortemente que você analise o
relatório de cientistas e
materiais adicionais .
EpílogoO jogo das telhas de DNA do jogo da velha não foi realizado por diversão, mas como um método de demonstrar um estudo muito sério, revelando a capacidade de manipular os processos de deslocamento de elementos em nanoestruturas construídas a partir do DNA.
É claro que ainda há muito a ser polido: o número de peças acessórias que afetam a taxa final de reconfiguração, a taxa de reações, a precisão do deslocamento, a expansão de possíveis opções de deslocamento e, é claro, o aumento nas dimensões da estrutura de DNA reconfigurável.
, . , , , — , , .
, google «-», . :) ( , )
Obrigado por ficar conosco. Você gosta dos nossos artigos? Deseja ver materiais mais interessantes? Ajude-nos fazendo um pedido ou recomendando a seus amigos, um
desconto de 30% para os usuários da Habr em um análogo exclusivo de servidores básicos que inventamos para você: Toda a verdade sobre o VPS (KVM) E5-2650 v4 (6 núcleos) 10GB DDR4 240GB SSD 1Gbps da US $ 20 ou como dividir o servidor? (as opções estão disponíveis com RAID1 e RAID10, até 24 núcleos e até 40GB DDR4).
VPS (KVM) E5-2650 v4 (6 núcleos) 10 GB DDR4 240 GB SSD de 1 Gbps até a mola gratuitamente, ao pagar por um período de seis meses, você pode fazer o pedido aqui .Dell R730xd 2 vezes mais barato?Somente nós temos 2 TVs Intel Dodeca-Core Xeon E5-2650v4 128GB DDR4 6x480GB SSD 1Gbps 100 a partir de US $ 249 na Holanda e nos EUA! Leia sobre
Como criar um prédio de infraestrutura. classe usando servidores Dell R730xd E5-2650 v4 custando 9.000 euros por um centavo?