Em meados de 2018, foi
publicado o
trabalho sobre eletrofisiologia do cérebro de ratos, juntamente com o qual um
conjunto de dados exclusivo foi disponibilizado ao público. O conjunto de dados é único, pois contém gravações simultâneas do potencial de campo local usando um novo eletrodo de
neuropixels de alta densidade (sonda ou sonda) e um eletrodo de patch de uma célula localizada perto da amostra. O interesse em tais gravações não é apenas fundamental, mas também aplicado, pois permite validar modelos para a análise da atividade neuronal registrada por amostras modernas. E isso, por sua vez, está diretamente relacionado ao desenvolvimento de novas próteses neuro. Qual é a novidade fundamental e por que esse conjunto de dados é tão importante - eu vou lhe dizer em detalhes.
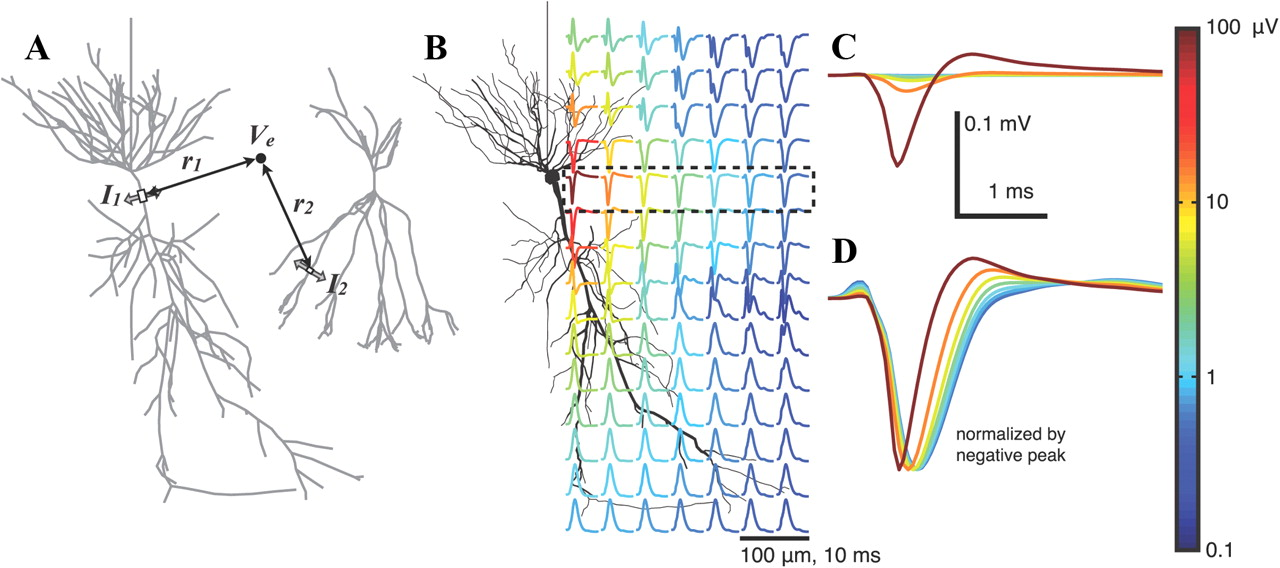 KDPV: resultado da modelagem do potencial extracelular próximo a um neurônio durante a geração do potencial de ação ( fonte ). A cor indica a amplitude do potencial. Esta ilustração será importante para uma melhor compreensão.Os métodos eletrofisiológicos para estudar o
KDPV: resultado da modelagem do potencial extracelular próximo a um neurônio durante a geração do potencial de ação ( fonte ). A cor indica a amplitude do potencial. Esta ilustração será importante para uma melhor compreensão.Os métodos eletrofisiológicos para estudar o cérebro são baseados no registro do potencial elétrico do cérebro. Eles podem ser divididos em não invasivos - principalmente eletroencefalografia (EEG) - e invasivos, por exemplo, eletrocorticografia (ECoG), patch-clamp (patch-clamp) ou registro do potencial de campo local (LPP = potencial de campo local, LFP) . Para este último, um pequeno eletrodo de 10 a 100 mícrons é injetado diretamente no cérebro e seu potencial é registrado. Para investigar a atividade do cérebro de mamíferos no nível celular, ou seja, para medir a atividade de células individuais, os métodos não invasivos disponíveis não podem ser aplicados, porque o potencial de uma célula decai no espaço muito rapidamente, literalmente por 100 μm (consulte KDPV). Portanto, em qualquer modelo animal, bem como em humanos, os métodos não invasivos fornecem informações apenas sobre a atividade coletiva dos neurônios e sobre o trabalho, provavelmente no nível do tecido, mas não nos neurônios individuais.
Mas com métodos invasivos não é tão simples. Para registrar a atividade de um neurônio, é necessário aproximar o eletrodo do neurônio, colocá-lo idealmente dentro da célula, como é feito no patch clamp, ou usar
eletrodos afiados , o que é difícil na prática, muito difícil. Por outro lado, qualquer eletrodo extracelular de aproximadamente 10 μm registrará potenciais de ação de 5 a 10 células, devido à alta densidade de neurônios e à alta condutividade iônica da solução extracelular. Portanto, a tarefa de registrar células individuais é tecnicamente resolvida aumentando a densidade de eletrodos localizados perto da célula. Nesse sentido, a eletrofisiologia moderna está avançando no sentido de aumentar a densidade dos eletrodos, aumentar o número e diminuir o tamanho. Entre os requisitos, há a necessidade de amplificar o sinal mais perto do local de registro para reduzir o ruído e colocar um multiplexador para reduzir o tamanho. Assim, em 2016, foi anunciado em uma pré-impressão, e em 2017 foi publicado na Nature, e em 2018 já apareceu
no mercado , uma nova amostra de neuropixels de alta densidade feita pela tecnologia CMOS, em 960 eletrodos, dos quais 384 estão disponíveis para gravação simultânea. O tamanho de um site de registro é de 12 mícrons. A espessura da amostra é de 24 microns. Além disso, com eletrodos de alta densidade, bem como com amplificação ativa, as pessoas começaram a trabalhar por um longo tempo, mas a Neuropixels foi a primeira a atingir produção e vendas, portanto, em um futuro próximo, esse teste específico em artigos será cada vez mais encontrado.
 Fig. Esquema de neuropixels. Em um substrato de silício monolítico, existem 960 locais, além de um multiplexador completo e uma interface AD para 384 canais.
Fig. Esquema de neuropixels. Em um substrato de silício monolítico, existem 960 locais, além de um multiplexador completo e uma interface AD para 384 canais.Estrutura de dados
Além dos
ritmos clássicos de
atividade (alfa, beta, gama, etc.) responsáveis pela sincronização do grupo, os dados obtidos com essas amostras também contêm os potenciais de ação das células individuais (PA = potenciais de ação, PA, espigões, aderências) , que no registro parecem picos curtos de aproximadamente 1 ms de duração.
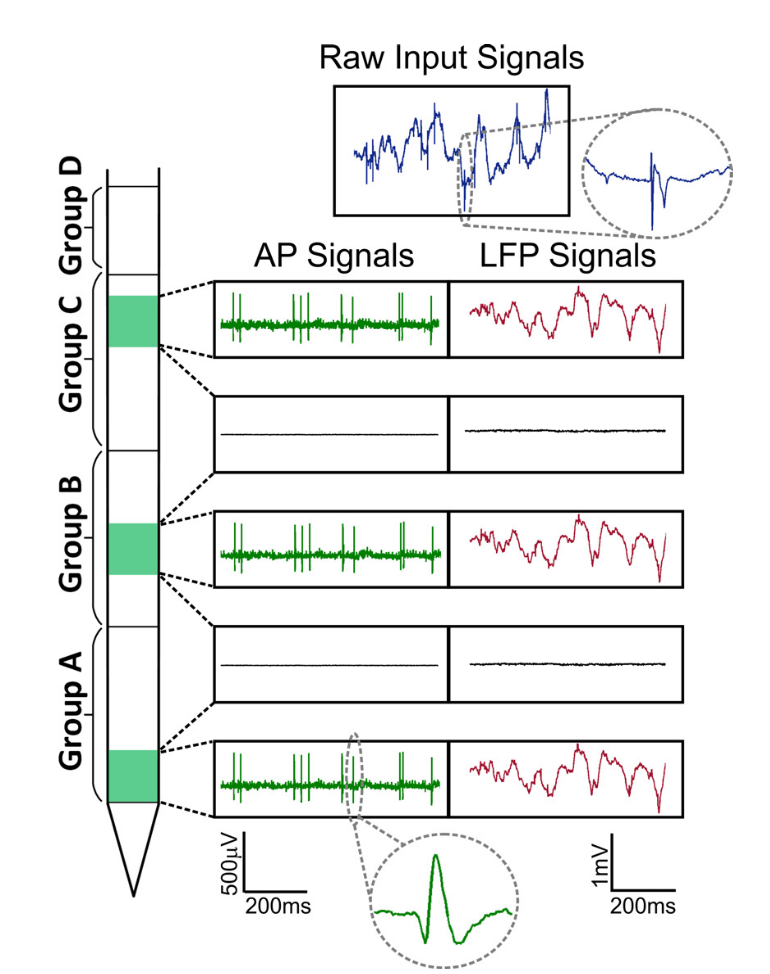 Fig. Sinais Neuropixels. Duas partes do sinal são distinguidas: potencial de campo local (LFP, até ~ 300 Hz) e atividade celular (AP, de 300 Hz).
Fig. Sinais Neuropixels. Duas partes do sinal são distinguidas: potencial de campo local (LFP, até ~ 300 Hz) e atividade celular (AP, de 300 Hz).Além disso, se o potencial de campo local de baixa frequência é geralmente analisado dentro da estrutura de oscilações e a análise espectral ou de onda é usada como no EEG, a atividade celular contém os potenciais de ação das células individuais, consiste em eventos discretos em um cenário de ruído. A tarefa de isolar a atividade de células individuais reduz formalmente a tarefa de um problema de coquetel, quando um orador separado deve ser diferenciado do conjunto de oradores. O big data aparece quando avaliamos o fluxo de dados de uma amostra. Para análise de picos, a amostragem é realizada em 30-40 kHz com digitalização de 16 bits por ponto (uint16), portanto, a gravação de 100 eletrodos em 1 segundo já pesará 8 MB. Ao mesmo tempo, os experimentos geralmente duram horas, o que equivale a centenas de gigabytes de apenas um dia útil e para um estudo completo, digamos, de dez desses registros. Portanto, o potencial desta amostra também depende fortemente de algoritmos de aprendizado de máquina, que são usados para análise de dados.
Aprendizado de Máquina e Atividade Celular
Normalmente, um pilar para análise da atividade celular consiste em pré-processamento, segmentação de pico e agrupamento. Essa parte da pesquisa é comumente referida como análise de cluster ou classificação de pico. Como pré-processamento, a filtragem passa-baixo (> 300 Hz) é normalmente usada, pois acredita-se que não há outros ritmos fisiológicos acima de 300 Hz, e apenas informações sobre a atividade celular individual permanecem. Além disso, durante o pré-processamento em amostras densas, é possível reduzir o ruído correlacionado, por exemplo, captadores a 50 Hz. A segmentação é geralmente tomada como um limiar simples; por exemplo, qualquer coisa acima de 5 desvios de ruído padrão pode ser considerado um evento. Ocorre que a segmentação de dois limiares é usada, com um limiar suave e rígido, para distinguir eventos relacionados no espaço e no tempo, como no algoritmo de
segmentação de bacias hidrográficas, apenas no clustering de cluster a distribuição de marcadores leva em consideração a topologia da amostra. Após a segmentação, uma janela com duração de 1-2 ms é tomada perto do centro de cada evento, e o sinal nessa janela, coletado de todos os canais, torna-se uma amostra para aglomeração adicional. Essa amostra é chamada de forma de onda de pico. Células diferentes e sua distância diferente do local de registro levam ao fato de que suas formas de onda variam (consulte KDPV). O próprio algoritmo de agrupamento de formas de onda usa EM, pesquisa de correspondência de modelos, aprendizado profundo e muitas variações (
tópico no github ). O único requisito é treinar sem um professor. Mas há um problema. Ninguém sabe ao certo quais parâmetros você precisa adotar para seu pipeline para que a análise seja a mais eficaz. Normalmente, após o clustering, o analista analisa manualmente os resultados e faz alterações a critério. Assim, os resultados da análise podem incluir erros de algoritmo e erros humanos. E eles podem não ser, portanto, a questão da validação objetiva permanece em aberto.
Existem várias maneiras de validar um pipeline. Primeiro, alterando as condições externas para o objeto de estudo. Por exemplo, durante um experimento, se você estudar as partes visuais do córtex, poderá alterar a textura, a cor e o brilho da imagem. Se na análise houver uma célula que altere sua atividade dependendo do estímulo, você estará com sorte. Em segundo lugar, você pode aprimorar ou diminuir farmacologicamente a atividade de um tipo específico de célula, por exemplo, usando determinados bloqueadores de canais. Então, a atividade da sua célula aumentará / diminuirá e você verá a diferença no agrupamento. No entanto, essa modulação da atividade também levará a alterações nas formas de onda, porque o perfil do potencial de ação no tempo é completamente determinado pela cinética dos canais iônicos. Em terceiro lugar, você pode medir ou induzir a atividade de determinadas células de forma optogênica ou usando uma pipeta de correção, como neste conjunto de dados. Devido à grande relação sinal / ruído e à estabilidade do eletrodo, você estará totalmente confiante na atividade de uma única célula. Conceitualmente, a publicação foi dedicada à montagem de um conjunto de dados de validação usando um grampo de correção.
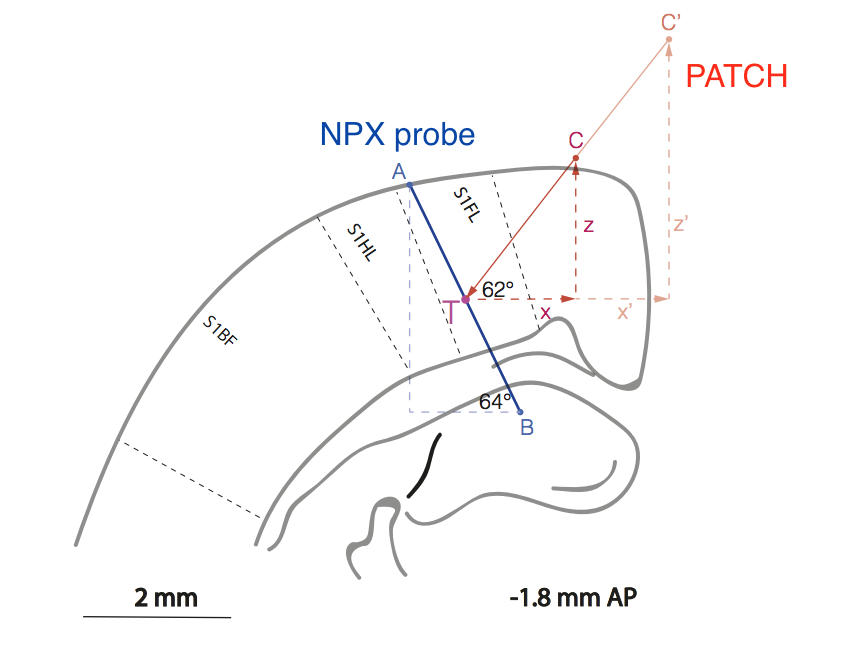 Fig. Representação esquemática da posição relativa da amostra (linha AB) e da pipeta de remendo (linha C'CT) na região do córtex de rato, responsável pelo processamento de informações sensoriais da pata dianteira (S1FL = córtex sensorial 1 do membro anterior.
Fig. Representação esquemática da posição relativa da amostra (linha AB) e da pipeta de remendo (linha C'CT) na região do córtex de rato, responsável pelo processamento de informações sensoriais da pata dianteira (S1FL = córtex sensorial 1 do membro anterior.Escusado será dizer que o trabalho metodológico é extremamente difícil, porque os pesquisadores tiveram que desenvolver um método para o posicionamento relativo de dois eletrodos no córtex cerebral sem controle visual com uma precisão de ~ 10 μm.
O efeito da densidade do eletrodo no aglomerado de espigões
Por que é tão importante aumentar a densidade dos sites de registro? Para uma analogia, consideramos o fato conhecido entre os pesquisadores de EEG que, a partir de um certo limiar, um aumento no número de eletrodos na tampa não leva a um aumento perceptível na informação recebida, ou seja, o sinal do eletrodo difere um pouco da interpolação linear de sinais dos eletrodos vizinhos. Alguém diz que esse limite já é atingido aos 30, alguém aos 50, alguém aos 100 eletrodos. Aqueles que trabalham com o EEG em detalhes podem ser corrigidos. Porém, no caso da atividade celular, o limiar para a densidade dos locais de registro em uma amostra ainda não é conhecido, portanto a corrida de amostras de alta densidade continua. Para isso, a equipe do Kampff Lab continua trabalhando com uma amostra em um site de 5x5 μm
2 , e
dados preliminares foram publicados. Os especialistas que trabalham com eletrodos densos compartilham sua experiência de que, inesperadamente, o número específico de células individuais que podem ser isoladas de amostras da mesma área é maior, onde a densidade dos locais de registro é maior. Esse efeito é bem ilustrado em
outro estudo dos mesmos co-autores, que selecionaram artificialmente apenas uma parte dos locais de uma amostra densa e avaliaram visualmente a qualidade dos clusters resultantes após a conversão de tSNE em valores de PCA devido a picos de formas de onda. Isso não é um cânone para cluster, mas é bom para ilustrar dependências. Como teste, o Neuroseeker atuou em 128 canais com um tamanho total de 700x70 μm
2 com um site de 20x20 μm
2 .
 Fig. Diagramas de TSNE sobre PCA em formas de onda brutas, reduzindo artificialmente a densidade dos locais na amostra. Os locais de trabalho são mostrados esquematicamente na parte superior de cada diagrama. É claramente mostrado como exatamente o número de clusters segregados cresce com o aumento da densidade do local, A é o melhor, F é o pior.
Fig. Diagramas de TSNE sobre PCA em formas de onda brutas, reduzindo artificialmente a densidade dos locais na amostra. Os locais de trabalho são mostrados esquematicamente na parte superior de cada diagrama. É claramente mostrado como exatamente o número de clusters segregados cresce com o aumento da densidade do local, A é o melhor, F é o pior.Qual é a essência do trabalho
Nos dados de Marques-Smith et al. há gravação simultânea do grampo de remendo e amostras. Usando dados do patchclamp, os cientistas encontraram momentos de potenciais de ação e usaram esses momentos para segmentar e calcular formas de onda já existentes na amostra. Como resultado, eles foram capazes de criar distribuições de alta qualidade do potencial de ação no tempo e no espaço em toda a área da amostra.
 Fig. À esquerda, os traços de atividade celular são mostrados simultaneamente no clipe de remendo (preto) e no canal Neuropixels mais próximo (azul). No meio - 500 amostras individuais e sua média. À direita está a distribuição do potencial de ação no espaço sobre a área da amostra e no tempo.
Fig. À esquerda, os traços de atividade celular são mostrados simultaneamente no clipe de remendo (preto) e no canal Neuropixels mais próximo (azul). No meio - 500 amostras individuais e sua média. À direita está a distribuição do potencial de ação no espaço sobre a área da amostra e no tempo.Em seguida, levanta-se a questão sobre a variação da forma de onda extracelular de pico para pico - sim, é palpável e deve ser levado em consideração. Depois, mostram que é fundamentalmente possível rastrear a distribuição do potencial de ação ao longo da membrana celular usando seus eletrodos densos, mas isso já foi demonstrado anteriormente no trabalho de outros grupos. Em conclusão, eles oferecem aos colaboradores em potencial algumas questões fundamentais da neurofisiologia, que você pode tentar responder com a ajuda do conjunto de dados e também oferece o uso do conjunto de dados para validar os pipelines para agrupar a atividade das células. O último parece um desafio ousado, porque agora existem muitos algoritmos de agrupamento e a concorrência entre os métodos é muito grande. Nem todos os métodos, em primeiro lugar, funcionam com um número tão grande de canais e, em segundo lugar, nem todos podem fornecer clustering objetivamente de alta qualidade.
O que vem a seguir
Em primeiro lugar, uma nova versão do
Neuroseeker em 1300 canais também usando tecnologias CMOS está se aproximando, dados preliminares
já estão disponíveis .
Em segundo lugar, estamos aguardando outro conjunto de dados, já do Allen Institute for Brain Science, anunciado na conferência FENS em 2018. Ele usará simultaneamente 4 (!) Amostras de neuropixels para estudar as seções visuais do córtex do rato com vários estímulos visuais. Eles prometeram publicar no final de 2018
aqui , ao lado dos dados biphoton (também um conjunto de dados muito poderoso), mas até agora nada.
Em terceiro lugar, a tarefa de agrupar células para registrar o potencial extracelular me parece esteticamente bonita. Ele converge os métodos de microeletrônica, neurofisiologia e aprendizado de máquina. Além disso, possui grande valor fundamental e aplicado. Suponho que o público do habr esteja interessado em aprender sobre a culinária técnica da eletrofisiologia, ou seja, sobre algoritmos de agrupamento, porque nesta área já desenvolveu seu próprio zoológico. Por sua vez, acumulei várias perguntas para esses algoritmos e esse conjunto de dados não pode ser ignorado. Portanto, na próxima parte, passaremos à análise de alguns algoritmos, começando com o Klustakwik canônico, continuando com os métodos de modelo Kilosort ou Spyking Circus e depois o YASS, que
declara fortemente
sobre si mesmo que ele funciona mais rápido e melhor do que todos os outros, porque DL e porque pode. Tópico no github com uma lista de alguns clusterers
aqui . Antecipando algumas perguntas, não vejo o objetivo de desenvolver meu próprio algoritmo, porque a concorrência já é muito grande e muitas idéias já foram implementadas e testadas por outros. Mas se houver demônios, contribuirei com prazer.
Sugestões e desejos são aceitos. Obrigado pela atenção!